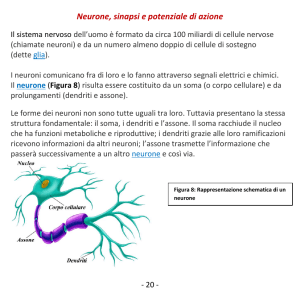
Sulla classificazione di famiglie proteiche attraverso Self Organizing Maps
Dalla scoperta della struttura del DNA nel 1953, la cosiddetta era genomica persegue come obiettivo il
sequenziamento del genoma di un organismo e la comprensione delle sue funzioni biologiche: un
gene, codificando una proteina, conserva in sé stesso l’informazione necessaria per esplicare una
specifica funzione.
La costruzione di modelli per la predizione delle strutture secondarie e terziarie, e quindi della
funzione di una proteina, a partire dall’allineamento multiplo di sequenze proteiche e dal
riconoscimento di regioni aminoacidiche ben conservate ad elevata similarità, appare come un
passaggio estremamente seducente, oltre che praticamente utile. Ricercare similarità ed omologie fra
proteine permette un loro raggruppamento in famiglie, e la possibilità di inferire proprietà dalla
definizione stessa della famiglia di appartenenza, partendo da una dato effetto per predirre un’ipotesi
casuale, che a sua volta può essere verificata con il metodo sperimentale.
Scopo di questa Tesi è costruire una Self Organizing Map o Rete di Kohonen, dedicata alla
classificazione di famiglie proteiche: addestrando questo particolare tipo di Rete Neurale Artificiale
non supervisionata su classi proteiche già riconosciute, è possibile determinare un loro
rappresentante, ed usare la similitudine ai clusters così costruiti per predirre l’omologia di nuove
proteine.
Un tale approccio è ragionevole in virtù del fatto che i processi evolutivi tendono a conservare la
struttura e la funzione di una proteina, e quindi la similarità fra proteine può effettivamente indicare
la loro omologia. Si può già intuire come l’affidabilità di un tale metodo predittivo risieda nella
definizione di una funzione di similarità per sequenze proteiche, nel riconoscimento di opportune
features caratterizzanti, e nell’indicazione di un valore soglia discriminante l’appartenenza ad una
famiglia.
Le cosiddette Self Organizing Maps, o SOMs, sono state introdotte da T. Kohonen tra il 1979 e il 1982,
ispirandosi agli studi biologici sul cervello umano: sono reti neurali a due strati non supervisionate ad
apprendimento competitivo, costituite da uno Strato di Input, e da uno Strato di Kohonen. Ogni
neurone di uscita è collegato con tutti i neuroni di ingresso, e con tutti quei neuroni di uscita che
rientrano in suo intorno; i pesi inter-strato sono modificabili, mentre i pesi intra-stato sono fissi e
dipendono dalla distanza dei neuroni connessi.
I neuroni dello strato di Kohonen sono utilizzati in competizione, rispondendo ad ogni ingresso con
un dato neurone vincente, ovvero quello i cui pesi sinaptici sono più vicini agli ingressi, autoorganizzandosi per rappresentare differenti classi di ingresso e specializzandosi nel riconoscere uno
stimolo: attraverso l’inibizione o l’eccitazione laterale, neuroni vicini a quelli attivi rinforzano i legami
in modo tale che ad input simili rispondano neuroni vicini.
1
In altre parole, la Mappa tenta di rappresentare le caratteristiche topologiche dello spazio vettoriale di
Ingresso, cogliendone alcune regolarità ed effettuando un raggruppamento o cluster dei dati, con
riduzione di dimensionalità e cardinalità sullo spazio di Uscita.
Il valore di uscita di una SOM può essere rappresentato sia dalla posizione nella griglia del neurone
vincente, realizzando una quantizzazione vettoriale adattiva da un vettore reale in ingresso ad un
valore discreto intero, sia dal vettore dei pesi sinaptici del neurone vincente, implementando un
codificatore vettoriale con una compressione delle coordinate di ingresso.
Per poter impiegare la SOM come classificatore di famiglie proteiche è necessario rappresentare le
famiglie in un qualche Spazio Vettoriale Normato, dove poter utilizzare una funzione distanza come
misura di similitudine.
Alternativamente, ricollegandosi agli studi sulla cosiddetta Protein Homology Network (PHN),
potrebbe essere interessante rappresentare la topologia di famiglie proteiche in un dominio
strutturato gerarchico, da proiettare su un piano bidimensionale come Training Set per una SOM.
In entrambi i casi, il punto di partenza è la similarità proteica espressa in termini di similitudine di
sequenze.
Mentre i primi studi connessionisti sui metodi predittivi della struttura secondaria e terziaria di una
proteina si fondano su Sistemi Esperti come il Multi Layer Perceptron, il nostro lavoro ha esplorato la
possibilità di utilizzare algoritmi di Clustering, in particolare le Self Organizing Maps, nella speranza
di preservare la topologia dello spazio di Ingresso nello spazio di Uscita a ridotta dimensionalità e
cardinalità.
Un incoraggiamento a proseguire è dato dal limite degli approcci classici connessionisti, dove la Back
Propagation richiede in fase di apprendimento un’enorme mole di calcoli sia in modalità on-line, con
aggiornamento iterativo su ogni ingresso, sia in modalità batch, con aggiornamento iterativo dopo la
presentazione dell’intero Training Set.
La strada che sembra più promettente, ma anche più complessa, è quella di combinare più
metodologie, integrando conoscenze biologiche, Soft Computing, algoritmi di Clustering.
Uno sviluppo di questa Tesi potrebbe combinare le varie tecniche di Soft Computing, costruendo una
SOM fuzzy, che possa sfruttare il calcolo parallelo intrinseco nelle Reti Neurali Artificiali, e possa
rilevare le sfumature di appartenenza di elementi multi-dominio, come spesso accade per le proteine:
una SOM con vicinato fuzzy sulla mappa ed uscite fuzzy, da ottimizzare attraverso algoritmi genetici
e da validare attraverso metodi statistici.
D’altra parte, dobbiamo osservare un limite intrinseco alle SOMs: la necessità di conoscere
preventivamente il numero di clusters. Per questo motivo, usualmente una SOM viene utilizzata per
ridurre la dimensionalità e la cardinalità di un Training Set, i cui dati così compressi sono
successivamente classificati attraverso un Multi Layer Perceptron.
2
Sebbene i metodi connessionisti riscuotano attualmente un successo maggiore di quelli classici logicosimbolici, vogliamo concludere suggerendo la possibilità di una loro rivalutazione. In particolare, il
Calcolo della Riscrittura di Termini, diretta semplificazione del Calcolo del Rimpiazzamento di Eguali
con Eguali, con il suo motore inferenziale basato sulla Riduzione di un termine in forma normale
rispetto ad un sistema di riscrittura, potrebbe adattarsi naturalmente a dati strutturati in grafi, siano
essi sequenze aminoacidiche o grafi etichettati rappresentati relazioni di omologia: una Macchina a
Riduzione che tenesse traccia del suo stato precedente potrebbe proporsi come un’interessante
implementazione delle cosiddette Reti Neurali Ricorrenti 1 , ed in particolare delle Reti di Elman.
1 Le Reti Neurali Ricorrenti sono caratterizzate da più cicli di retroazione, con 1 o più strati, e connessioni fra tutti i nodi, dove il
flusso di propagazione dei segnali è bidirezionale fra i vari nodi o addirittura ricorsivo su un stesso nodo.
3