Attivazione assegno di ricerca periodo 01/05/2012 – 31/04/2013
Titolo: Progettazione e sintesi di peptidi per la mappatura epitopica di proteine
di interesse diagnostico
Settore scientifico-disciplinare: CHIM/08 Chimica Farmaceutica
Responsabile Scientifico: Prof. Paolo Rovero
Per “epitope mapping” si intende il processo che porta all’identificazione dei siti di
legame degli anticorpi sui loro antigeni bersaglio di natura proteica. L’identificazione
e la caratterizzazione di tali regioni di legame può essere utile sia nella ricerca che
nello sviluppo di nuovi farmaci o vaccini, sia nel campo della diagnostica.
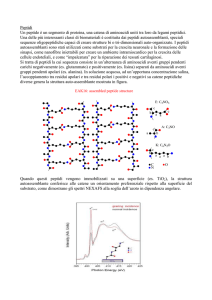
Gli epitopi (ovvero i siti di legame per gli anticorpi presenti sulle proteine) possono
essere di due tipi: lineari o conformazionali Gli epitopi lineari sono formati da
aminoacidi adiacenti, mentre gli epitopi conformazionali sono costituiti da regioni
aminoacidiche discontinue che vengono portate in vicinanza all’interno della struttura
tridimensionale della proteina. La maggior parte delle interazioni antigene-anticorpo
avviene mediante epitopi conformazionali [1]. I peptidi sintetici sono in grado di
mimare in maniera efficiente epitopi sia di tipo lineare che conformazionale.
Eseguire un epitope mapping su antigeni complessi è spesso problematico, perché tali
antigeni sono difficoltosi da esprimere e purificare. A questo proposito, un metodo
efficace è basato sullo scanning di peptidi sintetici (metodica chiamata pepscan
analysis). Questa tecnica utilizza una libreria di sequenze peptidiche costituita da
segmenti sovrapposti della proteina target e testa la loro capacità di legare gli
anticorpi di interesse tramite opportune metodiche, ad es. saggi immunoezimatici su
piastra (ELISA). Il metodo è rapido, piuttosto economico e particolarmente adatto per
effettuare l’identificazione di epitopi riconosciuti su una particolare proteina bersaglio
da parte di diversi anticorpi. La mappatura di epitopi conformazionali può essere
effettuata con alta affidabilità e precisione combinando sequenze peptidiche non
adiacenti presenti in zone differenti della proteina bersaglio ed imponendo la rigidità
conformazionale sulla struttura così ottenuta (peptidi conformationally constrained).
Sulla base di quanto detto, questo programma di ricerca sarà basato sul disegno e sulla
sintesi di peptidi da utilizzare per l’epitope mapping di proteine d’interesse
diagnostico, come ad esempio proteine di origine batterica. Sarà necessario analizzare
sia semplici peptidi lineari (epitopi lineari) che peptidi conformazionalmente vincolati
(mimetici di epitopi conformazionali).
Riferimenti
1. Westwood MR., Hay. FC. (2001). Epitope Mapping: a practical approach. Oxford,
Oxfordshire. Oxford University Press.