Corso di Dottorato di Ricerca in Biologia Ambientale
Presentazione progetto di ricerca
Dottorando: Federica Piazza
Ciclo: XXVII
Tutor: Prof. Alberto Pallavicini Co-tutor: Dr. Piero Giulianini
Titolo: Analisi genetica della popolazione di Austropotamobius complex nella regione Friuli
Venezia Giulia.
mente compromesso a causa del
nelle sue svariate forme, negli ultimi 50 anni.
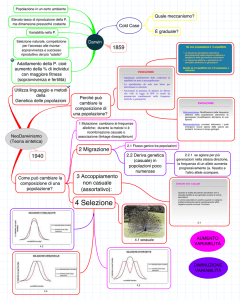
Il gambero di fiume Austropotamobius pallipes (Lereboullet,1858) è la specie autoctona più diffusa
in Italia. La sua distribuzione appare notevolmente contratta rispetto al secolo scorso, mentre le altre
due specie di gamberi autoctoni presenti in Italia il gambero nobile, Astacus Aastacus, e il gambero
di torrente, Austropotamobius torrentium, sono ad oggi rappresentate in Italia da un numero esiguo
di popolazioni altamente minacciate.
In questo contesto si inserisce il progetto europeo LIFE RARITY che ha come obiettivo il
ripopolamento del gambero di fiume autoctono Austropotamobius pallipes
specie invasiva Procambarus clarkii in Friuli Venezia Giulia.
Nel corso di questo dottorato mi occuperò della caratterizzazione genetica delle popolazioni di A.
pallipes presenti in FVG per poter in un secondo momento selezionare dei riproduttori
geneticamente idonei al rafforzamento delle popolazioni esistenti nel territorio regionale.
ETP in 216 stazioni prescelte nei 16 collegi di
pesca della regione, nel corso dei prossimi mesi estivi.
Le analisi molecolari saranno articolate in 2 parti:
specie/sottospecie presenti in FVG, dal momento che
A. pallipes è
controversa e il taxon è attualmente considerato come un complesso di specie (A. pallipes complex),
sulla base delle indicazioni fornite da recenti studi molecolari. Sono state finora identificate 2 specie
o linee evolutive geneticamente ben differenziate: A. pallipes e A. italicus, entrambe presenti nel
territorio italiano, la prima confinata nelle regioni nord occidentali, la seconda distribuita lungo
. Il taxon A. italicus a sua volta appare costituito da quattro sottospecie, A. i. italicus
-emiliano, A. i. carsicus nelle regioni nord-orientali, A.i. carinthiacus nelle
regioni centrali e nord occidentali e A. italicus meridionalis nelle regioni centro-meridionali.
Il primo obiettivo sarà quindi determinare quali sottospecie siano presenti in regione e quale sia la
codifica per Rna ribosomiale 16s (16S rDNA) e il gene per la subunità 1 del citocromo c
ossidasi (CO1). Il 16s rDna è il marcatore tradizionalmente utilizzato come orologio molecolare
negli studi di filogenesi molecolare, mentre la co1 è attualmente il marcatore prescelto come
in numerosi taxa animali nel progetto Barcoding of Life , che mira alla
la sequenza parziale
di questo gene mitocondriale.
marcatori ci permetterà una maggior
disponibilità di informazioni dai database pubblici, e verosimilmente una maggior affidabilità dei
filogeografica, cioè sulla
risultati. Inoltre questi marcatori mitocondriali ci permetteranno
distribuzione geografica degli aplotipi, fornendo informazioni sulla passata storia demografica
delle sottospecie/popolazioni individuate (la presenza di barriere geografiche o al contrario di
migrazione
La seconda parte è rivolta a uno studio della variabilità genetica inter e intra popolazione ,
uso di marcatori nucleari altamente polimorfici, i microsatelliti, che permettono
maggior risoluzione nello studio della struttura genetica delle popolazioni/ecotipi.