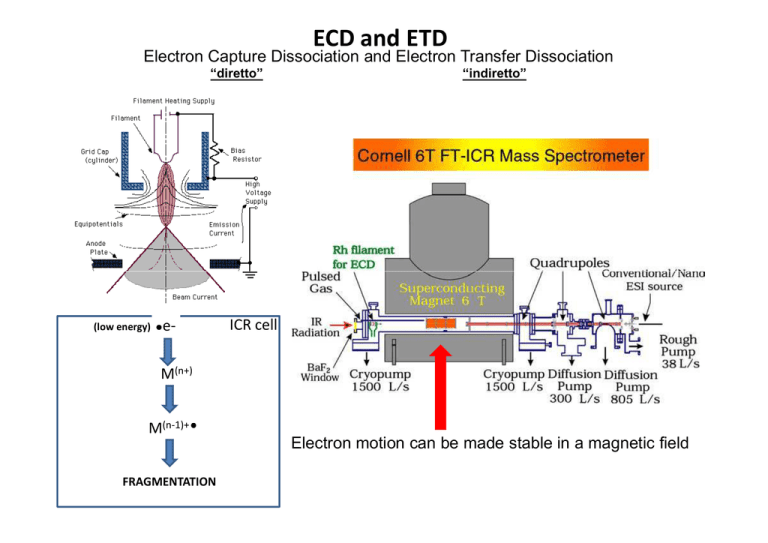
ECD and ETD
Electron Capture Dissociation and Electron Transfer Dissociation
“diretto”
(low energy)
e-
“indiretto”
ICR cell
M(n+)
M(n-1)+
Electron motion can be made stable in a magnetic field
FRAGMENTATION
ECD/ETD fragmentation process
Principalmente K
H2
(low energy)
ECD/ETD fragmentation process
Multiply
Reactant
charged
analyte
(n≥ 2)
n+
odd-electron
protonated
peptide
radical
anion
+
-
Electrontransfer
(n-1)+
Prerequisite: multiply charged precursor ions, n ≥ 2 !
ETD is not applicable to 1+ or negatively charged ions
Cleavage of
N-Cα bond
ETD versus CID
ETD
CID
• electron transfer surpasses internal heating
• rapid bond cleavage (no energy dissipation)
• random fragmentation of peptide backbone
• leaves labile bonds like from PTMs intact
• N-Cα bond cleavage yields c- and z-ion
• preferable charge state z > 2
• via several collisions with Helium
precursor ion is internally heated
• preferences for weak bond cleavages
(labile bonds like from PTMs)
• nearby selected amino acids (E, D, P)
backbone cleavage is preferred
• b- and y-ions (and internal fragments)
• best fragment spectra from 2+ ions
y3 z3
b 1 c1
y2 z2
b 2 c2
y1 z1
b 3 c3
Electron Capture Dissociation - ETD
Electron Carrier
A
+
Reactant Anion
e-
Electronattachment
m/z = 202
•(Idrocarburi aromatici)
Flouranthene
Radical Anion
C16H10•-
Generation of Reactant
Negative Chemical Ionization
(nCI) Source
1st Step:
Generation of low energy electrons
by EI of Methane (Mediator)
M+e
M • + 2e M
Electron attachment
to flouranthene
-
A
+
-*
2nd Step:
A+e
M
eA
e
A
-•
m/z 202
A
M e
M
e*
e
* e- = high energy electrons
ca. 60-80 eV
ETD in Non-linear Paul Trap (conventional Ion Trap – IT)
Dual injection and storage of ions of both polarities peptide cations &
reactant anions
Cations and anions are pushed
towards the center of the trap
Direct ETD reaction as soon as
anions enter the trap
Better cross sections for ion-ionreactions in 3D trap due to
compression into the same
globular volume
Ions (both cations (analyte) and anions
(reactant) are stored inside the trap
highly efficient ETD reaction
ETD – ions path
1. ESI ion
accumulation
(positive mode)
Gate lens block
2. precursor ion
isolation
3. nCI ions
accumulation
4. ETD
fragmentation
5. scan
Gate lens open
•Protein
•Multiple charge state precursor selection
RI (%)
ETD – High efficiency fragmentation for n>2 peptides (MnH+ )
TOP down proteomics
m/z
•Fragmentation by ETD => highly charged fragments
difficult to interpret – low resolution
=> no charge state determination
•Charge state reductution by PTR (proton transfer reaction)
•Singly charged fragrment of N- C-terminal portion
(m/z detactable depending on the m/z range of the ion trap)
•Sequence assignement
ELETTROFORESI – principi generali
Una molecola con una carica netta non nulla posta tra due elettrodi di segno opposto migra verso
l’elettrodo con segno opposto alla sua carica netta
d
+
v
-
q
-
ddp (V)
ddp (V): differenza di potenziale applicata tra gli elettrodi
d: distanza tra gli elettrodi
q: carica della molecola
E (campo elettrico) = V/d
La molecola si muove verso l’elettrodo di segno opposto con una velocità(v) che è proporzionale
all’entità del campo elettrico (V) e della sua carica (q) e inversamente proporzionale all’attrito che
incontra nel muoversi (f – coefficiente d’attrito)
v = Eq/f
ELETTROFORESI – principi generali
Molecole biologiche presentano gruppi acidici o basici che sono quindi ionizzabili
Per essere analizzata in elettroforesi una molecola deve avere una carica non nulla
pH del mezzo in cui sono in soluzione le molecole deve condizionare la loro carica
(soluzione tampone)
N°di ioni non basso
ioni dati dalle proteine (spesso trascurabili come entità numerica)
ioni dati dai costituenti del tampone: non trascurabili
Applicare una ddp (V) in un mezzo che contiene ioni equivale a generare una corrente (I) che è
inversamente proporzionale alla resistenza (R) del mezzo stesso
V=RI (legge di Ohm) => I = V/R
ELETTROFORESI – principi generali
Per la legge di Joule in un sistema elettrico caratterizzato da una resistenza R e nel quale viene fatta
passare una corrente I si sviluppa sotto forma di calore una potenza che è data da
W = VI = I2R
Cosa provoca il calore in un sistema del genere?
a) Moti convettivi nel sistema
b) Aumento diffusione molecole
c) …
Effetto negativo sulla
separazione delle molecole
– Risoluzione -
d
+
v
-
q
ddp (V)
-
Strategie
Elettroforesi condotta in un
mezzo che contrasta/minimizza i
moti convettivi e la diffusione
delle molecole
=>
ELETTROFORESI su
SUPPORTO SOLIDO
Rimozione estremamente
efficiente del calore attraverso
la riduzione delle dimensioni
del sistema elettroforetico
=>
ELETTROFORESI
CAPILLARE
(Free Flow Electrophoresis)
Analisi elettroforetica – workflow Su Chip
Rilevazione on
line
In capillare (CE)
Preparazione
del campione
Campione
deve
essere
sciolto in una soluzione che
non
interferisce
con
il
processo separativo e che
contemporaneamente
lo
condiziona per la stessa
separazione
Per le separazioni in gel di
solito è previsto l’uso di un
addensante (glicerolo) per
facilitare il caricamento e di
un tracciante per visualizzare
la corsa (in tamponi basici e
con corsa anodica si usa il
BBF (Blu di Bromofenolo)
Separazione
elettroforetica
Rilevazione in
gel
In condizioni:
• native
• denaturanti
Su supporto solido
Su:
• Acetato di cellulosa
• Agarosio
• Poliacrilamide
Trasferimento
su membrane
Western-blot (proteine)
Southern-blot (DNA)
Northern-blot (RNA)
Nitrocellulosa
PVDF
Per capillarità
Sotto campo elettrico
• Spettrofotometrica
• Spettrofluorimetrica
• CE/MS (Mass Spectrometry)
• Metodi di colorazione
Blue Coomassie
Argentica
Coloranti fluorescenti
• Radioattiva
…
Rilevazione su
membrana
•Metodi di colorazione
Rosso Ponceau
Blue coomassie
…
• Anticorpale
• Radioattiva
…
Elettroforesi su supporti solidi
“Su fogli sottili”
“In Gel”
⇒Carta
⇒Acetato di cellulosa
⇒ Gel di agarosio
⇒Strati sottili di silice, cellulosa, etc.
su supporti solidi
(TLE Thin Layer Electrophoresis)
⇒Gel di poliacrilamide
L’elettroforesi su acetato di cellulosa, che pur
essendo una metodica che non raggiunge le
risoluzioni ottenibili con i più moderni gel di
poliacrilamide ed agarosio, riveste ancora un ruolo
in ambito clinico per la valutazione delle proteine del
siero ed altre specifiche applicazioni.
Metodiche relativamente veloci
=>
Buona applicabilità in campo diagnostico/clinico
(test rapidi)
L’elettroforesi su gel di agarosio e su gel di
poliacrilamide offrono una buona risoluzione e
vengono comunemente impiegati nei laboratori di
ricerca rispettivamente soprattutto per l’analisi di
DNA e delle proteine.
L’elevato tempo richiesto per effettuare una corsa
con queste metodiche non li rende attrattivi per quel
che concerne un uso diagnostico ad alta
processività.
Elettroforesi (schema)
Elettroforesi su acetato di cellulosa
Campione viene deposto sulla superficie del supporto di
acetato di cellulosa che è imbibito del tampone di corsa e
successivamente viene applicata la ddp. Il pH del tampone
determina la carica delle molecole che si separano
secondo il loro rapporto m/z e l’attrito che incontrano nel
muoversi.
Elettroforesi
Striscia di acetato di cellulosa
Ddp (V)
Elettroferogramma
Albumin
+
-
Globuline
Buffer
α1
α2β1 β2
γ
pH ≈ 8.5
Buffer
Analisi condotta in circa 15 minuti
Esempio di un referto riguardante le Sieroproteine
Alcune patologie sono caratterizzate da specifiche
alterazioni del profilo elettroforetico delle proteine
del siero. Sono in commercio anche kit per la
valutazione di proteine urinarie, lipoproteine,
emoglobine, etc.
Gel di Agarosio
Polimero di D-galattosio
e
3,6 anidro L-galattosio
Transizione di stato
(macroreticolo sulla base di
legami idrogeno)
Transizione gel-sol tramite
riscaldamento
Agarosio modificato mediante
aggiunta di gruppi CH3 sui
gruppi OH ha temperature di
transizione minori (low melting
agarose)
Reticolo di agarosio polimerizzato
“setaccio molecolare”
Gel di Agarosio – Separazione molecole di DNA
Molecole di DNA, essendo costituite da unità
ripetitive recanti ciascuna la stessa carica, hanno
tutte lo stesso rapporto massa/carica.
Se messe in una soluzione e poste sotto l’azione di
un
campo
elettrico
esse
migrerebbero
sostanzialmente
alla
stessa
velocità,
indipendentemente dalla loro grandezza.
Se la migrazione viene fatta avvenire in un setaccio
molecolare
esse
si
separano
in
base
all’attrito/difficoltà che incontrano nel muoversi
lungo la matrice stessa.
Molecole di piccole dimensioni (numero di paia di
basi minori) presenteranno una migrazione
elettroforetica maggiore rispetto a molecole più
grandi (numero di paia di basi maggiore)
Log (bp o MW)
Tipiche condizioni di corsa:
Tampone: TAE o TBE (Tris/Acetato/EDTA o
Tris/Borato/EDTA) – pH circa 8
ddp: circa 5 V/cm
% Agarosio: dal 0.5 al 1-2%
Visualizzazione tramite intercalazione di Etidio
Bromuro (fluorescenza su transilluminatore UV)
Distanza percorsa
Polimerizzazione di un gel di poliacrilamide
(APS + TEMED + Acrilamide + Bis-Acrilamide)
Catena di poliacrilamide polimerizzata
senza la presenza di un agente reticolante
Radicale libero
Ammoniopersolfato (APS)
Radicale libero
agente reticolante
CH2=CH-CONH2
T: (g acril. + g bis-acril.) / volume (espressa come percentuale)
x grammi/100 ml => x %
C: g bis-acrilamide / (g bis-acril. + g acril.) (espressa come percentuale)
y grammi bis-acril / 100 gr (bis-acril. + acril.) => y %
T e C devono essere determinati empiricamente. Nella IEF, si usano bassi valori (soprattutto
per quanto concerne T; T=4%, C=3%) poiche le proteine devono essere libere di raggiungere
la zona dove il pH del gel è uguale al loro pI. In altre metodiche elettroforetiche, la frizione
che le molecole incontrano muovendosi durante la corsa elettroforetica è parte integrante del
processo separativo => è opportuno valutare quali siano le migliori combinazioni di T e C per
ottenere una buona separazione.
Catena di poliacrilamide polimerizzata in presenza di un agente
reticolante (“bis-acrilamide”)
Elettroforesi su gel di poliacrilammide (PAGE) - Proteine
Native
Condizioni
Denaturanti
Viene mantenuta la struttura secondaria /
terziaria ed evenualmente quaternaria delle
proteine (o di loro complessi con altre
molecole come ad esempio il DNA)
La struttura secondaria / terziaria ed
eventualmente quaternaria delle proteine
viene persa a causa dell’utilizzo di detergenti
(i.e. SDS) o agenti denaturanti (i.e. UREA).
Condizioni:
Si opera con tamponi ad un pH non lontano
dalla neutralità.
Si evita assolutamente il riscaldamento del
sistema cromatografico che può causare la
denaturazione delle proteine/complessi per
effetto termico
Non è possibile utilizzare detergenti non-ionici
ne tanto meno ionici, come anche non si
possono utilizzare sostanze riducenti.
Condizioni:
Possono essere adottati tamponi di corsa
anche a pH lontani dalla neutralità.
Generalmente è accettabile un blando
riscaldamento del sistema elettroforetico.
Si fa in modo che eventuali dimeri o complessi
macromolecolari uniti da ponti disolfuro
vengano rotti per azione di agenti riducenti
(DTT o beta-mercaptoetanolo).
La migrazione elettroforetica è influenzata sia
dalla carica della molecola che dall’ingombro
sterico della stessa che comporta un
rallentamento a causa dell’effetto setaccio del gel
in cui avviene la migrazione.
Non si possono fare alcune ipotesi circa il peso
molecolare delle proteine analizzate.
Nel caso in cui non vengano utilizzati detergenti
ionici la migrazione è influenzata dalla carica
della molecola e dall’attrito che essa incontra
muovendosi nel gel, con la differenza rispetto ai
gel nativi che l’attrito non è collegato alla sua
struttura secondaria/terziaria ne tanto meno
quaternaria ma solo all’estensione della sua
struttura primaria. Nel caso si utilizzino dei
detergenti ionici questi vanno a conferire
sostanzialmente a tutte le proteine il medesimo
rapporto m/z e pertanto esse si separano in base
al loro peso molecolare
Laurea Specialistica in Genomica Funzionale
Metodologie per lo studio del proteoma
Sgarra Riccardo
Dipartimento di Scienze della Vita
Università degli Studi di Trieste
AA 2011-2012
Programma del Corso
1)
Introduzione alla proteomica
2)
Estrazione delle proteine e preparazione del campione
•
Metodi di estrazione delle proteine
•
Metodi per la quantificazione di proteine
•
Metodi diretti
•
Metodi indiretti
•
Preparazione del campione per analisi elettroforetiche
•
Preparazione del campione per analisi cromatografiche
•
Metodi di prefrazionamento proteico
3)
Proteomica basata sulle analisi elettroforetiche (bidimensionali)
a) Introduzione alla gel elettroforesi
b) Elettroforesi SDS-PAGE
c) Elettroforesi in condizioni native
d) Blu native elettroforesi
e) Acido acetico urea
f)
Isoelettrofocalizzazione (IEF)
g) Combinazioni bidimensionali (IEF-SDS PAGE)
h) Metodi di colorazione delle proteine
•
Blue coomassie
•
Argentica
•
DIGE
•
Altre colorazione particolari
i)
Recupero degli spot/bande e loro processamento per analisi MS
4)
Proteomica “gel free” o “shot gun” (I parte)
a) Introduzione alla cromatografia e alla strumentazione
b) Cromatografia a scambio ionico
c) Cromatografia d’affinità
d) Cromatografia a fase inversa (RP e ion pair RP)
e) Cromatografia d’esclusione
5)
Spettrometria di massa
a) Concetti base della spettrometria di massa
b) Metodi di ionizzazione
•
MALDI
•
ESI
c) Analizzatori di massa
•
Quadrupoli
•
TOF
•
Trappole ioniche
•
FT-ICR
•
Orbitrap
•
Sistemi ibridi
Q-TOF
LIT-FT-ICR
LIT-Orbitrap
Ion Mobility Mass Spectrometry
6)
Identificazione delle proteine mediante MS
a) Concetti base della tandem mass spectrometry
b) CID – Collision induced dissociation
c) ETD ed ECD
d) Sequenziamento delle proteine mediante MS
e) Peptide Mass Fingerprinting
7)
Proteomica “gel free” o “shot gun” (II parte)
a) Cromatografia multidimensionale
b) SILAC
c) ICAT
d) ITRAQ
8)
Tool bioinformatici per l’identificazione delle proteine
9)
Approcci proteomici per lo studio di interazioni proteina-proteina
a) Dalla Co-IP alla Tandem Affinity Purification – TAP
b) Protein Array
c) BRET / FRET
d) Surface Plasmon Resonance
Metodologie per lo studio del Proteoma
(Sgarra Riccardo – [email protected] – 040 5583676 – Dip.to
Scienze della Vita, Ed. C11, II° Piano, Stanza 220)
Orari di Lezione: Come da calendario fornito (9 cfu)
Question Day: da concordare (corso finito, prima del 1° appello)
Date di esame:
Indicativamente quelle riportate sull’orario.
Solitamente due altri appelli in autunno (distanziati di almeno un
mese, uno a dicembre e uno a febbraio.
Date da Concordare.
Ricevimento: Tutti i giorni (su appuntamento concordato tramite
e-mail )
Testi consigliati: nessuno – tutte le informazioni sulle slide
presentate a lezione – disponibili in rete
Esame:
a)
b)
c)
d)
e)
Compito Scritto;
31 domande con risposta puntuale o “semi aperta”;
Possibilità di sostituire 1 domanda con una domanda a scelta
(Formulazione di una domanda e relativa risposta);
Punteggio: Ciascuna domanda può essere valutata 1, 0.75, 0.5, 0.25 o 0;
Voto finale: Sommatoria dei punteggi relativi alle 31 domande con
possibile aumento di 1 punto durante la correzione del compito in
dipendenza delle risposte fornite relativamente ai chiarimenti richiesti.
Esempio:
1) Operare con un metodo per l’estrazione delle proteine che preveda molti passaggi e che richieda molto
tempo cosa potrebbe comportare? (si consideri naturalmente conseguenze a livello del campione
proteico estratto)
……………………………………………………………………………………………………………………………
……………………………………………………………………………………………………………………
…………………………………………………………….
2) I campioni proteici sottoposti ad analisi di tipo proteomico sono solitamente estremamente complessi (in
termini di numero di componenti). Per analizzare questi campioni è dunque necessario adottare delle
metodiche ……………….. caratterizzate da un’elevata ………………………
DNA
RNA
GENOMICA
Mantenimento e
trasmissione
Trasferimento
informazione
informazione
L’informazione di base
di un organismo è uguale
per tutte le sue cellule
L’utilizzo dell’informazione
è differenziale a seconda
del tipo di cellula e del particolare
contesto biologico preso
in considerazione
PROTEINE
PROTEOMICA
Effettori
molecolari
Il complemento proteico è differente
a seconda del tipo di cellula e
del particolare
contesto biologico preso
in considerazione
Approcci globali nello studio della biologia cellulare
FINGERPRINT – IMPRONTA DIGITALE
Genomica
Studia l’informazione utilizzata
- profilo di espressione genica ovvero quali geni sono attivi in un determinato momento
STIMOLO
=>
Risposta cellulare in termini di geni attivati/disattivati
Proteomica
Studia il complemento proteico di una determinata cellula
- profilo di espressione proteica ovvero quali proteine sono presenti in un determinato momento
STIMOLO
=>
Risposta cellulare in termini di variazioni del complemento proteico
Approcci allo studio della biologia cellulare/biochimica
Classico
Proteomico
?
?
?
?
?
?
Singola Proteina
Sequenza
Struttura
Funzione
Processi biologici
?
Singola Proteina
Sequenza
Struttura
Funzione
Processo biologico
Proteine coinvolte
Relazione funzionale tra di esse
Proteomica
Differenziale
Comprensione di quali sono
le variazioni a livello dei
componenti proteici
coinvolti in un determinato
processo cellulare
Totale
Comprensione di quali sono
i costituenti proteici di una
determinata cellula,
organello, complesso
macromolecolare etc.
Analisi
Analisi su un
Comparativa
singolo sistema
PROTEIN FUNCTIONAL NETWORK
Conoscere quali sono le molecole “coinvolte” in un determinato processo non è un’informazione sufficiente a
spiegare quel determinato processo, è necessario conoscere le loro attività e le relazioni che esse hanno con gli
altri fattori proteici e cosa queste relazioni significhino/comportino
=>
Costruire network molecolari a cui vengono attribuiti dati funzionali.
Effetti “diretti” – i.e conformazionali, PTMs, etc
Effetti “indiretti “– i.e. regolazione trascrizionale, etc
Genomica
Proteomica ?
equivale a
DNA (x)
mRNA (x3)
trascrizione
mRNA (x)
Traduzione
Pro-Proteina (x3)
miRNA
Processamento RNA
(splicing alternativo)
mRNA (x1)
mRNA (x2)
mRNA (x3)
mRNA (x4)
Processamento
proteolitico
Proteina (x3.1) Proteina (x3.2) ....
PTMs
Proteina (x3.1A) Proteina (x3.1B) ....
Localizzazione
Compartimentalizzazione
Proteina (x3.1A-α ) Proteina (x3.1A-β ) ....
Degradazione
Proteina (x3.1A-α1 ) Proteina (x3.1A-α2 ) ....
miRNA
Una proteina è, in un determinato istante, il risultato dell’azione di
tutta una serie di attività in serie e/o in parallelo su di essa.
Fenotipo di un organismo:
proprietà
osservabili
di
un
organismo prodotte a partire dal
suo genotipo in combinazione con
l’ambiente.
? livello di osservazione
fenotipo:
a) morfologico;
b) fisiologico;
c) biochimico;
d) molecolare.
del
L’acquisizione di un particolare
fenotipo non è dovuta (nella
maggior parte dei casi) ad un
singolo gene ma ad una
cooperazione fra diversi di essi.
A livello molecolare un gene (o
meglio il suo prodotto proteico)
acquisisce una funzione/esercita la
sua influenza solo se inserito in un
appropriato contesto molecolare
Dinamicità del proteoma
Complemento proteico di una cellula
VARIAZIONI
QUANTITATIVE
=> Cambiamenti nei livelli di espressione
=> Stabilizzazione proteica (strettamente
connessa a variazioni qualitatitive)
=> degradazione proteica
QUALITATIVE
=> Cambiamenti nell’utilizzo dell’informazione:
Attivazione / repressione genica
Splicing alternativo
=> processamento proteolitico
Attivazione /disattivazione proteica
degradazione proteica
=> Modificazioni post-traduzionali
(fosforilazione, acetilazione ....)
Variazioni del livello
di una determinata proteina
Variazioni delle forme
di una determinata proteina
Proteine diverse
Stimolo
Vie di trasduzione
del segnale
GENOMA
attivazione
effettori cellulari
produzione
effettori cellulari
Risposta
TEMPO: analisi cinetica dello sviluppo di una risposta cellulare
Circuito (network) genetico – Aspetti temporali - Cinetica
Gestione dell’immagine
(scanner,etc)
Colorazioni proteiche in gel
(Coomassie, argentiche,
Derivatizzazioni Chimiche
Fluorofori differenziali (2D)
(scanner con lapossibilità di
illuminare i gel
a determinate lunghezze d’onda)
dalla tecnologia dei
DNA array
Protein array
(nanotecnologie)
Metodologie di studio per
le interazioni proteina-proteina
2D gel
Proteomica
Spettrometria di massa
MS Protein sequencing
MS peptide figerprinting
Derivatizzazioni Chimiche
ICAT (MS) -shotgun proteomics
Cromatografia
RP-HPLC / Multi dimensional chromatography
(Miniaturalizzazione colonne)
Preparazione del campione
sviluppo di detergenti, agenti riducenti, etc
Robotica
(Dalla preparazione del campione
all’interpretazione dei dati)
Bioinformatica
Banche dati
Swiss-prot. etc
Peptide fingerprinting
MS sequencing
Sequenziamento genomi
Principali riviste di proteomica......
.............buona lettura












