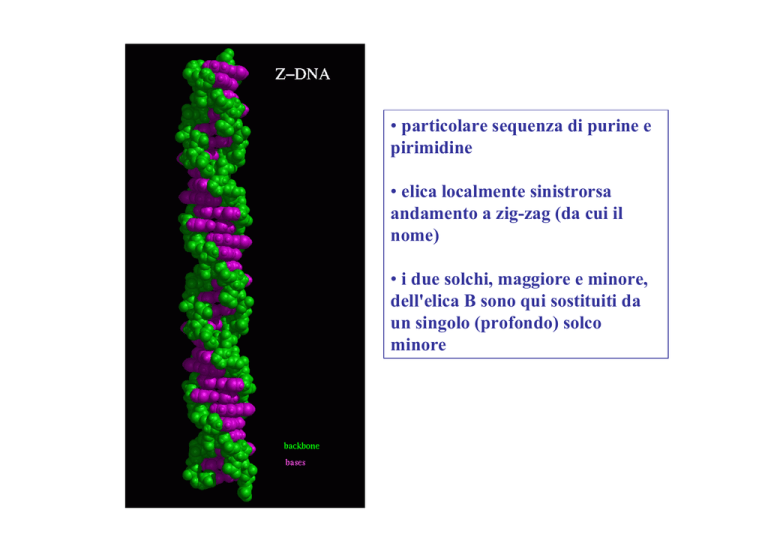
• particolare sequenza di purine e
pirimidine
• elica localmente sinistrorsa
andamento a zig-zag (da cui il
nome)
• i due solchi, maggiore e minore,
dell'elica B sono qui sostituiti da
un singolo (profondo) solco
minore
"ripetizioni invertite" o "repliche inverse"
Sn = RC[Sn]
S
A
T
O
R
A
R
E
P
O
T
E
N
E
T
O
P
E
R
A
R
O
T
A
S
"Il contadino con il suo lavoro
mette le ruote all'aratro"
Sn = RC[Sn]
C : operatore che trasforma ciascuna base nella base complementare
A→T, T→A, C→G e G→C
R: inverte l'ordine di lettura della sequenza
CGATCG→GCTAGC
Esempio:
Sn = GCTAGC
C[Sn] = C[GCTAGC]= CGATCG
RC[Sn] = RC[GCTAGC]= R[CGATCG] = GCTAGC = Sn.
Duplicazione dell’informazione genetica
mediante replicazione del DNA
In questo processo le due catene di una doppia elica di DNA
sono separate e ciascuna serve da stampo per la sintesi
della nuova catena compementare
Nota:
Le SNPs (snips) Single
Nucleotide
Polymorphisms
http://www.ncbi.nlm.nih.gov/Genbank/genbankstats.html
Le SNPs (snips) Single
Nucleotide Polymorphisms e
le differenze genetiche tra
individuo e individuo
Qualche dato
•
•
•
•
Negli esseri umani il 99.9 % delle basi sono identiche da individuo a
individuo.
Il rimanente 0.1 % determina l’unicità dell’individuo
Il 90% di tutte le differenze da individuo a individuo sono SNPs
2/3 delle SNPs comportano la variazione da Citosina, C, a Timina, T
(entrambe pirimidine)
• Aspetto esteriore
• Tendenza a contrarre malattie
•
Le variazioni da individuo a individuo possono essere:
– Innocue (modifiche del fenotipo)
– Dannose (diabete, tumore, malattie cardiache, Huntington's disease, emofilia )
– Latenti (le mutazioni non sono di per sè dannose, ma possono diventarlo in
particolari condizioni: es. Tendenza a sviluppare il cancro ai polmoni
nell’individuo fumatore)
Il genoma umano è composto da ~4x104 geni
In quasi tutti gli individui, quasi tutti i geni sono quasi identici.
(i geni devono essere molto simili altrimenti l’organismo non può
funzionare correttamente).
Alcune basi o sequenze nel DNA possono essere diverse da persona a
persona senza che ci sia nessun effetto sulle funzioni dell’organismo
Alcune variazioni producono diversi fenotipi come: diverse altezze,
colore degli occhi, impronte digitali o la capacità di piegare la lingua.
In alcuni casi, particolari variazioni genetiche possono portare ad una
maggiore suscettibilità o resistenza ad una malattia.
L’esistenza di una diversità genetica in una popolazione è detta
polimorfismo
• Molte mutazioni sono all’origine di malattie
molto diverse fra loro
Come si possono identificare e studiare?
• La risposta ai farmaci è spesso individuale
Come si può spiegare?
La risposta è negli ‘SNPs’
• Cosa sono?
• Come sono coinvolti in problemi di
salute così diversi fra loro?
Cos’è uno SNP ?
• Con il termine SNP (Single Nucleotide
Polymorphismis) si indica la mutazione di
una singola base nella sequenza di DNA
che sia presente in una porzione
significativa (più dell’1%) di una
popolazione.
Sequenza originale
Sequenze mutate
polimorfismo
delezione
inserzione
cromosoma
traslocazione
Tipi di mutazioni
nella sequenza
genica
Dati sulle SNPs
• Le SNPs sono presenti
– nelle regioni codificanti, ma soprattutto in quelle non codificanti.
• Appaiono con frequenza molto elevata
– Da circa 1 ogni 1000 basi a 1 ogni 100 ÷ 300 basi.
• Le SNPs vicine ad un gene particolare diventano dei
marker per quel gene.
• Le SNPs nelle regioni codificanti possono alterare
significativamente la proteina codificata.
Le SNPs possono o no alterare la
struttura della proteina
SNP sul DNA
da C a G
Codone sull’ RNA
da CUG a CUC
Proteina
da leucina a leucina
Proteina
Nessun cambiamento strutturale
Mappa degli SNP
• Sequenze genomiche di un gran numero
di persone
• Confronto delle sequenze per scoprire gli
SNPs.
• Costruzione di una mappa unica del
genoma umano contenente tutti i possibili
SNPs
MAPPATURA
Si
sequenziano
tutti i genomi
Si localizzano
e
Si registrano
tutti gli SNPs
Si eleborano le
informazioni
SNP #1 nella
posizione x sul
cromosoma n
Profilo individuale degli SNPs
• Il genoma di ciascun individuo contiene un pattern diverso
di SNPs
• Le persone possono essere riunite in classi in base al loro
profilo di SNPs
• Il profilo di SNPs è importante per identificare la risposta
ad un farmaco
• Si possono individuare delle correlazioni tra il profilo di
SNPs e la risposta ad uno specifico trattamento
farmacologico
• Si ritiene che molte malattie importanti, quali alcuni tumori,
siano dovute a mutazioni multigeniche
SNP Profiles
Tecniche per individuare i
polimorfismi
• Tecniche di ibridizzazione
– Micro arrays
– Real time PCR (Polymerase Chain Reaction)
• Tecniche basate su enzimi
Gruppi coinvolti nella Mappatura
• U.S. Human Genome Project (HGP)
– Identificazione di migliaia di markers lungo il genoma e
semplificazione nella “navigazione” lungo la mappa
genomica
• Gruppo di compagnie farmaceutiche
SNP Consortium
– Farmacogenomica, diagnostica, ricerca biomedica
Gli SNPs non sono causa di malattie
ma possono aiutare a determinare la
propensione individuale a contrarle
¾ ApoE (apolipoproteina E): gene associato all’Alzheimer (AD)
¾ Contiene tre SNPs ⇒ tre alleli: E2, E3 e E4
•
Ciascuna allele differisce per 1 base
•
La proteina prodotto di ciascun gene differisce per 1 aminoacido
•
Individuo con almeno 1 E4 (dal padre o dalla madre) ⇒ alta probabilità
di contrarre l’AD
•
Individuo con E2 ⇒ minore probabilità di contrarre l’AD
Identificazione legale per mezzo del DNA
Polizia Scientifica
13 regioni del DNA scelte tra lo 0.1% diverso da individuo a individuo
The International HapMap Project is a partnership of scientists and
funding agencies from Canada, China, Japan, Nigeria, the United
Kingdom and the United States to develop a public resource that
will help researchers find genes associated with human disease and
response to pharmaceuticals.
The International HapMap Project is a multi-country effort to
identify and catalog genetic similarities and differences in human
beings.
Using the information in the HapMap, researchers will be able to find
genes that affect health, disease, and individual responses to
medications and environmental factors.
The goal is to compare the genetic sequences of different individuals
to identify chromosomal regions where genetic variants are shared.
By making this information freely available, the Project will help
biomedical researchers find genes involved in disease and responses
to therapeutic drugs.
In the initial phase, genetic data are being gathered from four
populations with African, Asian, and European ancestry.
Dal DNA alla proteina
L’informazione genetica è letta per
mezzo di un processo a due passi
Nel primo passo, la trascrizione, segmenti
di DNA sono utilizzati per dirigere la
sintesi di molecole di RNA
Nelle traduzione quindi le molecole di
RNA sono utilizzate nella sintesi di
proteine
Trascrizione
Procarioti
DNA
→ mRNA → Proteina
Regioni di DNA trascritte
ma non utilizzate nella traduzione
⇓
Regioni non-codificanti
Alcune Mutazioni nelle regioni non-codificanti
possono impedire al gene di esprimersi
DNA
⇓
Trascrizione
⇓
mRNA
⇓
Traduzione
⇓
Proteina
Eucarioti
Geni interrotti ⇒ Non-colinearità Trascrizione
Esoni
Introni
≡ Regioni codificanti
≡ Regioni non-codificanti
Qual è la funzione degli Introni?
DNA
⇓
Trascrizione
⇓
RNA-precursore
⇓
Maturazione o
splicing
⇓
mRNA (maturo)
⇓
Traduzione
⇓
Proteina










![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)