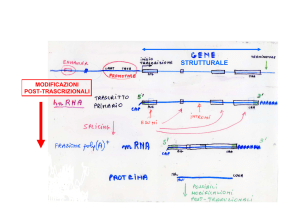
18. L’RNA regolatore contiene materiale protetto da copyright, ad esclusivo uso personale; non è
consentita diffusione ed utilizzo di tipo commerciale
Espressione geni è controllata da regolatori che interagiscono con una struttura specifica dell’mRNA 1
Regolazione mediata da RNA nei batteri
RNA regolatori batterici che agiscono in trans
2
sRNA (80-­‐110 nt) sequenza complementare al bersaglio (RNA antisenso): Formazione duplex RNA-­‐RNA Ex. OxyS
3
RNA regolatori batterici che agiscono in cis
• attenuazione negli operoni
• riboswitch
regolazione genica co-­‐traduzionale per ATTENUAZIONE nei procarioti la traduzione ha inizio mentre l’mRNA è ancora in corso di trascrizione. 4
Ex. L’operone Trp di E. Coli
La trascrizione della regione leader porta alla sintesi di un mRNA che va incontro a cambiamenti di conformazione con la formazione di strutture secondarie a stelo ed ansa 5
6
Box 20.1
Inoltre: presenza di trp-­‐tRNA ribosomi traducono il peptide leader e sono rilasciati Si forma la forcina e la RNApol termina la trascrizione assenza di trp-­‐tRNA ribosoma è bloccato la forcina di terminazione non si forma e la RNApol continua 7
RIBOSWITCH
si possono trovare nella regione 5’-­‐UTR dell’mRNA: controllano l’espressione dell’mRNA Organizzazione dei riboswitch
8
Ex. Riboswitch sensibile a S-adenosilmetionina (SAM)
Cambiamenti nella struttura secondaria del riboswitch che risponde alla SAM 9
Ex 1. produzione di glucosammina-­‐6-­‐fosfato (GlcN6P): intermedio sintesi parete batterica Sull’mRNA dell’enzima che sintetizza GlcNP6 si trova un RIBOZIMA che ha attività di endonucleasi (taglio) se attivata dal prodotto metabolico (GlcNP6) !GlcNP6 "attiva RIBOZIMA "taglio mRNA Il Riboswitch è dunque un RNA la cui attività è controllata dal prodotto metabolico Ex 2: geni per la biosintesi delle purine trascritti in base a livelli di G e conformazione dell’mRNA In assenza di G questa struttura a doppio filamento non si forma perché uno dei filamenti di RNA è accoppiato con una regione del biointerruttore 10
Es 3: Listeria monocytogenes mRNA della Listeria contiene sequenza di RNA termosensibile che controlla la traduzione di un gruppo di geni per la virulenza Nell’uomo la T alta denatura sequenza termosensibile ed i geni si esprimono Struttura di 7 riboswitch e dei metaboliti cui essi rispondono 11
Regolazione mediata da RNA negli EUCARIOTI
Caratteristiche RNA regolatore • è in grado di legarsi ad una molecola di mRNA impedendone la traduzione. • riconosce il suo bersaglio in base al principio di appaiamento complementare delle basi. • ha una certa struttura secondaria, ed una regione a singolo filamento complementare ad una regione a singolo filamento dell’mRNA bersaglio. 12
tipi di ncRNA • tRNA (RNA transfer): adattatori per la conversione del codice genetico a triplette nel codice amminoacidico delle proteine. • rRNA (RNA ribosomiali): all’interno dell’apparato traduzionale svolgono sia funzione strutturale che catalitica (formazione del legame peptidico). • snRNA (Piccoli RNA nucleari): componenti critici dello spliceosoma e svolgono un ruolo importantissimo nella maturazione degli mRNA. • snoRNA (Piccoli RNA nucleolari): implicati nel processamento e nella maturazione degli RNA ribosomali e di altri tipi di RNA, aumentandone l’attività. • miRNA e siRNA: piccoli RNA in grado di regolare l’espressione genica a livello post-­‐trascrizionale, silenziando specifiche molecole di mRNA. • piRNA: coinvolti nel silenziamento trascrizionale dei retrotrasposoni nelle cellule germinali • L’appaiamento di una molecola di RNA regolatore ad una regione a singolo filamento di mRNA può provocare: – L’inibizione della traduzione senza distruzione della molecola di mRNA – La degradazione della molecola di mRNA • In entrambi i casi si ha il “silenziamento” del gene, il cui effetto è la mancata produzione della proteina corrispondente. • Perché un RNA regolatore possa legarsi ad una molecola di mRNA bersaglio: – La regione di legame sul bersaglio deve essere accessibile , ovvero meno stabile (deve contenere una regione, anche piccola, a singolo filamento). – Il legame con l’RNA regolatore deve essere energeticamente favorevole. Accessibile
Non accessibile
13
L’interferenza dell’RNA (o RNAi) Ė una forma di “silenziamento” genico mediata da un doppio filamento di RNA (dsRNA). Quale è la funzione biologica dell’RNA interference? • L’iRNA svolge un ruolo protettivo del genoma da situazioni di instabilità causate da transgeni e trasposoni. • Costituisce un meccanismo di difesa contro attacchi virali. • Regola l’espressione genica (microRNA). 14
Andrew Z. Fire Craig C. Mello certe regioni genomiche codificano per alcune brevi molecole di RNA (dette genericamente microRNA), in grado di ripiegarsi a formare una forcina (short hairpin RNA, o shRNA). 15
I miRNA sono trascritti da RNAPol II come precursori piu’ lunghi (pri-­‐miRNA) con struttura a forcina, maturati nel nucleo a pre-­‐miRNA ed esportati nel citoplasma Nel citoplasma, i pre-­‐miRNA vengono maturati a miRNA ad opera di nucleasi chiamate DICER: 16
I miRNA maturi vengono inglobati in un complesso RISC (RNA-­‐
Induced Silencing Complex), che svolge la doppia elica e viene guidato dal filamento anti-­‐senso sull’mRNA bersaglio, inibendone la traduzione NB Il complesso RISC attivato riconosce sequenze complementari di RNA, le lega e degrada: questo puo’ avvenire diverse volte in quanto l’RNA antisenso, protetto dal complesso proteico è stabile e in cellule che si dividono lentamente puo’ agire per 3-­‐7 giorni. 17
Meccanismo d’azione miRNA due modi diversi d’agire a seconda della complementarietà che vi è tra il miRNA e il suo target 1. Complementarietà imperfetta • bloccano l’espressione dei loro geni target a livello post-­‐trascrizionale • Per inibire la traduzione legano solitamente le regioni non tradotte al 3’(3’ UTR), per le quali presentano omologia 2. Complementarietà perfetta • legano i loro RNA bersaglio con complementarietà perfetta inducono il taglio del bersaglio che non può più essere tradotto • In questo caso i miRNA trovano la loro regione di omologia o nell’ORF(open reading frame) o nella sequenza codificante microRNA (miRNA) 1.
Un solo miRNA può regolare un intero gruppo di mRNA diversi con sequenze comuni 2. Gene che codifica miRNA occupa poco spazio nel genoma 3. Nell’uomo oltre 400 miRNA diversi 18
Riassumendo miRNA: • La loro espressione e’ altamente regolata durante lo sviluppo • regolano l’espressione dei geni bersaglio a livello della traduzione, bloccando l’attacco del ribosoma -­‐> non mediano la degradazione dell’RNA bersaglio (tranne che nelle piante) • L’omologia con l’RNA bersaglio e’ a livello della regione 3’UTR, e non e’ completa. Quando l’omologia e’ completa mediano DEGRADAZIONE DELL’RNA BERSAGLIO • Rappresentano un importante livello di regolazione dell’espressione genica! 19
Funzioni dei miRNA
• controllo di numerosi processi fisiologici:
– Sviluppo
– Differenziamento cellulare
– Apoptosi
• down up regulation miRNA sono correlate a diversi tipi di
patologie:
– Cancro
– Malattie neurodegenerative
– Malattie cardiache
• Si tratta dunque di molecole molto importanti, il cui studio è
fondamentale nella comprensione dei processi biologici, dei
fenotipi patologici e, di conseguenza, nel design di terapie
innovative.
miRBase banca dati ufficiale dei miRNA
http://microrna.sanger.ac.uk/sequences/
Contiene le sequenze dei miRNA maturi e degli stemloop precursori, oltre a riferimenti bibliografici ed
informazione sui target.
• si conoscono circa500 miRNA
nell’uomo ma solo per il 10% di
essi è stato individuato almeno
un target.
• servizi di predizione di target per
miRNA:
– TargetScan
– PicTar
– miRanda
20
piccoli RNA interferenti: siRNA, small interfering DNA rappresentano un’altra classe di piccoli RNA non codificanti pur presentando numerose analogie con i miRNA in termini di struttura e biogenesi, svolgono funzioni essenzialmente diverse • siRNA si producono a partire da lunghe molecole di RNA prodotte da elementi genetici normalmente silenti o estranei alla cellula, quali trasposoni,virus o transgeni: un sistema di difesa contro l’invasione di elementi genetici estranei e di conservazione della stabilità del genoma • siRNA agiscono solo per complementarietà perfetta per cui ogni siRNA può avere un unico mRNA bersaglio • i miRNA costituiscono una numerosa classe di geni endogeni filogeneticamente conservati, la cui funzione è di inibire l’espressione genica principalmente attraverso l’inibizione della traduzione • Ogni miRNA, per il suo caratteristico meccanismo d’azione può avere più di un mRNA bersaglio 21
22
siRNA IN STUDI CLINICI 23
Potenziali effetti collaterali della terapia con siRNA: 1. Attivazione del sistema immunitario 2. Effetti off-­‐target 3. Saturazione di vie di silenziamento endogene (miRNA) 24