Maturazione dell’RNA e controllo
dell’espressione genica
26 Aprile 2017
Corso di Genetica Molecolare
CeciliaMannironi
Is.tutodiBiologiaePatologiaMolecolari
CNR
[email protected]
Marzo 2015
TotalNoofGenes: 60498
Protein-codinggenes:19797
TotalNoofTranscripts:198619
Protein-codingtranscripts:79795
TotalNoofGenes:46517
Protein-codinggenes:21936
TotalNoofTranscripts:111706
Protein-codingtranscripts:50162
Nell’uomo e nello scimpanze’ le regioni codificanti del genoma sono identiche
al 99.7%
….….Theresultsofthisstudythussupportanimportantroleforalterna3ve
splicinginestablishingdifferencesbetweenhumansandchimpanzees.
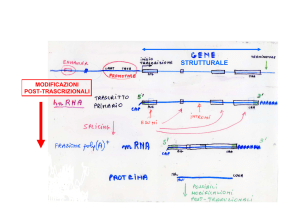
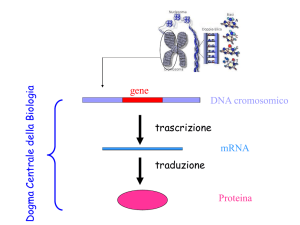
Maturazione dell’mRNA
La maturazione dell’mRNA, che comprende capping, splicing, editing (indicato
dagli asterischi) e poliadenilazione, precede la traslocazione dal nucleo al
citoplasma, dove avviene il processo della traduzione. Si noti la struttura ripartita dell’mRNA con le regioni non tradotte al 5' e al
3' (5'UTR e 3'UTR) e la regione codificante (CDS).
Maturazione del trascritto primario
Editing dell’RNA
Edi.ngC-U
Edi.ngA-I
Editing C-U
del gene per l’Apolipoproteina B
Entrambi i prodotti proteici del gene ApoB sono coinvolti nel metabolismo dei lipidi, ma
con funzioni diverse: la forma di 4563 aa epatica è coinvolta nel trasporto del
colesterolo e dei trigliceridi endogeni, quella di 2153 aa intestinale nel trasporto ai vari
tessuti dei lipidi assunti con la dieta. L’enzima che catalizza l’editing è
APOBEC1(apolipoprotein-B editing enzyme, catalytic polypeptide –like-1). Editing A-I
Meccanismo di deaminazione dell’adenosina
SanazFarajollahiandStefanMaas2010
L’inosina si comporta come una guanina L’inosina si appaia con la citosina
Recoding indotto da Editing
Se l’editing interessa una sequenza di RNA codificante puo’ determinare
sostituzione amminoacidica nel prodotto genico
Val
AUU viene normalmente
caricato da un tRNA Ile.
In seguito ad editing A-I il
codone Ile (AUU) è
convertito in un codone Val
(IUU)
c
IUU
AAAAA
AUU
Il cambiamento di lettura del codice genetico determinato da editing dell’RNA è detto
Recoding. Questo costituisce un meccanismo molecolare di espansione del landscape
proteico. Editing A-I
Editing Q/R della subunita’ GluR2 dei recettori ionotropici
L’editing Q/R (glutammina sostituita da arginina) determina modificazioni
funzionali del recettore e riduzione della permeabilita’ al Ca2+ dei recettori
ionotropici per il glutammato
I recettori GluR sono etero-oligomeri e GluR non è mai presente come
omopolimero GluR2 L’arginina nel loop del poro costituisce
un segnale di ritenzione per il Reticolo
Endoplasmatico .
Questo meccanismo impedisce la formazione di canali GluR2 omo-oligomerici
che interferirebbero con una normale trasmissione sinaptica eccitatoria La reazione di editing A-I è catalizzata dalle ADAR (Adenosine
Deaminase Acting on RNA).
• L’espressione delle ADAR è universale nei metazoi
• Nei mammiferi esistono 3 isoforme, ADAR 1-3. • ADAR 1 è ubiquitaria, ADAR 2 è espressa maggiormente nel cervello,
ADAR
espressa esclusivamente nel cervello e non ha attivita’ catalitica ma regolativa Le ADAR riconoscono una struttura secondaria dell’RNA Duplex imperfetto di 9-15
nucleotidi, che si puo’
formare tra sequenze
distanti anche 1000 nt 3 è
L’editing interessa sia RNA codificanti che RNA non codificanti
L’editing precede lo splicing dell’mRNA
La maturazione dell’mRNA, che comprende capping, splicing, editing (indicato
dagli asterischi) e poliadenilazione, precede la traslocazione dal nucleo al
citoplasma, dove avviene il processo della traduzione. Si noti la struttura ripartita dell’mRNA con le regioni non tradotte al 5' e al
3' (5'UTR e 3'UTR) e la regione codificante (CDS).
Tipica struttura di un introne da splicing nucleare
Sequenze introniche consensus
Sito donatore GU al 5’, sito di ramificazione contenente una A che si trova in
prossimità di un tratto polipirimidinico (Y)n, che precede il sito accettore AG al 3'.
Questi segnali, che permettono il riconoscimento e la rimozione degli introni, sono
contenuti in motivi oligonucleotidici più estesi, rappresentati in basso, dove la
dimensione del carattere corrispondente alla base è proporzionale al suo grado di
conservazione (notare che qui il sequence logo è dato come DNA e quindi contiene T
anziché U).
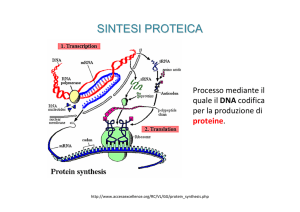
Il processo di splicing nucleare
Nel processo di splicing nucleare avvengono due reazioni di trans-esterificazione:
1.
la prima reazione di trans-esterificazione fa si’ che l’adenina del sito di
ramificazione sia coinvolta in tre legami fosfodiesterici (ingrandimento
mostrato a destra)
2. la seconda reazione di trans-esterificazione concatena gli esoni e libera
l’introne sotto forma di struttura a forma di cappio (lariat).
Il processo di splicing nucleare richiede l’intervento di un grande complesso
macromolecolare denominato spliceosoma, costituito da 200 proteine e 5 RNA
Un singolo gene puo’ generare anche centinaia di trascritti maturi diversi
L’AS puo’ modificare l’output proteico Pattern piu’ comuni di splicing alternativo (AS)
Lo splicing alternativo (AS) è un fenomeno altamente regolato
•
•
•
Piu’ del 95% dei geni umani vanno incontro a splicing alternativo Pattern alternativi di processamento dell’RNA costituiscono un meccanismo di
espansione del proteoma
Il suo malfunzionamento è alla base di molte patologie
L’interazione dei componenti dello splicing snRNP (U1 ecc) con proteine di
regolazione dello splicing (SRF) permette la scelta di siti alternativi di splicing. Le
SRF sono fattori che lavorano in trans riconoscendo elementi in cis presenti sul
pre-mRNA
Il processamento alternativo di un mRNA puo’
interferire con l’espressione e la regolazione dell’mRNA
3’UTRdiversiconferisconoinoltrediversastabilita’elocalizzazionedelmRNA
Controllo di qualita’ dell’ mRNA
nucleo
• Il complesso del poro nucleare riconosce ed esporta solo mRNPs contenenti
mRNA completi e completamente processati .
• Il complesso che lega il Cap e le proteine che legano la coda di poly(A) devono
essere presenti nei mRNP.
• Se avviene ritenzione di un introne questo inibisce il trasporto nucleocitoplasma dell’mRNA che viene quindi degradato.
Controllo di qualita’ dell’ mRNA
citoplasma
L’inizio della traduzione dell’mRNA richiede l’interazione tra le 5’-capbinding proteins e le poly(A)-binding proteins. RNA mancanti di queste
strutture non sono tradotti
mRNA danneggiati sono degradati da esonucleasi che attaccano il 5’ e 3’
non protetti
Degradazionedell’mRNAnellievito
Nonsense-mediated mRNA decay (NMD)
L’AS o una mutazione del gene possono portare alla comparsa di
un segnale di stop prematuro (non sense o premature
termination codon, PTC). Questo fenomeno attiva negli eucarioti un meccanismo di controllo di
qualita’ dell’RNA che porta alla degradazione del messaggero.
NMD
EJC:exonjunc.oncomplex
NMD
NMD
IlNMDèunprocessocitoplasma.cocherivelalapresenzadiPTCall’internodiun
mRNAenedeterminaladegradazione.
RNA Interference (siRNA)
HowardHughesMedicalIns.tute(HHMI)ScienceEduca.onProgram