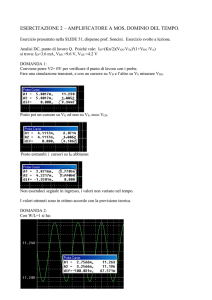
Nuove metodiche
per la rilevazione
e la genotipizzazione
dell’acido nucleico
ESTRAZIONE
1h
AMPLIFICAZIONE
(PCR)
3 - 4h
SEQUENZIAMENTO
giorni
RILEVAZIONE
(elettroforesi)
1h
MINI-ARRAY
LATERAL FLOW
ESTRAZIONE
1h
AMPLIFICAZIONE
RILEVAZIONE
e
GENOTIPIZZAZIONE
MINI ARRAY
• Metodo di rilevazione/riconoscimento dei prodotti di amplificazione
mediante l’ibridazione con specifiche probes e l’utilizzo di anticorpi
marcati con un enzima
1- Amplificazione
• Normale amlificazione dell’acido nucleico target con l’incorporazione di
dUTP legato con DIG
• DIG: digossigenina (steroide alternativo alla Biotina)
• Amplificazione di un tratto di acido nucleico che permette di
distinguere geneticamente diverse specie, varianti, ecc…
dUTP
DIG
DIG
DIG
2- Allestimento Mini array
• Membrana di nylon con diversi pozzetti
• Un pozzetto per ogni probe
PROBE SPECIFICA
Pozzetto nella membrana di nylon
3- Anticorpo anti-DIG marcato con enzima
(fosfatasi alcalina)
AF
Anti-DIG
AF
Ab anti-DIG marcato con fosfatasi alcalina
Anti-DIG
+
PRODOTTO di PCR
DENATURATO
DIG
+
PROBE SPECIFICA
Pozzetto nella membrana di nylon
A
B
C
D
Controllo
positivo
A+
B+
9
C+
negativo
D+
• 100 volte più sensibile dell’elettroforesi
• 10.000 volte più sensibile del dot-blot
Vantaggi
• Sensiblità: molto elevata
• Specificità: molto elevata (legata alle sequenze dei primers di
amplificazione e delle probes del mini-array)
• Facile da usare e da interpretare
• Poco costoso
• Rapido (4-5 h dall’estrazione al risultato)
• Permette di distinguere diversi genotipi
Svantaggi
• Non può essere utilizzata in condizioni di campo ma solo in
laboratorio, a causa delle attrezzature e della preparazione del
personale che richiede
LATERAL FLOW
• È la tecnica attualmente più utilizzata tra quelle che costituiscono le
cosidette “Microfluidic platforms” nelle applicazioni “Lab-on-a-chip”
• Lab-on-a-chip (LOC)
Supporti che integrano una o più funzioni di laboratorio in un unico
chip delle dimensioni di pochi mm o cm
• Microfluidic platforms
Tecniche che sfruttano le caratteristiche fisico-chimiche dei fluidi per
controllare e manipolare piccoli volumi dei fluidi stessi, in supporti di
piccole dimensioni e con un basso consumo di energia
• Microfluidic platforms nelle applicazioni Labon-a-chip
Supporti di piccole dimensioni in cui avvengono più funzioni di
laboratorio manipolando piccoli volumi di fluido
Come funziona un lateral flow?
• Si basa sul movimento per capillarità di microquantità di fluidi
Test rapidi per la ricerca di Ag
Lateral flow nella biologia molecolare
Rilevazione dei prodotti di amplificazione
utilizzando lateal flow coniugato
con dyed microspheres
Anziché rilevare un Ag mediante Ab,
rileva una determinata sequenza nucleotidica mediante probe specifica
1- Amplificazione
• Amlificazione dell’acido nucleico target (LAMP) con l’utilizzo di primers
marcati con BIOTINA
• BIOTINA: si lega ad alta affinità all’avidina ed alla streptavidina. In
alternativa vengono usati Ab-antiBIOTINA
primer
BIOTINA
BIOTINA
2- Ibridazione con sonda specifica
•
•
•
•
Denaturazione del prodotto dell’amplificazione marcato con BIOTINA
Ibridazione con una probe specifica marcata con FITC
FITC: fluoresceina isotiocianato, è un fluoroforo
Dall’ibridazione si forma un “bi-complesso” dato dal prodotto di
amplificazione marcato con BIOTINA e la sonda marcata con FITC
• Rappresenta la fase specifica della reazione
BIOTINA
BIOTINA
+
FITC
FITC
3- Allestimento lateral flow
Area di migrazione
Area di coniugazione
Ab-antiFITC coniugato ad una
Dyed microsphere
Caricamento “bi-complesso”
Area di adsorbimento
Test line del K+
Ab-antiAb
Test line del campione
Ab-antiBIOTINA
Caricamento “bi-complesso”
BIOTINA
FITC
Coniugazione
Si forma un “tri-complesso”:
- Prodotto amplificazione
- Probe
- Ab
BIOTINA
BIOTINA
FITC
+
Anti-FITC
FITC
Migrazione del “Tri-complesso” e degli Ab-antiFITC non legati
Test line del campione
BIOTINA
FITC
FITC
+
Anti-BIOTINA
BIOTINA
Test line del K+
Anti-FITC
+
Anti-Ab
Vantaggi
• Facilità di utilizzo
• Rapidità: 10-15 min
• Costo basso
• Utilizzo in campo
Svantaggi
• Impossibilità di distinguere in una sola reazione genotipi diversi,
occorre utilizzare una striscia per ogni variante che si desidera
discriminare