Tipi di ricombinazione
Omologa
– tra sequenze molto simili
(durante la meiosi)
Sito-Specifica – tra sequenze con limitata similarità.
Coinvolge siti specifici
Transposizione – movimento di elementi di DNA
da un sito ad un altro (trasposoni)
Ruolo biologico della
ricombinazione
1.
2.
3.
4.
Generare nuove combinazioni geniche o alleliche (crossing
over durante la meiosi)
Generare nuovi geni (riarrangiamento nell produzione di
immunoglobuline)
Integrazione di elementi di DNA
Riparo del DNA
Utilità pratiche
Mappatura dei geni sul cromosoma
Generazione di organismi transgenici
Ricombinazione negli eucarioti
Crossing-over
Modello di scambio dei filamenti secondo Holliday
1° sito
2° sito
Ricombinazione indotta da tagli a doppia elica
Ricombinazione indotta da tagli a doppia elica
RecA: forma attiva filamento nucleoproteico
(100 subunità di ReaA e 300 nucleotidi )
(Aumento della lunghezza del DNA legato a RecA)
Ricerca della complementarità
Appaiamento delle basi complementari (+/- 15coppie di basi)
RuvB ATPasi esamerica
La ricombinazione del DNA
Ricombinazione omologa
I modelli di Holliday e di Meselson-Redding.
Il complesso RecBCD genera il filamento di DNA
“invasore”.
La proteina RecA promuove lo scambio dei filamenti di
DNA.
Il complesso RuvABC promuove la migrazione del
chiasma e la risoluzione degli intermedi di Holliday.
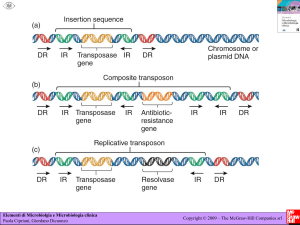
Trasposoni
I TRASPOSONI
I trasposoni sono elementi mobili che si trovano nel genoma di tutti gli organismi.
Sono in grado, con meccanismi diversi, di saltare da un punto all’altro del genoma.
Questi salti possono determinare drastici cambiamenti nella struttura e nelle
modalità di espressione dei geni.
Appartengono a diverse classi, accomunate dalla caratteristica di avere la
sequenza trasposta fiancheggiata da corte sequenze ripetute generate dalla
duplicazione di una sequenza nel sito bersaglio (Ripetizione intersperse o IR)
Sotto certi aspetti possono essere considerati come elementi parassitari, e in
effetti alcuni di essi sono imparentati con particolari famiglie di virus (retrovirus).
Tuttavia rappresentano un formidabile fattore positivo per i processi evolutivi.
La trasposizione è un fenomeno diverso dalla ricombinazione omologa ed è
chiamata ricombinazione non-omologa.
I trasposoni possono causare mutazioni geniche e mutazioni cromosomiche
Hanno un ruolo importante nell’evoluzione dei genomi
Elementi trasponibili
Sequenze che hanno la capacità di muoversi da un sito all’altro
del cromosoma
sito donatore
sito accettore
Diversamente dagli altri processi coinvolti nella ricombinazione
genomica, per la trasposizione non c’è nessuna omologia tra le
sequenze del sito donatore e accettore
Ripetizioni intersperse
Trasposoni a DNA
si originano attraverso un
intermedio a DNA, secondo
meccanismo di trasposizione
conservativa o replicativa
Retrotrasposoni
si originano per eventi di
retrotrasposizione, attraverso un
intermedio ad RNA
• elementi LTR
• LINEs: long interspersed nuclear
elements
• SINEs: short interspersed nuclear
elements
Trasposoni a DNA
I trasposoni a DNA
Codificano e utilizzano l’enzima trasposasi (capace di tagliare e
riunire il DNA in corrsispondenza di ripetizioni terminali invertite o
sequenze di insersione)
trasposasi
L’enzima trasposasi ha due diversi domini funzionali:
• un dominio catalitico che catalizza le reazioni necessarie per la
trasposizione del DNA, l’idrolisi dei legami fosfodiestere e una
reazione di inserzione del trasposone nel nuovo sito
• un dominio che lega il DNA
I Trasposoni a DNA possono essere distinti in due
categorie:
•Trasposoni a DNA che si spostano replicandosi: una
copia rimane nel sito originale, mentre la nuova copia si
inserisce altrove nel genoma
•Trasposoni a DNA che si spostano in maniera
conservativa, da un sito all’altro del genoma senza
aumentare il numero di copie.
Retrotrasposoni
Trasposoni che si muovono indirettamente
(retrovirus endogeni)
IR LTR
LTR IR
Proteasi Integrasi
RT
RNAsi-H
mRNA
cDNA
Integrazione
Gli pseudogeni che si ritrovano nel genoma umano sono spesso derivati
da eventi di retrotrasposizione
LINEs:long interspersed nuclear elements
promotore
Pol II
RNA binding
anche endonucleasi
ripetizioni
dirette
Gli elementi LINEs o trasposoni non-LTR hanno una lunghezza di
circa 6-7kb, contengono una o due ORF e un segnale di
poliadenilazione all’estremità 3’.
Le LINEs si inseriscono preferibilmente nelle regioni eucromatiche
ricche in A+T. Le copie attive inserendosi in punti critici del genoma
possono inattivare dei geni con conseguente insorgenza di
patologie.
Trasposizione degli elementi LINE
Trascrizione inversa innescata dal sito bersaglio
citosol
SINEs: short interspersed nuclear elements
A B
AAAA
SINE
Gli elementi SINEs sono elementi non-autonomi, hanno una
lunghezza compresa tra 0.1 e 0.4 kb.
Hanno una regione ricca in A all’estremità 3’ ma non contengono
un segnale di poliadenilazione.
Gli elementi SINEs non contengono alcuna ORF codificante per
una trascrittasi inversa, ma sono in grado di trasporre utilizzando la
trascrittasi inversa sintetizzata da altri retroelementi (trasposizione
LINEs-dipendente).
SINEs: short interspersed nuclear elements
Gli elementi SINEs sono distribuiti ad alta densità nelle regioni
ricche in CG del genoma
Nel genoma dei primati sono presenti tre differenti famiglie di
elementi SINEs: l’elemento Alu, ancora attivo, e gli elementi
inattivi MIR e Ther2/MIR3.
L’elemento Alu, il più comune nei primati, è lungo 0,3kb; è
presente in circa 1.200.000 di copie nel genoma umano e
rappresenta quindi oltre il 10% di tutto il genoma. Presenta una
regione ricca in A/T all’estremità 3’, coinvolta nel meccanismo di
retrotrasposizione.
Le sequenze Alu sono localizzate a monte o a valle dei geni, negli
introni, nelle regioni 5’ e 3’ non tradotte dell’mRNA. Non è noto il
loro ruolo funzionale, nonostante siano molto diffuse nel genoma
di tutti i primati.
EFFETTO DEI TRASPOSONI
Che cosa possono fare i trasposoni saltando nel genoma?
MUTAZIONE INSERZIONALE
Inserzione nella sequenza codificante (o nelle sequenze di controllo) con
inattivazione totale o parziale del gene.
Trasposone
A
B
C
D
E
F
*
A
B
C
D
E
F
Per questa proprietà, i trasposoni sono molto utilizzati per mutagenizzare e
successivamante identificare i geni responsabili di particolari fenotipi. Un
organismo che si presta molto bene a questo è Drosophila Melanogaster.
Inattivazione del gene bersaglio
trasposone
promotore
DNA
mRNA
proteina assente o tronca
Nel caso di trasposizione in un operone si osserva un effetto “polare”
P
gene 1
gene 2
gene 3
mRNA
policistronico
P
Anche i geni 2 e 3 non vengono espressi in quanto la trascrizione
è bloccata dall’ elemento trasponibile
Che cosa possono fare i trasposoni saltando nel genoma?
Attivazione trascrizionale del gene
Retroposone
LTR
LTR
A
B
C
D
E
F
A
B
C
D
E
F
promotore
forte
LTR
Elementi trasponibili hanno spesso promotori direzionati verso l’esterno
Che cosa possono fare i trasposoni saltando nel genoma?
Produzione di nuove varianti alternative di una proteina
Trasposone
Promotore
A
B
C
D
E
F
A
B
C
D
E
F
Genoma nucleare umano
(Nature, 431: 931-945, 2005)
(da: Molecular Biololgy of the Cell, Fig. 4.17)
La porzione codificante rappresenta l’1-2% del genoma (geni codificanti per
proteine, tRNA e rRNA.
La porzione non-codificante è costituita da sequenze uniche e sequenze ripetute.
Queste si suddividono in: 1) ripetizioni intersperse LINEs, SINEs, LTR, trasposoni a
DNA); e 2) ripetizioni in tandem (blocchi ripetuti in tandem dei centromeri e dei
telomeri, micro- e mini-microsatelliti, duplicazioni segmentali).
La distribuzione degli elementi ripetuti varia trai i cromosomi, con alcuni
43
cromosomi che contengono anche il 90% di DNA non codificante
Elementi trasponibili nell’uomo
Insersione di un elemento trasponibile nel gene che
codifica per l’antocianina



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)