IDENTIFICAZIONE DI NUOVE ANOMALIE GENICHE IN LLA PEDIATRICHE A
CARIOTIPO NORMALE O NON CLASSIFICATE GENETICAMENTE: ANALISI
INTEGRATA DEL PROFILO GENOTIPICO E DI ESPRESSIONE.
Bungaro S,1 Campo Dell'Orto M,2 Basso D,3 Zampieri M,3 Leszl A,2 Basso G,2 Biondi A,1 Te
Kronnie G,2 Bicciato S,3 And Cazzaniga G,1.
1 Centro Ricerca Tettamanti, Clinica Pediatrica Università di Milano-Bicocca, Monza, Italy
2 Laboratorio di Emato-Oncologia Pediatrica, Università di Padova, Padova, Italy
3 Dipartimento di Processi Chimici dell'Ingegneria, Università di Padova, Padova, Italy
Introduzione: Circa il 20% delle LLA pediatriche non possono essere classificate sulla base di
marcatori genetici rilevabili con tecniche di citogenetica classica o di biologia molecolare. Studi
recenti hanno dimostrato che l'analisi genomica dei polimorfismi a singolo nucleotide (SNP)
consente di rilevare con elevata risoluzione la perdita di eterozigosi (LOH), un fenomeno frequente
sia nelle LLA che nelle LMA, che può essere causata da variazioni del contenuto di DNA (delezioni
o amplificazioni geniche) o associata a disomia uniparentale (UPD).
Obiettivi: Questo studio è finalizzato all'identificazione di anomalie cromosomiche criptiche in
LLA pediatriche a cariotipo normale o non classificate geneticamente, attraverso l'analisi del profilo
genomico di LOH e di variazione del contenuto genico (CNC). Le caratteristiche genomiche del
clone leucemico verranno integrate con i profili di espressione allo scopo di meglio comprendere
l'elevata eterogeneità biologica e clinica della malattia.
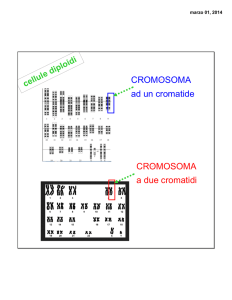
Metodi. Ad oggi sono stati analizzati 23 pazienti che hanno soddisfatto i seguenti criteri di
inclusione: a) diagnosi di LLA a precursori B; b) DNA index 1; c) negatività all'analisi di RT-PCR
per t(4;11), t(12;21), t(9;22), t(1;19); d) cariotipo normale o non eseguibile; e) profilo di espressione
non associato ad alcuno dei sottogruppi genetici di LLA. Tutti i pazienti analizzati sono arruolati nel
protocollo di terapia AIEOP-LAL2000. Il DNA genomico dell'esordio di malattia è stato analizzato
con la piattaforma Affymetrix GeneChip Mapping 100K SNP array allo scopo di identificare
regioni genomiche di LOH e CNC. In parallelo è stata eseguita l'analisi del profilo di espressione
attraverso la piattaforma Affymetrix HG-U133Plus2, nel contesto dello studio internazionale MILE
(Roche).
Risultati e conclusioni. Sono state riscontrate diverse anomalie, uniche o concomitanti. In 7 pazienti
è stata riscontrata la delezione del cromosoma 9p21, con una regione comune deleta in omozigosi
che coinvolge i geni CDKN2A e CDKN2B. Due pazienti hanno mostrato una delezione in
emizigosi del cromosoma 9p13. Una delezione in emizigosi del cromosoma 12p13.2 coinvolgente
circa 100 kb del gene ETV6 è stata riscontrata in due pazienti: in uno di essi associata ad una
traslocazione fra l'esone 1 di ETV6 ed un gene partner non noto, come indicato dall'analisi FISH;
nell'altro paziente concomitante con l'amplificazione intracromosomica del cromosoma 21, descritta
nell'1.5% delle LLA. Microdelezioni e regioni di UPD sono state riscontrate in casi singoli. Quattro
pazienti sono risultati iperdiploidi, mentre in tre casi, non sono state rilevate alterazioni genomiche
visibili.
L'analisi preliminare dell'integrazione dei profili genomici e di espressione ha mostrato la presenza
di concordanza tra le regioni delete e la “down-regolazione” dei geni compresi in tali regioni. La
presenza di regioni di alterata espressione non associate ad alterazioni genomiche potrebbe
permettere di individuare nuovi geni-malattia e di comprendere come i meccanismi di regolazione
del tumore siano correlati con le alterazioni genomiche delle cellule tumorali.