Programma per l’AA 2012/2013 del corso di
Elaborazione di Dati e Segnali Biomedici 1
Prof. GianCarlo Filligoi
Periodo delle lezioni: Marzo – Maggio 2013
Per il III° anno della Laurea Breve in Ingegneria Clinica e Biomedica
1)-SISTEMA DI ACQUISIZIONE DI DATI TRAMITE PC
1.1)-Considerazioni introduttive
Bioingegneria, Ingegneria medica e Ingegneria clinica
Il mercato delle apparecchiature medicali e possibili aree di ricerca della BME
La situazione Americana, Europea e locale della BME
1.2)-Con quali segnali abbiamo a che fare?
Origine dei segnali biomedici;
Classificazione dei biosegnali;
Principali caratteristiche dei segnali biomedici;
1.3)-Acquisizione di segnali tramite computer:
1.3.1)-Problematiche dei Bioelettrodi
Elettrodi per basse correnti (I0)
Ag-AgCl; Alpacca; Nichel
Elettrodi per alte correnti
Acciaio Inox (defibrillatori)
Platino-Iridio (pace-maker)
Piombo (reografia o impedenzometria)
Modello equivalente di coppie di elettrodi cutanei applicati sulla cute:
Interfaccia elettrodo elettrolita
Potenziale di mezza cella (HCP= Half Cell Potential)
Elettrodo standard all’idrogeno (SHE=Standard Hydrogen Electrode)
Rumore di elettrodo
Resistenza di trasporto di carica e capacità di doppio strato
Impedenza di Warburg
Resistenza dell’epidermide
Resistenza delle zone ad alta densità di corrente
Resistenza del mezzo interposto tra gli elettrodi
Schema equivalente semplificato
La polarizzazione degli elettrodi
Tempo di recupero della tensione di polarizzazione
Il rumore di elettrodo
L’impedenza di interfaccia
Principali connessioni di Trasduttori e Sensori:
Potenziometrica
Con Amplificatore operazionale
A ponte di Wheatstone;
1.3.2)-Analog Signal Conditioning:
DC offset removal e AC coupling;
Rapporto segnale/rumore (SNR) espresso in dB;
Banda Passante e Roll-off di un filtro;
Filtraggio PassaAlto, PassaBasso, Antialiasing;
Detezione di eventi e trigger; Peritriggering
Riduzione dei rumori di interferenza;
1.3.3)-Conversione Analogico-Digitale:
Modulo S/H (Sampling & Holding);
Quantizzazione: Tecniche dirette e indirette;
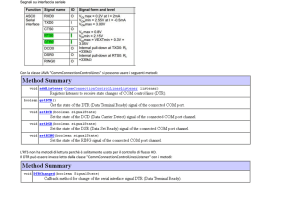
1.3.4)-Comunicazioni tra pC e I/O interface card:
Gestione delle porte di I/O: Control port, Status Port e Data Port;
Fasi della comunicazione: inizialization e data transfer phase;
Programmed I/O, Interrupt driven, DMA e shared memory;
1.3.5)-Cenni al Multiplexing:
Specifiche dei MUX
Il crosstalk
2)-METODI DI STIMA SPETTRALE--------2.1)-Introduzione:
Leakage e risoluzione spettrale:
Effetti delle finestre temporali sullo spettro stimato
2.2)-Alcuni concetti di base sui processi aleatori:
Stazionarietà ed ergodicità: statistiche d'insieme e temporali;
Teorema Wiener-Khinchine;
2.3)-Alcuni concetti di base sulla teoria della stima:
Qualità della stima: polarizzazione, efficienza e consistenza;
2.4)-Stime spettrali non parametriche o classiche:
Spettro campione e Periodogramma:
Utilizzo dell’algoritmo della FFT:
lo zero-padding
valutazione del BIN nella rappresentazione dello spettro;
Smoothing in frequenza (stima di Daniell);
Smoothing nel tempo senza sovrapposizione (stima di Bartlett);
Smoothing nel tempo con sovrapposizione (stima di Welch);
Correlogramma:
Stima polarizzata e non polarizzata della funzione di autocorrelazione;
2.5)-Stime spettrali parametriche:
Modelli AR, MA, ARMA:
Equazioni Yule-Walker e trattamento dei dati in modalità a blocco;
Algoritmo ricorsivo di Levinson-Durbin;
Confronto tra i vari tipi di modelli;
Scelta dell’ordine del modello (Criteri di Akaike, Rissanen e Parzen)
Cenni al Metodo di Burg;
3)-ANALISI DELLE SEQUENZE NUCLEOTIDICHE
3.1)-Cenni storici: dalla Società delle Scienze alla Società della Biologia
3.2)-Struttura della cellula
Gli eucarioti
I procarioti
Membrana citoplasmatica
Doppio strato fosfolipidico
Pori
Canali Ionici
Pompe elettrochimiche
Organelli e loro funzioni biologiche:
Mitocondri
Cloroblasti
Ribosomi
Reticolo endoplasmatico
Complesso di Golgi
Lisosomi
Perossisomi
Il Nucleo:
Membrana nucleare
Materiale genico nucleare
Il dogma della biologia genetica dal DNA alle proteine:
trascrizione e traduzione
3.3)Il Genoma
Genoma nucleare
Cellule diploidi e aploidi
I cromosomi:
Cromatidio e Fibre di cromatina
Nucleosomi
Doppia Elica DNA
Istoni
L’esperimento di Griffith del principio trasformante
La struttura spaziale del DNA secondo Watson e Crick
Genoma mitocondriale
3.4)- Gli Acidi nucleici
Il Nucleotide
Gruppo fosfato
Zucchero con 5 atomi di 12C
Base azotata
Acido DesossiriboNucleico (DNA):
Lo zucchero desossiribosio
Basi puriniche e pirimidiniche
Struttura primaria, secondaria, Terziaria, Quaternaria
Accoppiamento delle basi azotate
La doppia elica
Legami forti e deboli nella molecola del DNA
Eliche antiparallele
Direzioni 3’-5’ e 5’-3’
Acido RiboNucleico (RNA):
Lo zucchero Ribosio
I 7 tipi di RNA
Struttura primaria, secondaria e terziaria del RNA
Gli elementi fondamentali della struttura secondaria
L’energia libera di legame delle basi accoppiate
Funzioni di mRNA, t-RNA e r-RNA nella trascrizione e traduzione
Esoni, introni e splicing
Il codice genetico
3.5)-Trattamento statistico delle sequenze genomiche e proteomiche
Allineamento delle strutture primarie
Punteggi (score) di somiglianza e penalità per i gaps
Le banche dati
Le percentuali di dinucleotidi (GC, AT, ..)
3.6)-Necessità di “moltiplicazione” dei dati biologici
In-vivo: DNA ricombinante
Enzimi di restrizione
RNA-polimerasi
Frammenti di restrizione
Vettore di clonazione
Plasmide
In-vitro: PCR (Polymerase Chain Reaction)
Gli inneschi (primer)
Denaturation, Annealing ed Elongation
3.7)-Sequenzamento del DNA
Southern/Northern Blotting
Elettroforesi su gel di agarosio
Blotting su foglio di nitrocellulosa
Probes marcati radioattivamente per l’ibridazione
Metodo dideossi di Sanger
3.8)-Strategie di sequenzamento
Metodo dei Contig
Cenni al Metodo shotgun
3.9)-Elaborazione di segnali genomici
Allineamento di sequenze e punteggi delle somiglianze
Senza gap e senza esclusione di basi nucleotidiche
Con GAP e/o esclusione di nucleotidi
Ricerca di Motif Finding tramite intercorrelazione/convoluzione
Il tool di Bioinformatica di Matlab
Strategie di codificazione delle sequenze genomiche
Con numeri reali
Con numeri complessi
I cammini casuali (DNA walks)
4)- RISONANZA MAGNETICA NUCLEARE
4.1)-Acquisizione di Immagini Biomediche
Cenni ai segnali bidimensionali e relativi concetti matematici
Cenni alle Trasformate di immagini: Trasformata di Fourier
4.2)-Principi fisici della risonanza magnetica
Le proprietà dei nuclei atomici
Massa (Numero atomico, Numero di protoni, Numero di protoni)
Carica elettrica
Spin
L’esperimento di NMR
Saturation-Recovery
Allineamento parallelo ed antiparallelo in presenza di campi magnetici esterni
Il TESLA come unità di misura del campo magnetico
Il salto di energia e la radiofrequenza
Il F.I.D. (Free Induction Delay)
Trasformata di Fourier del FID
Legami delle grande spettrali con quelle fisiche
Sommazione degli scan per incremento SNR
Tempo di rilasciamento spin-spin T1
Tempo di rilasciamento spin-lattice T2
Relazione di Larmour
Le frequenze di risonanza dei nuclei atomici
4.3)-Spettroscopia a Risonanza Magnetica Nucleare (MRS Magnetic Resonance Spectroscopy)
Il chemical shift
Rappresentazione nella scala
Gli spettroscopi a RM
Schema a blocchi dello spettroscopio
Le bobine del campo di induzione principale
Omogeneità del campo magnetico
La deriva del campo magnetico ed il deuterio
Bobine di compensazione (shimcoils)
Effetti delle shimcoils sullo spettro NMR
Il probe e lo spinning probe
Taratura del probe
Acquisizione del FID
Importanza del Tempo di acquisizione
Le oscillazioni (sinc wiggles)
Il numero di punti durante il tempo di acqusizione
Il guadagno del ricevitore e l’effetto di clipping
Rivelazione del FID in quadratura
Correzione degli errori di fase
Lo zero-filling e la sua influenze sul BIN
Relazione con la risoluzione spettrale
Data windowing ed apodisation con finestra esponenziale
Correzione della linea di base (isoelettrica)
La matematica dei picchi di MRS
La correlazione con Lorentziane
Posizione, ampiezza ed altezza
L’integrale dei picchi
Detezione dei massimi
Parametri che influenzano MRS
Frequenza dello spettrometro (Spectrometer Frequency [sfrq])
Durata dell’impulso di eccitazione a RF (Pulse Width [pw])
Angolo dell’impulso
Angolo di Ernst
Tempo di acquisizione (Acquisition Time [at])
Numero di punti in at (Number of Points [np])
Sweep (Spectral) Width [sw]
Ritardo di ripetizione (Recycle Delay [d1])
Numero di sprove o scansioni (Number of trials (scans) [nt])
Applicazioni della spettroscopia
Spettri 1H-NMR
Spettro dell’acqua tissutale e del grasso
13
Spettri C-NMR
Spettro dell’- e –glucosio nel fegato
31
Spettri P-NMR
Il metabolismo dell’ATP ed il suo spettro in-vivo
Lo zero della scala
Tetrametilsilano (TMS) e fosfocreatina (Pi)
4.4)-Tomografia a Risonanza Magnetica (Magnetic Resonance Imaging, MRI)
La misura dei tempi di rilassamento T1 e T2
La ripetizione dell’esperimento di Saturation-recovery per valutare T1
Importanza del Tempo di Ripetizione (TR)
La sequenza Spin-echo per valutare T2
Gradienti magnetici e loro utilizzo per la formazione delle immagini
La mappatura spazio-frequenza
Selezione del piano di tomografia
Eccitazione selettiva (Selective excitation)
Il piano nullo (Null plane)
Ricostruzione delle immagini con la Back-Projection e la 2-D Fourier Trasf.
L'esperimento di NMR secondo la fisica classica
Misura dei tempi di rilasciamentoT1 e T2:
Sequenza Inversion-recovery (180°-TI – 90°)
Esperimento spin-echo (90° - TE/2 – 180°)
Esperimento di Carr-Purcell e del Multiplanar Excitation
Influenza dei parametri dell'esame NMR sull'intensità e il contrasto dell'immagine
4.5)-Cenni ai problemi costruttivi dei tomografi NMR
4.6)-Influenza sul MRI dei parametri di immagine e delle proprietà tissutali
5)-PRIMI PASSI NEL SOFTWARE MATLAB
5.1)-Interfaccia utente:
Command window
Workspace
Current directory
Help in linea
I toolbox
5.2)-Strumenti di base
Come leggere/scrivere files
Come disegnare grafici
Plot
Sub-plot
Hold-on Hold-off
Arrays e matrici
Grafici 3-D e a colori
5.3)Graphic Unit Interface (GUI)
Push-button
Axes
CallbacK
5.4)-Tecniche segnalistiche di verifica dei programmi
Segnali campione per eseguire test su algoritmi implementati di signal processing
Onde sinusoidali e loro generazione
Onde quadre e loro generazione
Onde triangolari e loro generazione
Segnali aleatorii (random noise) e loro generazione
5.5)-Verifica in tempo ed in frequenza della correttezza di un algoritmo in MATLAB
Il debugging
L’esecuzione step-by-step


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)



![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)