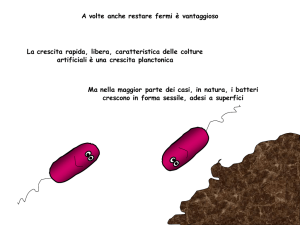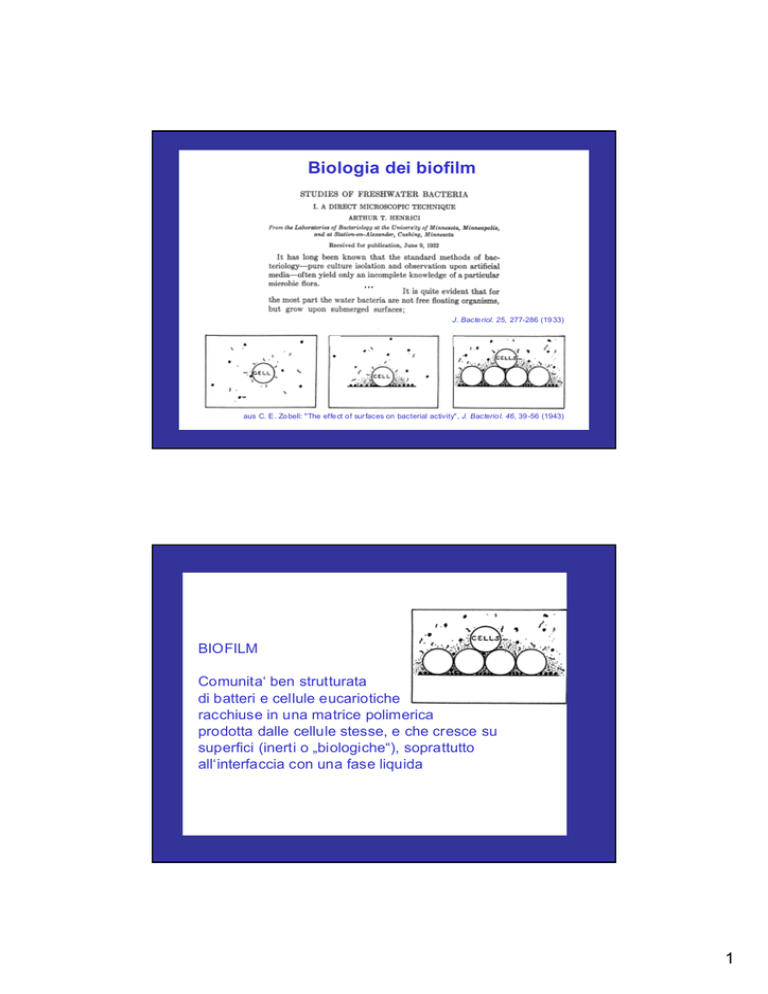
Biologia dei biofilm
...
J. Bacte riol. 25, 277-286 (19 33)
aus C. E . Zo bell: "The effe ct o f sur faces on bacterial activity", J. Bacterio l. 46, 39 -56 (1943)
BIOFILM
Comunita‘ ben strutturata
di batteri e cellule eucariotiche
racchiuse in una matrice polimerica
prodotta dalle cellule stesse, e che cresce su
superfici (inerti o „biologiche“), soprattutto
all‘interfaccia con una fase liquida
1
Importanza dei biofilm microbici
• Biofilm come fattore di virulenza: infezioni da
biofilm più virulente, più tendenti a
cronicizzarsi e più resistenti alle terapie
• Batteri adesi ad una superficie solida o
“flocculati” sono utilizzati preferenzialmente in
bioreattori industriali e nella depurazione
delle acque di scarico
• I biofilm sono ubiquitari
Un caso “reale” di biofilm misto come osservato in microscopia elettronica
2
Adesione
Colonizzazione
Maturazione
Interazioni fisico-chimiche
Pili
EPS: produzione/organizzazione
(idrofobicità, carica elettrica)
Polisaccaridi (alginato)
Quorum sensing
Flagello
Fattori di aggregazione cellulare
Proteine di membrana
(curli, ecc…)
esterna,
parete cellulare, LPS…..
La formazione di biofilm è correlata con la
coniugazione ed il trasferimento orizzontale
di materiale genetico
Pilus
Due effetti dell’espressione
del pilus:
consente la coniugazione
batterica e stimola la
produzione di biofilm
3
Trasferimento genico
orizzontale
• Permette di trasferire plasmidi coniugativi, (evtl.
con trasposoni annessi) o frammenti
cromosomali
• Tra i geni trasmessi, resistenza ad antibiotici ed
enzimi di degradazione di sostanze organiche
• Considerato come un’efficiente strategia di
adattamento
• L’alta frequenza di coniugazione nei biofilm
rappresenta quindi un vantaggio di questa forma
di organizzazione cellulare
Genomic analysis of biofilm-dependent gene expression
through microarray analysis
Biof.
Single
cells
4
Il numero di geni che si
esprime in maniera
diversa in singole
cellule e nei biofilm
comprende una %
molto alta del genoma
(10-30%).
Tutti coinvolti nella
formazione del
biofilm?
L’eterogeneità di espressione genica riflette anche (soprattutto?)
le diverse condizioni fisiologiche dei batteri in un biofilm complesso
Aerobi,
Crescita + veloce
[O2],
Nutr.
Anaerobi,
Crescita + lenta
5
Quorum sensing: da curiosità
evolutiva a processo biologico
globale
Eurpymna scolopes
Vibrio fischeri
Quorum sensing: espressione genica dipendente
dalla concentrazione cellulare
Molecola segnale
Molecola segnale
Attivatore
trascrizionale
Attivatore
trascrizionale
Gene target
Gene target
Bassa concentrazione
cellulare
Alta concentrazione
cellulare
6
La comunicazione intercellulare da quorum sensing
è favorita in biofilm piuttosto che in colture liquide
Cultura liquida: 108-109 cfu/ml
Molecole segnale (in Gram negativi):
acil-omoserin-lattoni (AHL o HSL)
Quorum Sensing in Pseudomonas aeruginosa:
Il sistema lasR/lasI e rhlR/rhlI costituiscono una cascata regolativa
C12-HSL
C4-HSL
7
Geni regolati da Quorum Sensing in Pseudomonas aeruginosa
Tre classi funzionali principali: fattori di virulenza, sintesi di EPS, uptake del ferro
Studi genomici stimano al 5-6% la percentuale di geni regolati da QS
Il cluster rhlR/rhlI è così chiamato per il suo
ruolo nella biosintesi dei rhamnolipidi
Rhamnolipidi: un esempio di biosurfattanti e i responsabili della
“gliding motility”
8
I promotori QS-dipendenti sono spesso co-regolati
da meccanismi legati a stimoli ambientali/fisiologici
(sito di legame per LuxR)
Geni quorum sensing-dipendenti determinano la
“struttura ordinata”all’interno di un biofilm batterico
9
La biosintesi degli AHL ha come unico scopo
la creazione di una molecola segnale
Le proteine regolatrici della famiglia LuxR
sono tipici regolatori di risposta
10
11
Il QS è direttamente coinvolto in meccanismi
di patogenesi nei mammiferi
Gli induttori (molecole segnale) appartengono a classi chimiche diverse
12
Un caso a sé: gli
Streptomyces
S. coelicolor
Streptomiceti/Attinomiceti:
Batteri (Gram +) del suolo
(30 °C temperatura ottimale di crescita)
Organismo multicellulare
(Uniche forme monocellulari: spore e protoplasti)
Sporigeno (su substrati solidi, spore in circa 7 gg)
Crescita come micelio in terreno liquido
Principale produttore di antibiotici
Geni regolatori:
Numero di ORF predette
S. coelicolor
E. coli
B. subtilis
S. cerevisiae
7825
4289
4099
6203
65 fattori sigma
TCRS:
85 sensor kinases
79 response regulators
“A factor”:
OH
O
O
O
identificato in S. griseus
Mutanti incapaci di produrre A factor sono Spo-, StrS and StrA factor aggiunto esternamente ripristina il fenotipo originario
A factor lega il repressore ArpA, inibendone l’attività
E’ in grado di agire a concentrazioni nM
Coinvolto nella produzione di numerosi composti antimicrobici
13
OH
A factor, S. griseus
O
O
O
OH
O
O
OH
S. bikiniensis, S. cyanofuscatus,
S. viridochromogenes
OH
S. virginiae
O
O
OH
γ-butirrolattoni in
Streptomyces
OH
S. bikiniensis, S. cyanofuscatus
O
O
OH
OH
S. virginiae
O
O
OH
OH
S. virginiae
O
O
OH
OH
S. virginiae
O
O
OH
OH
S. virginiae
O
O
OH
OH
Streptomyces sp. FRI-5
O
O
OH
Quorum sensing in batteri Gram +
• Molecole segnale: peptidi (modificati)
• Processi biologici regolati da QS:
Coniugazione, virulenza, produzione di
enzimi extracellulari, adesione cellulare,
formazione di biofilm
Competenza
Produzione di peptidi antimicrobici
14
Peptidi segnale (auto-induttori)
in batteri Gram positivi
Sistemi agr e com:
una struttura comune
agrD
(virulenza in S. aureus)
comX
(competenza in B. subtilis)
15
Secrezione e uptake del peptide
AgrD
AgrC/A: sistema a due componenti
AgrB: secrezione/modificazione
AgrC:
sensore
PO4
A
AgrA regolatore di risposta
Il sistema agr si autoregola tramite un
RNA (hld)
hld modula l’attività di AgrA
e di altri regolatori globali
16
Schema generale del QS in Gram +
Confronto tra processi di QS in
Gram - e Gram +
Molecole segnale o “feromoni”
Feromoni: HSL
Libera diffusione attraverso
la membrana
Sistemi a due componenti
(entrambi citoplasmatici)
Feromoni: peptidi
Secrezione attiva
Sistemi a due componenti:
Un sensore inserito nella membrana
Un regolatore citoplasmatico
17
La “doppia vita” dei peptidi segnale:
attività antimicrobica
• Un vasto gruppo di peptidi segnale
presenta un’attività antimicrobica
(generalmente mediata da
destabilizzazione della membrana)
particolarmente spiccata contro altri
batteri Gram+ (plantaricina, enterocina,
lantibiotici)
• Particolarmente presenti in batteri lattici
(importanti per biotecnologie alimentari)
Un peptide antimicrobico “utile”: la
nisina
Prodotta da lactobacilli; espressione richiede alta densità cellulare
(QS) unitamente a fase stazionaria di crescita.
Ampio spettro d’azione contro Gram + (a concentrazioni nM)
Prodotta spontaneamente (o aggiunta) nella preparazione dei formaggi
18
Se ricordiamo lo schema generale del
QS in Gram+…….
…..possiamo comprendere il sistema
di produzione e processamento della
nisina!
Oltre al sistema di modificazione/esporto e di trasduzione del segnale, i
ceppi produttori di nisina posseggono geni per l’immunità
19
Biosintesi e processamento dei
lantibiotici
• Auto-regolazione (+ regolazione da
segnali ambientali e fisiologici)
• Largo numero di geni coinvolti in:
modificazione, esporto, trasduzione del
segnale ed immunità (da 10 a 18)
• Interesse come “nuovo” agente
antimicrobico e per applicazioni
biotecnologiche (ind. alimentare)
Anche i geni dell’immunità sono sotto
il diretto controllo della nisina
20
Nisin-controlled expression (NICE)
Espressione di geni letali tramite
induzione con nisina
Enzimi intracellulari di
lactobacillo sono essenziali
nella maturazione dei formaggi
La lisi “controllata” potrebbe
accelerare e standardizzare
questo processo
21
Referenze biofilm/QS
•
Winzer, Hardie, and Williams. Bacterial cell-to-cell communication: sorry,
can’t talk now — gone to lunch! Current Opinion in Microbiology 2002,
5:216–222
•
Withers, Swift, and Williams. Quorum sensing as an integral component of
gene regulatory networks in Gram-negative bacteria. Current Opinion in
Microbiology 2001, 4:186–193
•
O‘ Toole and Kolter. Flagellar and twitching motility are necessary for
Pseudomonas aeruginosa biofilm development. Mol Microbiol. 1998,
30:295-304
•
O‘ Toole, Kaplan, and Kolter. Biofilm formation as microbial development.
Annu Rev Microbiol. 2000, 54:49-79.
•
Davies, Parsek, Pearson, Iglewski, Costerton, and Greenberg. The
involvement of cell-to-cell signals in the development of a bacterial
biofilm. Science 1998, 10:295-298
•
Kleerebezem. Quorum sensing control of lantibiotic production; nisin and
subtilin autoregulate their own biosynthesis Peptides 2004, 25:1405-1414
•
•
•
Web: http://www.erc.montana.edu/
http://gasp.med.harvard.edu/
http://www.nottingham.ac.uk/quorum/
(University of Montana)
(Kolter lab)
(Quorum sensing network)
22