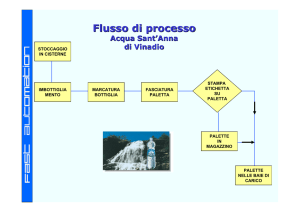
DIAPOSITIVA 1
L’obietivo principale del presente progetto consiste nell’individuare i diversi mecanismi genetici
che controllano la risposta della pianta allo stress idrico in frumento duro, mediante lidentificazione
dei loci per i tratti quantitativi (QTLs) correlati, in particolare quelli localizzati sul cromosoma 4;
questo studio permetterà di incrementare e stabilizare la produttività del frumento duro in presenza
di condizioni di siccità in Europa e nelle regioni del mediterraneo. Il progetto verra articolato
attraverso uno studio consistente in mappatura di QTLs, studio delle genetiche di associazione,
identificazione di nuovi geni e sviluppo di marcatori molecolari da applicare in esperimenti di
MAS. Il presente progetto e suddiviso nelle seguenti linee di ricerca:
Linea 1: Analisi di RILs per l’identificazione e la mappatura di QTLs associate a resistenza
a stress idrico
Linea 2: Analisi di popolazioni derivanti da zone aride ed umide per l’identificazione e la
mappatura di selective sweeps con conseguenti analisi di linkage disequilibrium
Linea 3: Sequenziamento delle regioni QTL di interesse mediante selezione clonale di
libreria BAC di frumento duro
Linea 4: Verifica dei geni candidati attraverso quantitative RT-PCR
DIAPOSITIVA 2
LINEA 1: Analisi di RILs per l’identificazione e la mappatura di QTLs associate a resistenza
a stress idrico
Materiale di partenza
Il materiale genetico è già disponibile ed è stato accuratamente selezionato in modo tale da
combinare tratti che in passato sono stati riconosciuti come importanti nella tolleranza alla siccità.
Una popolazione mappante disponibile è Haurani X Cham 1 (Ha/Ch) (97 recombinant inbred lines
(RILs)), Haurani rappresenta la landrace con il maggior grado di tolleranza alla siccità nelle zone
aride del Mediterraneo e presenta un alto potere di penetrazione delle radici nei suoli con strati
superiori induriti e poco permeabili. Cham 1 presenta invece una notevole stabilita produttiva nelle
regioni mediterranee.
Work flow
Allevamento delle varietà di frumento
Scelta dei QTLs e dei marcatori molecolari per resistenza a stress idrico:
Analisi di transcrittomi e gene network
Estrazione di DNA e quantificazione
Amplificazione PCR
Sequenziamento
Analisi statistica
Analisi di polimorfismi e segregazione di marcatori polimorfici
DIAPOSITIVA 3
LINEA 2: Analisi di popolazioni derivanti da zone aride ed umide per l’identificazione e la
mappatura di selective sweeps e analisi di linkage disequilibrium
Materiale di partenza
Verranno impiegate landraces suddivise in 9 popolazioni di frumento duro tetraploide appartenenti
alla specie Triticum turgidum subsp. Durum e derivanti da 3 regioni etiopiche (Tigray, Gonder and
Shewa) con differenti condizioni climatiche. Le 3 regioni dalle quali deriva il materiale analizzato,
sono caratterizzate da tre differenti condizioni climatiche e geologiche. Tigrai (Tigrè), con un
estensione di 50.286 km2, consiste in un altopiano collocato a 3000m sopra il livello del mare, con
un suolo scuro e con meno di 900 mm di precipitazioni annuali. La regione di Gonder (Gondar) con
una superficie di 40,27 km2, è caratterizzata da un suolo più argilloso, con un range di altitudine da
1.750 a 2.050 m a.s.l., con 1,200 mm di precipitazioni annuali ristrette al periodo intercorrente tra
maggio e settembre ed un secondo periodo compreso tra aprile è metà maggio derivanti
dall’incontro tra i venti provenienti da nord-est e da sud-est. La regione di Shewa (Scioa) è parte di
un altopiano montagnoso che definisce l’area centrale dell’Etiopia; con un altitudine compresa tra
1700 e 2500 m a.s.l., Shewa è principalmente arida, con un suolo tendenzialmente argilloso e con
precipitazioni annuali di circa 1000 mm comprese tra ottobre e marZO.
DIAPOSITIVA 4
Obiettivi
Work flow
Scelta dei marcatori molecolari:
analisi di trascrittomi di frumento duro, analisi delle mappe genetiche e gene network su
cromosoma 4
Estrazione del DNA e quantificazione
Amplificazioni PCR con marcatori SSR localizzati sul cromosoma 4
Sequenziamento
Analisi statistiche
Identificazione e mappatura di selective sweeps
Sviluppo di mappe di linkage
in ICARDA e IITA.
DIAPOSITIVA 5
MARCATORI SSR CON LOCALIZZAZIONE CASUALE SUL GENOMA: 28 MARKERS
WMC 24
WMS 493
WMS 459
BARC 213
WMS 181
WMC256
BARC 8
WMC 516
WMS 518
WMS 124
WMS 610
WMS 219
WMC 177
CFA 2091
CFA 2049
WMC 170
WMS 375
WMS 260
CFA 2278
WMS 205
WMS 46
WMS 120
WMS 291
WMS 302
WMC 532
WMS 159
WMS 5
WMS 371