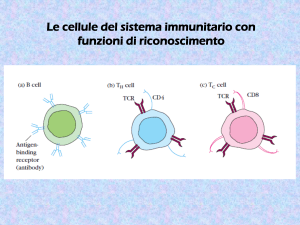
Riconoscimento dell’antigene
Risposta immune adattativa
• La risposta immune innata è molto efficiente
ma non sempre riesce ad impedire il
diffondersi dell’infezione
• È essenziale quindi che subentri la risposta
adattativa specifica per il patogeno che sta
attaccando in quel momento il nostro
organismo
BCR (B-cell receptor) ed anticorpi
• BCR: espresso sulla superficie dei linfociti B
• Anticorpo: secreto dalle plasmacellule
• Le due molecole hanno la stessa struttura, gli
anticorpi sono la forma secreta del BCR
• Ci sono diversi tipi di anticorpi (classi),
struttura di base simile
Struttura degli anticorpi
IgG
• Forma a Y, 3 porzioni grossolanamente uguali per dimensioni
• Regioni Variabili: coinvolte nel riconoscimento dell’antigene
• Regione Costante: interagisce con effettori cellulari e
molecolari della risposta anticorpale
• 5 classi di anticorpi: IgM, IgD, IgG, IgA e IgE, diversi nelle loro
regioni costanti
• Esempio struttura IgG: 150kd, composta da 4 molecole
• Due catene pesanti (heavy chain), di 50kDa, legate tra solo
da ponti disolfuro
• Due catene leggere (light chain), di 25kDa, legate alla
catena pesante da un ponte disolfuro
• Due tipi di catene leggere: k and l, con funzione
equivalente
• Una IgG possiede due siti leganti l’antigene per molecola, il che
aumenta la forza totale dell’interazione antigene-anticorpo
(avidità)
• Il BCR è identico come struttura ma la regione c-terminale della
catena pesante (regione costante) contiene una sequenza
idrofobica che lo ancora alla membrana
Il dominio Ig
• Lungo circa 110 aa
• Catene leggere: composte da due domini Ig
• Catene pesanti: composte da quattro domini
Ig
• Il primo dominio amino-terminale di
entrambe le catene è caratterizzato da
grande variabilità in funzione dell’anticorpo,
gli altri domini sono conservati per ciascuna
classe di anticorpo
• VH e VL
• CL e CH1-3
Frammenti proteolitici degli anticorpi
• Varie proteasi possono digerire in maniera limitata le molecole di anticorpi generando
frammenti che contengono porzioni diverse della molecola
• Fab (fragment antigen binding); Fc (fragment crystallizable)
• F(ab’)2: le due braccia leganti l’antigene restano unite tramite il ponte disolfuro
• Gli anticorpi sono molecole estremamente flessibili, il che consente variabilità nella
formazione del complesso antigene-anticorpo
Interazione antigene-anticorpo
•
•
•
•
La variabilità delle regioni variabili si concentra in alcune zone dette regioni ipervariabili
(HV1-3)
Le HV si intervallano con le regioni “framework” (FR1-4) meno variabili, più importanti nel
folding vero e proprio
Le sei regioni ipervariabili formano il sito di legame per l’antigene, e sono dette anche
“complementarity-determining regions” (CDRs), perchè sono complementari alla superficie
dell’antigene
Poichè le CDR sono formate dalle due catene, la specificità anticorpale può essere influenzata
anche dalla combinazione delle due diverse catene
Struttura del legame antigene-anticorpo
•
•
•
•
La superficie legante l’antigene comprende le CDR delle due catene (H ed L)
Un piccolo antigene (gli antigeni di piccole dimensioni vengono chiamati apteni e ne parleremo
più avanti) di solito interagisce con una superficie (un “solco”) che si genera fra le due catene
Molecole più grosse (regioni di proteine) possono avere multiple superfici di interazione molto
più estese
• Il tipo di legame non è sempre “a tasca”, ci possono essere anche delle protrusioni
dell’anticorpo che entrano all’interno di cavità presenti nell’antigene
La regione dell’antigene riconosciuta dall’anticorpo viene detta epitopo
• Lineare
• Conformazionale
Riconoscimento dell’antigene dalle
cellule T
• Le cellule T riconoscono gli antigeni solo nel
contesto della superficie cellulare “self”
– L’antigene deve essere “presentato” da cellule
dello stesso individuo
– La presentazione avviene ad opera di speciali
glicoproteine che si associano all’antigene –le
molecole del complesso maggiore di
istocompatibilità -MHC
Il recettore delle cellule T (T cell
receptor)
• Composto da due subunità dette TCRa e
TCRb, unite da un ponte S-S
– Una piccola parte delle cellule T ha un TCR
formato da due diverse subunità, la funzione di
questo subset di cellule non è chiara e non ce ne
occuperemo
• Il TCR ricorda come struttura il frammento Fab
degli anticorpi
TCR
•
•
•
•
•
Regione variabile
Regione costante
Linker contenente il ponte S-S
Regione transmembrana+
Coda citoplasmatica
• Il riconoscimento dell’antigene dal TCR è diverso da
quello effettuato dagli anticorpi
• Le cellule T rispondono a corte sequenze
peptidiche che appartengono a regioni diverse di
una determinata proteina
• A volte queste sequenze sono all’interno della
proteina
• Necessità del “processing” della proteina
• La corta sequenza peptidica che deriva dalla
proteina “estranea” viene riconosciuta solo quando
legata ad una molecola di classe MHC
Due classi di molecole MHC
Classe I
MHC di classe I
• Due catene
• Alpha (tre regioni, 1-3)
• Beta2-microglobulina
• Alpha1-2 formano la superficie legante il peptide da
presentare
• Molecole polimorfiche (centinaia di alleli, ne
parliamo dopo), e questo influenza la natura del
peptide presentato
• La ß2-microglobulina non è polimorfica
MHC di classe II
• Due catene (alpha e beta, entrambe sono
transmembrana)
• La tasca per il peptide si forma fra catene alpha e
beta
• Molecole polimorfiche
Classe II
Il legame MHC-peptide antigenico
• Le molecole di classe MHC devono essere in
grado di interagire con peptidi derivati da una
grande varietà di proteine diverse
• Una caratteristica del legame MHC-peptide è che
le molecole MHC sono più stabili
conformazionalmente dopo l’interazione con il
peptide
• I peptidi che legano a MHC di classe I sono corti
(8-10aa), ad MHC di classe II sono più lunghi
anche se di solito rientrano fra 13-17aa
Il legame MHC-peptide antigenico
Il legame TCR-MHC-peptide
• Il TCR stabilisce contatti sia con il peptide, che con la molecola di MHC
• Da cinetiche di binding si può ipotizzare che in una prima fase MHC-TCR sia
l’interazione predominante, per poi passare ad una seconda fase di più fine
interazione con il peptide antigenico
Il ruolo dei co-recettori CD4 e CD8
• CD4: presente sui linfociti T helper
• CD8: presente sui linfociti T citotossici
• CD4-CD8 si associano con I TCR e
interagiscono con regioni invarianti delle
molecole MHC
• Il legame con CD4-CD8 è essenziale per le
cellule T, altrimenti non si verifica una risposta
efficace in seguito all’interazione con
l’antigene
CD4 e CD8
•
•
CD4 è un’unica catena, che interagisce con MHC di classe II, lontano dal sito di legame con il
TCR in modo da permettere il legame simultaneo con il complesso
• Porzione intracitoplasmatica di CD4 lega la chinasi Lck
CD8 è omo- o eter-dimerico, interagisce con MHC di classe I, in modo simile a CD4-MHC II
Espressione di MHC
• MHC di classe I presentano peptidi patogeni
(specie virus) a linfociti T citotossici
– Tutte le cellule nucleate esprimono MHC I, anche se in
quantità differente
• MHC di classe II sono legati a linfociti T helper,
che devono assistere altre cellule del sistema
immune
– Espress quindi specie su cellule B, macrofagi e cellule
dendritiche
• Espressione di MHC è regolata da citochine
(specialmente gli interferoni)
La generazione dei recettori x
antigeni
Il repertoire anticorpale
• Il numero totale di anticorpi disponibili per
ogni individuo è altissimo: almeno 100 miliardi
di molecole diverse, e forse molte di più
• Questa diversità è solo minimamente
garantita da diversità nella linea germinale,
mentre è principalmente generata da
riarrangiamenti genici nelle cellule somatiche
che producono I recettori antigenici
Le regioni variabili sono generate a
partire da diversi segmenti genici
• Regioni variabili delle catene pesanti e leggere sono generate dalla
ricombinazione somatica di diversi segmenti genici
• Catene leggere:
– Due segmenti:
• Segmento V (I primi 100 aa circa)
• Segmento J (fino a 13 aa)
• Catene pesanti:
– Tra I segmenti V e J c’è il segmento D
• Produzione di una catena leggera completa di IgG
– Congiunzione dei segmenti V-J x riarrangiamento genico
– Questo porta VJ >> vicino alla regione C, quindi il trascritto che si forma genera
la catena leggera per RNA splicing
• Produzione di una catena pesante di Ig
– Produzione di DJ per riarrangiamento
– Produzione di VDJ per riarrangiamento
– Catena completa come per quella leggera (x splicing)
Le regioni variabili sono generate a
partire da diversi segmenti genici
I diversi segmenti genici della regione
V sono presenti in multiple copie
• Esistono multipli segmenti genici
• Molti sono pseudogeni non funzionali (scarsa
pressione genica vista la ridondanza, che
favorisce la variabilità e mutazioni)
• Per ogni catena immunoglobulinica (H e L, questa
ultima divisa in k e l) c’è un distinto locus genico
• Importante : il locus per la regione C della catena
pesante è organizzato in modo da contenere
diverse regioni C che corrispondono alla diversa
classe anticorpale
L’organizzazione germinale dei loci per
le varie catene delle Ig
Come è regolata la ricombinazione?
• I complessi riarrangiamenti genici necessari
devono avvenire correttamente nello spazio e
nel tempo
• Sequenze conservate: “recombination signal
sequences” (RSS), blocchi di 7 nucleotidi
+spacer+9 nucleotidi che si trovano in
adiacenza dei vari segmenti genici
• La lunghezza degli spacer determina
l’accoppiamento dei vari segmenti
Come è regolata la ricombinazione?
Enzimi necessari per la ricombinazione
•
•
•
Nella maggior parte dei casi, I due segmenti che vanno incontro
a riarrangiamento sono nello stesso orientamento sul DNA, il
risultato della ricombinazione è un circolo di DNA
extracromosomale che contiene la regione escissa
Gli enzimi che effettuano la ricombinazione sono chiamati V(D)J
ricombinasi
• Specifici per I linfociti: RAG-1 e RAG-2
• Quando sono espressi artificialmente in cellule non
linfocitarie le rendono in grado di fare
riarrangiamenti
• Protein coinvolte normalmente nel processo di riparo del
DNA
• DNA –PK
• Artemis
• DNA ligase IV
Il ruolo di questi enzimi è stato stabilito studiando le loro
mutazioni
• Nell’uomo la sindrome di Omenn è causata da mutazioni
di RAG1 o RAG2, con assenza di linfociti B circolanti e
problemi ai linfociti T, mutazioni in geni del DNA repair
causano altri tipi di malattie complesse
• Nel topo mutazioni nei geni della ricombinazione danno
origine ai topi SCID (severe combined immuno deficiency)
Come si genera la diversità del
repertoire anticorpale?
• Meccanismo combinatorio
– Riarrangiamento di segmenti diversi
– Utilizzo di catene diverse
– Insieme I due meccanismi garantiscono circa 2,000,000 di
combinazioni diverse
• Meccanismo di riarrangiamento impreciso
– Alcuni nucleotidi possono venire aggiunti o sottratti dagli
enzimi ricombinativi nel sito di giunzione, generando
cambi di sequenza proteica
• Meccanismo di ipermutazione somatica (lo spieghiamo dopo)
• Tutti insieme questi processi garantiscono > 1011 diversità
anticorpale
Meccanismi di riarrangiamento del
TCR
•
•
•
•
Il riarrangiamento avviene nel timo
Alpha locus: segmenti V e J
Beta locus: segmenti V, D e J
Simile nella struttura ai loci per le Ig
Meccanismi di riarrangiamento del TCR
• Meccanismo simile a quello delle Ig
• Sono coinvolti gli stessi enzimi
• Dal momento che la C-region delle IgG svolge > funzioni effettrici, c’è maggiore
variabilità nella C region delle Ig rispetto ai TCR, che contengono solo 1
segmento genico per C-alpha e due segmenti genici per C-beta
Variazione strutturale nella regione
costante delle Ig
• La cellula B naïve sfrutta solo I segmenti genici per due
regioni C (Cm e Cd, localizzate in vicinanza della regione
V)
• Questo porta all’espressione di IgM ed IgD
• Durante l’attivazione delle cellule B avviene il
cosiddetto “class switching”, ed abbiamo la produzione
di altre Ig con differenti funzioni
• La regione costante è importantissima per molte
funzioni effettrici degli anticorpi
– Legame a recettori cellulari
– Legame al complemento
– Trasporto (p.es. latte, secrezioni come saliva e lacrime…)
I diversi isotipi di Ig
Organizzazione genica della regione C
• La produzione di IgM o IgD avviene mediante la trascrizione di un lunga molecola che
viene poi sottoposta a splicing per generare la Ig matura
Espressione di IgM ed IgD
Produzione della forma di membrana vs la forma
solubile per uso di trascritti alternativi
Forme polimeriche di Ig
• IgM: come pentameri
• IgA: come dimeri (nelle secrezioni)
• Le regioni C di queste Ig contengono una coda di 18aa
essenziale per la polimerizzazione che contiene un residuo
di cisteina
• Una catena di 15kda chiamata catena J (niente a che fare
con il segmento genico J!) promuove la polimerizzazione
legandosi alla cisteina della coda delle regioni C
• La polimerizzazione potrebbe essere utile per il legame
delle Ig ad epitopi ripetitivi come quelli presenti sulle
superfici batteriche
• Inoltre l’aumento del numero di siti/molecola (10 per IgM)
porta ad un legame molto >>> con l’antigene (maggiore
avidità)
Struttura delle forme polimeriche di Ig
La diversificazione del repertoire
anticorpale dopo l’attivazione
• I meccanismi di diversificazione discussi finora –
riarrangiamenti genici- avvengono durante la
maturazione delle cellule B
• Quando la cellula B riconosce l’antigene va
incontro ad attivazione, inducendo nuovi
meccanismi di diversificazione
– Tentativo di ottimizzazione della risposta anticorpale
• Mutazione ipersomatica
• “class switch”
• Conversione genica
La risposta secondaria
AID e mutazione ipersomatica
• Mutazione ipersomatica: iniziata dall’enzima AID
)Activation-induced cytidine deaminase),
espressa solo nelle cellule B (e non T) attivate
• Malattia da deficit di enzima: I malati producono
solo IgM e non aumentano tramite mutazione
ipersomatica l’affinità delle Ig per l’antigene
• AID agisce da citidina-deaminasi sul DNA
– Quando agisce nei segmenti variabili dei geni per le Ig,
inizia la fase di mutazione ipersomatica
– Quando agisce nei segmenti di “switch” inizia lo
switch di classe di Ig
L’attività di AID
• AID induce lesioni del DNA il cui
riparo porta alla risposta secondaria
• Il meccanismo di riparo utilizzato
determina il tipo di evento
ricombinativo
• Mutazioni -> mutazione
ipersomatica
• ssDNA break -> conversione
genica
• dsDNA break -> class switch
Conseguenze delle mutazioni
ipersomatiche
• Le cellule B attivate sono indotte da cellule T
– Mutazione ipersomatica avviene nei centri
germinativi
• Centri germinativi: competizione di cloni di
cellule B
– Le mutazioni ipersomatiche distruttive portano
all’eliminazione della cellula B corrispondente
– Le mutazioni ipersomatiche che migliorano
l’affinità dell’Ab per l’antigene sono selezionate
• Maturazione dell’anticorpo
Maturazione anticorpale e clustering
delle mutazioni nelle CDR
Che cosa succede nelle cellule T?
• La diversità nei TCR è generata durante il
riarrangiamento genico
• Non avviene mutazione ipersomatica
– Probabilmente la mutazione ipersomatica è stata
selezionata nelle cellule B per permettere la
produzione di anticorpi secreti ad altissima
affinità, mentre le cellule T non secernendo
anticorpi hanno sufficiente diversità/affinità senza
ricorrere alla mutazione ipersomatica
“Class switch”
• Le cellule B esprimono IgM ed IgD
• Dopo che sono state attivate dall’antigene,
avviene lo switch di classe…
Lo switch di classe
• Switch da catena “mu” a catena
“epsilon”
• Rilevanza di trascrizione per generare
aree di ssDNA che fungono da substrati
per le varie attività enzimatiche
• Formazione di ssDNA break e poi di
dsDNA break
• Attivazione della machinery di riparo del
DNA, che congiungono le due regioni
switch ed eliminano il DNA che si
frappone ad esse