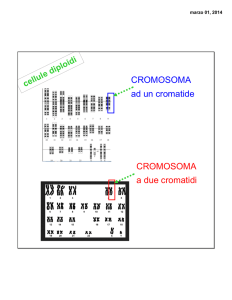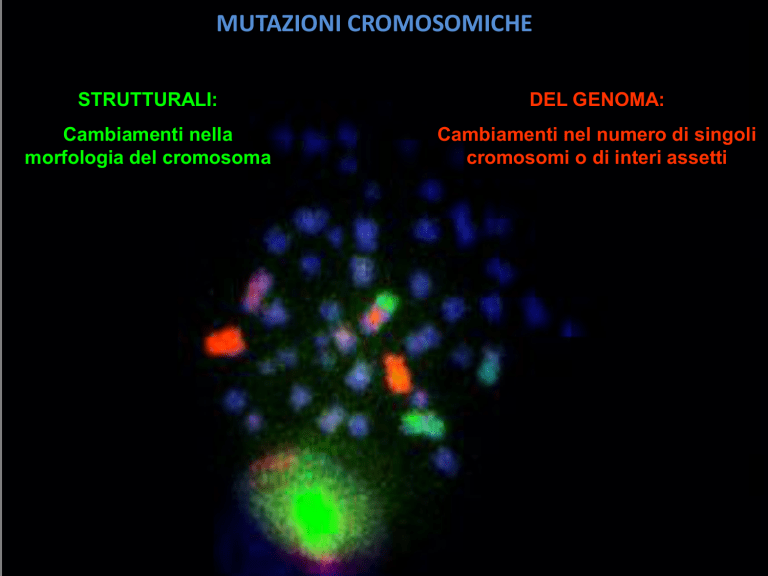
MUTAZIONI CROMOSOMICHE
STRUTTURALI:
DEL GENOMA:
Cambiamenti nella
morfologia del cromosoma
Cambiamenti nel numero di singoli
cromosomi o di interi assetti
1. Rottura;
GLI EVENTI: 2. Interazione tra due estremità di taglio;
3. Risaldatura: a) legittima = ripristino del
cromosoma originale
b) illegittima = cromosoma
aberrante
a) legittima:
prossimale al
centromero
distale al
centromero
b) illegittima:
oppure
Traslocazione reciproca
Cromosoma dicentrico +
frammento
PERCHE’ I CAPI DI ROTTURA RISALDANO? Le estremità di taglio sono instabili perché
non sono “protette” da sequenze telomeriche. Per questo vengono “processate” da
attività enzimatiche dei sistemi di riparazione, oppure degradate da esonucleasi.
COME? Il passo iniziale consiste sempre nell’interazione tra sequenze omologhe,
anche brevi, seguendo poi meccanismi di tipo ricombinativo.
Come si formano: MECCANISMI DI INSORGENZA
1 ROTTURA
2 ROTTURE sullo stesso
cromosoma (con risaldatura)
Le rotture al doppio filamento del DNA (interruzioni nell’impalcatura
zucchero-fosfato) tendono a risaldare, senza lasciare estremità di taglio
libere.
Alla base della
formazione di mutazioni
strutturali vi è la
produzione “accidentale”
di rotture alla doppia
elica: queste generano
delle “estremità di taglio”
libere ed instabili perché
prive del capping
telomerico presente
nelle estremità naturali.
Una volta prodotte,
queste sono ‘disponibili’
per interazione reciproca
che daranno origine a
nuove riorganizzazioni
attraverso risaldatura ad
opera di DNA ligasi, in
seguito a processi di
riparazione specifici.
1a)
2)
(in presenza di ripetizioni in tandem: 1’-1’’- 1’’’- 1’’’’ )
1b)
3)
Possono anche
essere originare
attraverso crossing
over in cellule
somatiche tra
sequenze di DNA
ripetitivo presenti nel
genoma:
conseguenza di
questi processi sono
tipicamente le
DELEZIONI e le
DUPLICAZIONI.
Produzione di rotture al doppio filamento di DNA (Double Strand Break, DSB):
1) ROTTURA singola
G1
G2
centromero
(oppure la DSB
risalda “a forcina” e
poi replica);
Frammento
acentrico
Le DSB presenti in G1, durante la fase Anafase:
S possono “replicare” insieme al
cromosoma
I FRAMMENTI ACENTRICI
vengono sempre persi,
dando luogo a DELEZIONI
(trazione in
anafase) rottura secondaria!
Se non c’è risaldatura:
delezione, per perdita del
frammento acentrico.
In caso di risaldatura
tra estremi di rottura
prossimali si forma una
aberrazione instabile
(isocromatidio) che
ostacola la segregazione
in anafase.
2) Due ROTTURE sullo stesso cromosoma:
a) sullo stesso braccio (paracentriche):
STABILI = inversioni
oppure
INSTABILI = delezioni
A
A
B
B
B
C
C
D
E
F
F
Inversione
paracentrica
D
F
Cromosoma deleto
(delezione interstiziale) +
frammento circolare
possiede il centromero
b) su bracci diversi (pericentriche):
A
A
B
A
C
B
B
C
D
D
E
E
F
F
Inversione
pericentrica
E
+
C
E
D
possiede il
centromero
A
C
+
D
E
F
Cromosoma ad anello +
frammento
2) Due ROTTURE su cromosomi diversi:
oppure
STABILI
INSTABILI
A
M
A
M
A
B
N
B
N
B
O
C
E
D
+
+
O
C
C
P
D
D
E
P
+
P
+
O
E
M
t:3-9
N
9
t:9-3
3
traslocazione reciproca
Cromosoma dicentrico + frammento
2’) a livello del Centromero:
Traslocazione Robertsoniana
(o fusione centrica)
1) Sindrome Down: Rb 14-21
L’assetto 2n= 46, X Y: -14 + t(14q21q) in cui è presente la duplicazione dell’intero cromosoma
21, risulta della fecondazione tra un gamete normale X (o Y) ed un gamete mutante: n= 23,
Y (o X) -14 + t(14q21q).
Al momento di insorgenza della traslocazione Rb (nell’es., il cromosoma Rb 14-21) l’assetto è
bilanciato ed il fenotipo è normale. L’individuo eterozigote per la traslocazione, con cariotipo
2n=45, Y: -14 + t(14q21q) è sano.
Ma in meiosi, per effetto dell’assortimento indipendente, la probabilità di gameti
sbilanciati con la duplicazione del 21 è pari al 25% . Questo individuo, eterozigote per
la traslocazione, è ‘portatore sano’ della sindrome di Down.
Mutazioni strutturali STABILI o INSTABILI: rapporto 1:1 tra numero di filamenti di cromatina
lineari e n. di centromeri presenti.
la stabilità di un cromosoma è data dalla presenza di uno ed unico centromero (funzionale) per
unità cromosomica (filamento di cromatina “aperto”, cioè con estremità telomeriche “libere”).
Le mutazioni STABILI (delezioni, le inversioni e le traslocazioni) mantengono, nella nuova
configurazione, le capacità di segregazione corretta e di equa ripartizione nelle cellule figlie dei
cromosomi coinvolti: sono quindi ereditabili nelle generazioni successive.
La presenza aberrazioni strutturali instabili si rileva facilmente,
mentre l’identificazione di quelle
stabili (traslocazioni e inversioni)
è più difficile
Aberrazioni cromosomiche in linfociti umani (c-metafasi). 1
cromosoma ad anello; 1 dicentrico; 1 frammento circolare;
2 dicentrici.
Esempi di aberrazioni cromosomiche
stabili: traslocazioni reciproche (FISH
chromosome painting).
Aberrazioni cromosomiche instabili: meccanica dell’instabilità
+
FIGURA 11.1 Aberrazioni instabili e conseguenze meccaniche (eliminazione) sia in cellule somatiche che germinali. La
comparsa di aberrazioni semplici (rotture), o complesse, come i dicentrici, ring, isocromatidi, è associata alla produzione di
frammenti, responsabili della produzione di micronuclei (MN) all’interfase immediatamente successiva. Le aberrazioni
instabili inoltre danno luogo a ulteriori riordinamenti alla mitosi successiva, in seguito a nuove frammentazioni dovute alla
trazione dei ponti intercentomerici di cromatina, nelle anafasi seguenti.
Quali sono le conseguenze delle mutazioni strutturali per le cellule o per
l’organismo?
1) Le rotture DSB possono causare interruzione di sequenze codificanti e portare ad
inattivazione del gene.
2)
Se comportano alterazione nel numero di geni/cromosomi causano
SBILANCIAMENTI nel dosaggio genico:
Incompatibilità con la sopravvivenza di sbilanciamenti (ipo- e ipervarianti) provocati da perdita di porzioni del genoma (frammenti
cromosomici=delezioni ) o dall’acquisto di ulteriori copie (duplicazioni).
3) Se comportano cambiamento di posizione di parti del cromosoma:
•
Effetto di posizione (traslocazioni) = inattivazione per eterocromatizzazione;
•
Formazione di nuovi geni ricombinanti (es. cromosoma Philadelphia;
emoglobina Lepore);
•
Aumento del livello di espressione (se posti davanti a “enhancer”);
•
conseguenze meccaniche dovute ai riordinamenti cromosomici instabili
(incompatibilità con la divisione mitotica/meiotica; sterilità).
*Instabilità meiotica delle aberrazioni
cromosomiche stabili
A
C
B
D
A
B
C
D
pericentriche
Inversioni:
paracentriche
A
A
B
C
D
E
F
B
C
E
D
F
Traslocazioni reciproche
Traslocazioni:
A
M
A
M
B
N
B
N
O
C
P
D
+
C
D
E
P
+
O
E
Traslocazioni Robertsoniane
(fusioni centriche)
21
Rb 21-14
14
Aberrazioni cromosomiche stabili:
in meiosi = instabilità secondaria
Inversioni (in eterozigosi)
paracentriche
A
B
C
E
D
F
pericentriche
A
C
B
D
E
F
Nella linea germinale la presenza di una inversione (in eterozigosi) si riconosce
inequivocabilmente nei meiotici primari (profase meiotica I) per la formazione di un’ANSA di
appaiamento, che comprende la porzione corrispondente nei due omologhi (parentale e
ricombinante), grazie all’appaiamento nella formazione dei bivalenti.
Meiosi I di topo (diplotene). Figure di appaiamenti meiotici con anse da
inversione.
*Instabilità meiotica delle aberrazioni
cromosomiche stabili
1) inversione pericentrica:
Inversione
pericentrica
A
B
C
D
A
C
B
D
Omologo con la regione
“B-C” invertita
Il centromero è
all’interno dell’ansa.
Solo se si verifica 1 (o un n. dispari) di
crossing over all’interno dell’inversione:
Nei 2 cromatidi ricombinanti:
le regioni esterne all’inversione
(A e D) saranno duplicate e
delete in modo complementare.
Inversione
paracentrica
A
A
B
C
D
E
F
B
C
E
D
F
Omologo con la regione
“B-D” invertita
Se si verifica 1
crossing over
all’interno
dell’inversione:
In ciascuno dei 2 cromatidi ricombinanti
saranno presenti duplicazioni e delezioni
“complementari” delle regioni esterne
all’inversione (A ed E):
in A è compreso anche il centromero,
che quindi in uno dei cromatidi è
duplicato (dicentrico) e nell’altro è
assente (frammento, =eliminato). In
anafase il dicentrico si spezza ed i
cromatidi segreganti porteranno un
ulteriore delezione.
Il centromero è
esterno all’ansa!
traslocazione reciproca
A
M
A
M
B
N
B
N
O
C
P
D
+
C
D
E
N2
N1
+
O
E
T2
P
T1
Solo la segregazione
alternata dà prodotti
bilanciati e vitali: con
questa modalità di
segregazione si trovano
nella stessa cellula o
entrambi i cromosomi
traslocati (T1 e T2) o
entrambi i parentali
(N1 e N2).
Appaiamento
omologhi:
FUSIONI CENTRICHE (traslocazioni Rb).
Inizialmente: in eterozigosi nel portatore
(genitore) o nelle cellule germinali;
Segregazione nel meiocita I
e
21
Rb 21-14
14
oppure
formazione dei gameti:
Gameti bilanciati
Gameti sbilanciati
Fecondazione e
trasmissione alla
progenie:
2n normale
eterozigote Rb
2n-1
eterozigote Rb
sbilanciato:
sbilanciato
bilanciato (45,
46,-14t(14q21q)
t14q21q)
IN MEIOSI
(gameti prodotti)
+
+
+
Riassumendo: gli effetti dei riordinamenti cromosomici (in
eterozigosi) dal momento della loro comparsa, possono
comportare:
in cellule della linea
SOMATICA:
Non trasmissibili
alla progenie
in cellule della linea
GERMINALE:
Trasmissibili alla
progenie
•letalità cellulare (aberrazioni
instabili; aberrazioni
sbilanciate);
•Formazione di un clone;
•Nessun effetto.
Possono essere considerate solo aberrazioni stabili, poiché
quelle instabili vengono eliminate durante le precedenti
divisioni mitotiche (linea goniale). In meiosi tuttavia
anche queste sono sottoposte a selezione ed
eliminazione, a causa di:
1) instabilità secondaria o per segregazione adiacente, in
meiosi I.
2) Se non ci sono effetti di “selezione meiotica”
(segregazione anomala) = 50% gameti mutanti
Il cromosoma Phladelphia (Ph) è considerato un ‘marcatore’ della leucemia mieloide cronica
(CML): risulta dalla traslocazione reciproca t-9:22, in cui le rotture interrompono i due loci
ABL e BCR, rispettivamente. Il cromosoma Ph è rilevabile citologicamente (cromosoma
‘extra’ del gruppo G). La giustapposizione dei loci ABL e BCR interferisce con i geni
soppressori cancro (oncogeni ), e si traduce in una forma specifica di tumore, leucemia
cronica Myologenous (CML).
Altri tipi di tumori
solidi hanno alla base
traslocazioni che
possono rendere
attivi alcuni geni.
Distrofia muscolare di
Duchenne (DMD): in questo caso
la patologia è data dalla
traslocazione reciproca t 21X (in eterozigosi). La rottura ha
reso inattivo l’allele DMD+.
L’altro allele è reso non
funzionale dall’inattivazione
dell’altro X.
Come riconoscere citogeneticamente le inversioni?
Nelle cellule somatiche le inversioni si possono distinguere solo utilizzando
tecniche di bandeggio di preparati cromosomici (o attraverso tecniche di
ibridazione in situ);
Solo alcune inversioni pericentriche si possono distinguere più facilmente, per
l’alterazione del rapporto della lunghezza tra i bracci, quando i 2 punti di
rottura hanno distanze molto diverse dal centromero.
TRASLOCAZIONI
Citogeneticamente
riconoscibili solo per:
•Cambiamento lunghezza totale e/ o posizione del centromero;
•Cambiamento n° e posizione delle bande (bandeggio).
Oppure attraverso ibridazione in situ (FISH)
Effetti genetici:
-Sterilità parziale (50% dei gameti
sbilanciati)
- Fenotipo di associazione nuovo: se 2
marcatori di 2 cromosomi non omologhi si
trovano vicino ai punti di rottura, nel
cromosoma traslocato essi si troveranno in
associazione (linkage).
Ad eccezione delle traslocazioni Robertsoniane:
Traslocazioni RB in spermatociti II di topo.
EFFETTO DI POSIZIONE
I geni contenuti in regioni eterocromatiche sono inattivi.
Se un gene attivo viene traslocato in una regione
eterocromatica, questo viene inattivato.
Esempio: Variegatura dell’occhio (drosofila). Se in un
eterozigote w+/w (fenotipo occhi rossi) il gene w+ viene
spostato dalla regione eucromatica dell’X ad una
regione eterocromatica del 4, può venire inattivato.
TUMORI NELL’UOMO. Il linfoma di Burkitt è causato dallo
spostamento di un proto-oncogéne vicino ad una regione che
stimola la produzione di anticorpi. Questo spostamento rende
attivo l’oncogéne ed innesca lo sviluppo del cancro.
Duplicazioni e delezioni
IN MEIOSI: CROSSING OVER INEGUALE
Genotipo parentale
(appaiamento
sfalsato +
crossing over tra
i due loop)
Dup
Del
Genotipi ricombinanti: 1 omologo duplicato (Dup) + 1
omologo deleto (Del).
Emoglobina
umana
Le duplicazioni
hanno un ruolo
evolutivo
importante
perchè fonte di
materiale
ridondande in cui
le mutazioni
possono creare
nuove funzioni
geniche
DELEZIONI
-Perdita di un frammento interstiziale
(2 rotture e risaldatura)
- Perdita di un frammento terminale
(1 sola rottura)
In funzione della dimensione del frammento deleto ci sono effetti diversi:
1.
intragenica: inattivazione del gene
2.
multigenica (eliminazione di intere bande o +): inattivazione di più
geni
EFFETTI GENETICI
•No
RETROMUTAZIONI
1) In ETEROZIGOSI:
DEFICIT O LETALITA’
Le delezioni in eterozigosi
sono scarsamente tollerate
da cellule/tessuti/organismi)
2) In OMOZIGOSI:
•Permette l’espressione di geni recessivi (alleli difettivi o
letali) = PSEUDODOMINANZA
• Sbilanciamento nel dosaggio proteico-enzimatico
La delezione di un gene dominante, in eterozigosi, permette
l’espressione dell’altro allele e nel nuovo fenotipo compare la
manifestazione dell’allele recessivo. Questo può sembrare l’effetto
di una mutazione dominante.
• LETALITA’ per mancanza totale del gene / dei geni contenuti
nella regione deleta, su entrambi i cromosomi omologhi.
Le delezioni in omozigosi sono sempre letali
(come fossero dei caratteri letali recessivi).
DELEZIONI: COME SI IDENTIFICANO
1) CITOGENETICAMENTE
(analisi strutturale al microscopio):
Ibridazione
in situ: rivela
la posizione
di regioni
attraverso la
marcatura
specifica con
sonde di
DNA
Se la delezione è sufficientemente grande (una
banda) si formano anse di appaiamento in cellule
somatiche (es. cromosomi politenici di drosofila) o
nei bivalenti (appaiamento tra omologhi in meiosi).
2) GENETICAMENTE
1)
FR ridotta nelle regioni adiacenti alla delezione
2)
si manifesta la pseudodominanza
3)
Si manifesta l’effetto letale dei geni recessivi “scoperti”
4)
Non c’è retromutazione
MAPPATURA PER DELEZIONE
Le delezioni possono essere utilizzate per localizzare nuovi alleli mutanti
sfruttando l’effetto della PSEUDODOMINANZA.
Nuovi alleli mutanti, mostrano il
loro effetto recessivo solo se
localizzati in corrispondenza della
regione deleta,
Sindrome Crie du chat:
perdita della regione
terminale del braccio
corto del cromosoma 5
(origine spontanea)
DUPLICAZIONI
Possono trovarsi
in posizione
adiacente:
oppure localizzate in altre posizioni
(sullo stesso o su altro cromosoma)
Se in ETEROZIGOSI si
distinguono perché formano
anse da appaiamento
In meiosi: appaiamento
tra omologhi
In OMOZIGOSI:
indipendentemente dalla loro localizzazione, in seguito ad appaimento sfalsato
producono ulteriori duplicazioni e delezioni (crossing over asimmetrico).
Occhio bar (drosofila):
duplicazione regione
16A su cromosoma X