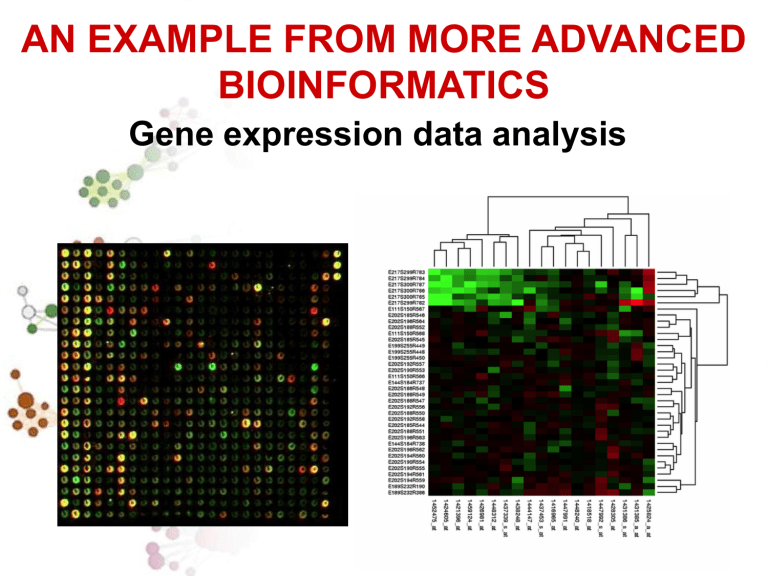
AN EXAMPLE FROM MORE ADVANCED
BIOINFORMATICS
Gene expression data analysis
VI LEZIONE
• Introduzione all'analisi di dati
d'espressione genica.
• Metodi per lo studio dell’espressione
genica su larga scala.
• Profili e matrici d'espressione.
• Ricerca di geni co-espressi e di geni
differenzialmente espressi.
Metodi per lo studio dell’espressione genica
su larga scala:
1. Basati su ibridazione: Microarray/Chip
2. Basati su conteggio di sequenze: EST
sequencing, SAGE, e deep sequencing di
librerie di cDNA
Deep seq. EST
SAGE
MICROARRAY
CHIP
Computational analysis of data by statistical methods
ESPRESSIONE DEL GENOMA UMANO
NELLE CELLULE DIFFERENZIATE
• Tutte le cellule di un organismo hanno lo stesso
corredo genomico
• L’espressione genica tessuto specifica determina il
fenotipo morfo-funzionale dei tipi cellulari e tissutali
• In ogni cellula differenziata ed in ogni particolare
momento dello sviluppo e’ attivo solo un sottoinsieme
dei geni
REGOLAZIONE DELL’ESPRESSIONE GENICA
• Puo’ agire su ciascuno dei livelli che caratterizzano
il passare dell’informazione genica dal DNA alle
proteine
• Negli Eucarioti superiori la regolazione
dell’espressione genica si svolge principalmente
come controllo della trascrizione
• Principali tipi di regolazione:
Controllo epigenetico
Controllo trascrizionale
Controllo post-trascrizionale
“One-gene approach”
Il gene di interesse e’ espresso in un tessuto o in un dato momento dello
sviluppo ? Quanto e’ attivo dal punto di vista trascrizionale ?
Real Time PCR
PCR semiquantitativa
Ibridazione DNA genico o cDNA con RNA
totale o poly(A)+RNA (Northern blot)
Ibridazione in situ
“Large-scale approach”
Quali geni sono espressi in un tessuto ed in un dato momento dello sviluppo ?
Quanto ciascuno di essi e’ attivo dal punto di vista trascrizionale ?
Profilo d’espressione del genoma
(TRASCRITTOMA)
METODI PER LO STUDIO SU LARGA SCALA
DELL’ESPRESSIONE GENICA BASATI SUL
SEQUENZIAMENTO
Sequenziamento sistematico di ESTs da librerie di
cDNA
Sequenziamento sistematico con metodi di terza
generazione di librerie di cDNA
SAGE (Serial Analysis of Gene Expression)
Deep seq.
SEQUENCING
mRNA of different genes
cDNA LIBRARY
EST
UniGene Human Release Statistics
Total sequences in clusters:
3115711
Total number of clusters sets:
95928
22094
sets contain at least one known gene
94710
20876
sets contain at least one EST
sets contain both genes and ESTs
EST
ESTIMATE OF THE LEVEL OF EXPRESSION
OF A GIVEN GENE
Sample of 12919 ESTs corresponding to 4460
genes/trascripts
eg. Rhodopsin:
65 retina ESTs 65 / 12919 = 0.503%
SAGE Serial Analysis of Gene Expression
SAGE
SAGE è un metodo sperimentale ideato per utilizzare i vantaggi
del sequenziamento su larga scala per avere informazioni
quantitative di espressione genica (Velculescu et al. 1995,
Zhang et al, 1997)
Con questa tecnica e’ possibile stimare il livello d’espressione
di ciascun gene, attraverso la misura del numero di volte in cui
la TAG che lo rappresenta compare in un campione
abbastanza grande di TAGs sequenziate a partire dal
messaggero del tessuto in analisi
Tag to Gene mapping Gene to Tag mapping
Consiste nel sequenziamento da messaggeri cellulari di brevi
oligonucleotidi, che fungono da etichette di sequenza (TAG)
SAGE
Isolamento delle “tag”
le "tag" possono essere unite
insieme in serie, a costituire lunghe
molecole di DNA, che vengono
clonate e sequenziate.
Ligazione
Sequenziamento
Livello di
espressione
Livello di
espressione
Quantificazione di ciascuna
“tag” e determinazione del
pattern di espressione
GENE
GENE
Normale
Normale
una sequenza di 9 paia di basi
permette di identificare 49 (262144)
diversi trascritti (una "tag" viene
ottenuta da una posizione specifica
di ogni trascritto).
GENE
GENE
Alterato
Alterato
il numero di volte in cui una singola
"tag" viene osservata permette di
quantificare l'abbondanza del
messaggero identificato nella
popolazione dei messaggeri e,
indirettamente, il livello di
espressione del gene
corrispondente.
MICROARRAY
DUE CANALI
Esperimenti di Microarray
Permettono l’analisi
dell’espressione di
migliaia di geni
simultaneamente
MICROARRAY
DUE CANALI
GeneChip
Affymetrix
SINGOLO
CANALE
Ibridizzazione della sonda marcata
Scansione del GeneChip con scanner laser
Analisi immagine
Normalizzazione
Espressione
differenziale
Clustering
Interpretazione
biologica
MICROARRAY
Analisi dell’immagine
GeneChip
Affymetrix
SINGOLO
CANALE
• Identificazione della
posizione degli spot
• Costruzione di un’area
locale intorno ad ogni
spot
• Calcolo dell’intensità di
ogni singolo spot
• Calcolo del background
locale
MICROARRAY
Elaborazione dei dati
GeneChip
Affymetrix
SINGOLO
CANALE
Deep seq. EST
SAGE
MICROARRAY
CHIP
Matrice dei risultati:
righe = geni, colonne = condizioni sperimentali
Cond. 1
Cond. 2
…
Cond. m
Gene 1
x11
x12
…
x1m
Gene 2
x21
x22
…
x2m
…
…
…
…
xn1
xn2
…
xnm
…
Gene n
• Quali geni sono differenzialmente espressi ?
• Quali e quanti geni sono co-espressi?
Obiettivi dell’analisi saranno…
Identificazione geni differenzialmente
espressi
Identificazione pattern di espressione comuni
Identificazione di geni co-espressi con geni di
funzione nota
Campione 2
Campione 3
Campione 4
Campione 5
Campione 6
Campione 1
Campione 2
Campione 3
Campione 4
Campione 5
Campione 6
Campione 7
Campione 8
10
12
15
50
34
10
12
13
22
10
10
10
10
10
100
150
100
90
70
110
400
345
300
400
300
300
300
409
1000 1110 1200
950
850
900 1100 1110 1200
800 1050
300
400
300
300
300
5
1
1
2
2
3
1
3
1
1
2
2
3
1
50
56
62
68
74
47
86
50
1100 1110 1300
950
850
2
3
3
2
1
2
1
1
1400
1200
1000
Gene 1
Gene 2
800
Gene 3
Gene 4
Gene 5
600
Gene 6
Gene n
400
COLON NORMALE
COLON CARCINOMA
Campione 8
Campione 7
Campione 6
Campione 5
Campione 4
Campione 3
0
Campione 2
200
Campione 1
1
Campione 6
5
Campione 5
1
Campione 4
2
790 1000 1000
Campione 3
1
800 1050
Campione 2
1
900 1100 1110 1000
Schema sperimentale
“semplice”:
Dati d’espressione in colon
normale e carcinoma
Domanda biologica:
Quali geni sono
differenzialmente espressi nel
confronto ?
790 1000 1000
345
Campione 1
Gene 1
Gene 2
Gene 3
Gene 4
Gene 5
Gene 6
Gene n
COLON CARCINOMA
Campione 1
COLON NORMALE
GENI DIFFERENZIALMENTE ESPRESSI
Fold Change: un primo criterio puo’ essere quello di
identificare i geni la cui espressione nei due gruppi di
campioni considerati varia di una certa proporzione
(raddoppia, dimezza, …) fold change = 2
Molti falsi positivi
I geni poco espressi risultano differenzialmente espressi
anche3 con variazioni non significative
Selezione basata sui p-values associati a Test T: si applica
un test statistico per il confronto delle medie di due
campioni a ciascun gene; ogni gene risulta associato ad
una probabilità (di essere differenzialmente espresso)
Si esegue uno stesso test statistico molte volte, serve una
correzione
Uso di metodi basati su permutazioni (SAM) e FDR
MUSCOLO
Campione 1
Campione 2
Campione 3
Campione 4
Campione 1
Campione 2
Campione 3
Campione 4
Campione 1
Campione 2
Campione 3
10
12
15
50
34
10
12
13
22
10
10
10
10
10
100
150
100
90
70
110
430
345
300
400
300
300
300
409
1000
1110
1200
950
850
900
1100
1110
1200
800
1050
790
1000
1000
345
300
400
300
300
300
5
1
1
2
2
3
1
3
3
4
2
5
6
7
30
56
62
68
74
47
86
50
1100
1110
1300
950
850
900
1100
1110
1000
800
1050
790
1000
1000
1
1
2
2
3
1
5
1
3
2
1
2
1
1
13
14
12
15
16
17
40
66
72
78
84
57
96
60
120
170
150
110
90
130
500
365
320
560
320
320
320
429
340 1400
440
45
60
200
250
333
1000
2
1
2
1
1
1200
Gene 1
Gene 2
Gene 3
1000
Gene 4
Gene 5
800
Gene 6
Gene 7
600
Gene 8
Gene 9
400
Gene 10
Gene 11
Gene n
200
0
BAMBINO
GIOVANE
ADULTO
MUSCOLO
ANZIANO
Campione 3
29
449
1000
Campione 2
55
340
790
Campione 1
60
200
1050
Campione 4
50
340
800
Campione 3
1000
Campione 2
1110
Campione 1
385
1100
Campione 4
470
900
Campione 3
150
850
Campione 2
110
950
Campione 1
130
Campione 3
180
1300
Campione 2
200
1110
Campione 1
150
1100
Schema sperimentale
piu’ complesso:
Dati d’espressione in
piu’ condizioni
Domanda biologica:
Posso identificare gruppi
di geni espressi in modo
simile ?
ANZIANO
Campione 3
ADULTO
Campione 2
Gene 1
Gene 2
Gene 3
Gene 4
Gene 5
Gene 6
Gene 7
Gene 8
Gene 9
Gene 10
Gene 11
Gene n
GIOVANE
Campione 1
BAMBINO
CLUSTER ANALISI
• Il CLUSTERING o analisi cluster o analisi di
raggruppamento è un insieme di tecniche di analisi
multivariata dei dati volte al raggruppamento di elementi
omogenei.
• Un insieme di oggetti grande e disomogeo viene classificato in
una serie limitata di gruppi omogeneei, ovvero “vicini” in
accordo con una specifica misura di distanza.
CLUSTER ANALISI
DUE STEPS:
Misura di similarita’
•
Diverse misure
•
Standardizzazione dei dati
Linking method
•
criterio per stabilire i gruppi
•
Metodi gerarchici e non gerarchici
CLUSTER ANALISI
I geni sono punti nello spazio:
punti vicini nello spazio sono raggruppati insieme
• Si parte dalla matrice dei dati X di dimensione nxp e la si
trasforma in una matrice nxn di dissimilarità o di distanze
tra le n coppie di osservazioni (vettori di p elementi).
• Si sceglie poi un algoritmo che definisca le regole su come
raggruppare le unità in sottogruppi sulla base delle loro
similarità.
• Lo scopo e’ di identificare un cero numero di gruppi tali
che gli elementi appartenenti ad un gruppo siano – in
qualche senso – piu’ simili tra loro che non agli elementi
appartenenti ad altri gruppi.
CLUSTER ANALISI
Distanza euclidea
Correlazione di
Pearson
1- Data Matrix
PROBESET/GEN
E
CD34
Eritroblas
ti
Mieloblas
ti
Monoblas
ti
MKC
Monociti
Neutrofili
Eosinofili
GC00U921857_at
-1.0
1.2
1.1
-1.1
-0.2
-1.0
1.1
-0.2
GC00U922066_at
-0.5
-1.0
-0.9
-0.2
-0.9
1.1
1.2
1.1
GC00U990452_at
-1.1
1.2
1.1
-0.2
1.0
-1.0
0.0
-1.1
GC00U990575_at
0.1
-1.0
-0.5
1.0
-1.0
1.2
1.2
-1.0
GC00U990668_at
1.1
1.1
1.0
0.3
-0.3
-1.0
-1.2
-1.0
GC00U990680_at
-0.8
-0.9
0.2
1.1
1.1
1.2
-0.9
-0.9
GC00U990706_at
-0.1
-1.2
-1.0
0.4
-1.1
1.2
0.9
1.0
GC01M033561_at
0.1
-1.0
-1.0
1.0
-1.2
1.2
1.2
-0.3
GC01M035219_at
1.1
1.1
0.5
-1.0
-0.5
-1.0
-1.1
1.0
GC01M035470_at
-1.0
-0.9
-1.2
1.1
-0.3
0.1
1.0
1.2
GC01M035671_at
1.2
-1.0
0.2
-1.2
0.0
-1.1
1.0
0.9
GC01M035737_at
1.2
1.2
1.2
-0.8
-0.4
-0.4
-0.8
-1.1
GC01M035952_at
1.3
-0.2
1.1
-0.9
1.0
-0.1
-0.9
-1.2
GC01M035958_at
-0.1
-1.2
-1.0
-0.8
-0.4
1.1
1.1
1.2
GC01M036333_at
-0.9
1.2
1.0
0.0
1.2
-0.9
-1.2
-0.4
Eosinofili
Neutrofili
Monociti
Monoblasti
MKC
Mieloblasti
Eritroblasti
CD34
2- Data representation
1.5
1.0
0.5
0.0
-0.5
-1.0
-1.5
3-Distance and linking method
selection
Pearson QT clustering
1.5
Insieme
disomogeneo
di 40 geni
1.0
0.5
0.0
-0.5
-1.0
Eosinofili
Neutrofili
Monociti
Monoblasti
MKC
Mieloblasti
Eritroblasti
CD34
-1.5
6 cluster,
gruppi
omogenei
4 - Result