
VIRUS A DNA
1)
.a
.b
2)
DNA lineare
DNAds = Adenovirus e Herpesvirus. Poxvirus
DNAss = Parvovirus
DNAds circolare = Hepadnavirus, Poliomavirus e
Papillomavirus
Replicazione nucleare
Replicazione citoplasmatica
POXVIRUS
Replicazione nucleare e citoplasmatica
HEPADNAVIRUS
1
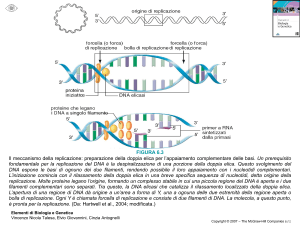
REPLICAZIONE del DNA VIRALE
SEMICONSERVATIVA
Parvovirus - Papillomavirus e Poliomavirus: DNA pol cellulare
Adenovirus - Herpesvirus: DNA pol virale
> velocità
> errori
Target per agenti antivirali
(es. acyclovir, AZT)
2
REPLICAZIONE DEI VIRUS a DNA
PROBLEMI
Richiesta di un primer di inizio della replicazione del DNA
lo stesso problema della cellula ospite: le DNA polimerasi non
sono in grado di replicare il DNA a partire da uno stampo a
ssDNA. Possono solo iniziare la replicazione a partire da
regioni ds (5’ 3’)
•come replicare le estremità senza perdere
informazione?
•soluzione: specifiche caratteristiche strutturali dei
genomi
3
Le sequenze Ori
siti di legame per proteine (Ori recognition proteins): Palindromi
sequenze dentro o vicino a regioni di controllo trascrizionale
siti di legame per fattori trascrizionali e proteine con funzione di
enhancer , virali e/o cellulari (aumento dell’efficienza di
replicazione)
4
sequenze ricche di AT: facilitano lo srotolamentote
From Flint et al. Principles of Virology (2000), ASM Press
Replicazione del genoma dei virus a DNA :
le proteine di riconoscimento della regione ORI
Tutti i virus a DNA devono codificare almeno una proteina per
iniziare la replicazione del genoma
I virus più grandi codificano anche la loro DNA polimerasi e
altre proteine essenziali per la replicazione del genoma
Caratteristiche comuni:
legame di specifiche proteine alla regione ORI del genoma virale.
il legame con le proteine tende a distorcere la regione ORI
Molte delle proteine reclutate sulla regione ORI hanno attività di elicasi
ATP-dipendente per lo srotolamento del DNA virale
eventuale reclutamento di proteine cellulari
per la replicazione del DNA virale
5
From Flint et al. Principles of Virology (2000), ASM Press
TRASCRIZIONE DEI VIRUS A DNA
Presenza di proteine regolatrici virali e/o cellulari che
interagiscono con sequenze “promoter”al 5’ dei geni virali
Utilizzano RNA pol II cellulare
Trascrizione di geni su filamenti diversi di DNA ed
in direzione opposta (es. SV40 = early e late su filamenti opposti)
5’ sequenze “cap”
3’ poli A (100-200 residui di adenina)
mRNA con introni
Splicing
mRNA policistronici
lettura in ORF differenti
6
TRASCRIZIONE DEI VIRUS A DNA
nel nucleo della cellula ospite
ecc: Poxvirus:
Utilizzano enzimi presenti nel core del virione
Trascritti senza introni. Assenza di splicing
- Organizzazione temporale (in 2 tempi) con:
mRNA precoci
Proteine non-strutturali
Replicazione del DNA
mRNA tardivi
Proteine strutturali
7
TRASCRIZIONE DEI VIRUS A DNA
- Organizzazione temporale (in 3 tempi) con:
mRNA precoci
immediati precoci (CHX insensibili)
Precoci ritardati (CHX sensibili)
Replicazione del DNA
mRNA tardivi
Proteine strutturali
Es. HERPESVIRUS
8
(45 nm)
Capside icosaedrico nudo
VP1, VP2 e VP3
DNA ds circolare (5.2 kbp)
legato a istoni cellulari (H2A, H2B, H3 e H4)
mRNA E e L trascritti in modo divergente a partire da ORI
Espressione temporale
geni precoci
replicazione DNA
geni tardivi
9
Proteine di SV40
precoci: proteine T e t ( regolatorie e multifunzionali)
- regolano la loro stessa sintesi
- attivano l’espressione dei geni tardivi
transizione della trascrizione
da precoce a tardiva
- essenziale per la replicazione del DNA virale
interazione con pRB e p53 (induzione della fase S nella cellula ospite ed
inibizione dell’apoptosi cellulare
QuickTime™ and a
GIF decompressor
are needed to see this picture.
tardive: proteine strutturali (VP1, VP2, VP3)
10
Replicazione del Genoma di SV40
sito di origine della replicazione (ORI)
la replicazione del DNA è
bidirezionale (replicazione a Theta)
2 esameri di
proteina T
legano un sito
(sito II) della
ORI
ORI = sequenza unica
proteina T (elicasi) insieme a RpA
cellulare (proteina di legame ssDNA)
srotolano il DNA in modo da
permettere la sintesi bidirezionale
del DNA
Terminazione della sintesi del DNA
a 180o da ORI ( congiunzione delle
11
forche di replicazione)
QuickTime™ and a
GIF decompressor
are needed to see this picture.
(55 nm)
Capside icosaedrico
nudo (L1,L2)
DNA ds circolare (8 kbp)
legato a istoni cellulari
>100 tipi di HPV (Human Papilloma Virus)
alto rischio: HPV-16, HPV-18, HPV-31, HPV-45
Specie-specifici
Tropismo per cellule dell’epitelio squamoso
Infezione produttiva solo in cellule epiteliali differenziate
12
PAPILLOMAVIRUS
7 geni E
QuickTime™ and a
GIF decompressor
are needed to see this picture.
2 geni L ( L1 e L2 strutturali)
E6 si lega a p53 : segnale per ubiquitina e degradazione
E7 attiva la proteina Rb
13
I PAPILLOMAVIRUS: il ciclo virale
14
Proteine Virali
• E2: fattore regolatore della trascrizione e della replicazione del DNA
virale.
• E1: fattore importante per la replicazione del DNA virale, si lega
all’origine di replicazione posto nel LCR
• E4: e’ espressa come gene late. Overlaps E2, ma ha un differente ORF.
Viene sintetizzata contemporaneamente alla relicazione del DNA, prima
della sintesi di L1 ed L2
• E5: è altamente idrofobica, associata a membrane cellulari. Sembra induca
una down regulation del sistema MHC
E6: oncogene virale, interagisce con p53
• E7: oncogene virale, interagisce con pRb
•
15
isolati da adenoidi umane - 1953
QuickTime™ and a
Photo - JPEG decompressor
are needed to see this picture.
Capside icosaedrico
nudo 60-90 nm
Circa 100 sierotipi umani
Infezioni respiratorie ed enteriche
Congiuntiviti acute
infezioni latenti nel tessuto linfatico
di tonsille, adenoidi e placche del Peyer
(alcuni tipi oncogeni per roditori neonati)
16
ADENOVIRUS
Genoma:
dsDNA lineare
(30-35 Kbp)
ITR
ITR = Inverted Terminal Repeat
ITR
Terminal protein
P55
5’
desossicitosina
17
ADENOVIRUS
Espressione del genoma
Geni precoci = E1a, E1b, E2a, E2b (DNA pol), E3, E4
Geni tardivi = L1, L2, L3, L4, L5 (proteine strutturali)
18
Adenovirus: replicazione del DNA lineare
la PROTEINA TERMINALE funge da PRIMER
1. la Polimerasi virale forma un legame
fosfodiesterico tra dCMP e pre-TP
il legame di Nf-1 e Oct-1 alla sequenza
core Ori facilita la formazione del
complesso
2. il 3’OH di dCMP serve da innesco
per la sintesi (in continuo) del
filamento di DNA complementare
Ad ssDBP (E2), e Topoisomerasi
3. l’elica di DNA dislocata forma un
“panhandle” via gli “inverted
terminal repeats”
4.associazione Pol-pre-TP e ……
5. sintesi (in continuo) del filamento
complementare
19
From Flint et al. Principles of Virology (2000), ASM Press
Sottofamiglia
Alphaherpesvirinae
Genere
Simplexvirus
Tipo
Herpes simplex
virus tipo 1
Herpes simplex
virus tipo 2
Varicellovirus
Virus Varicella
Zoster
150 nm
Cytomegalovirus
Citomegalovirus
Betaherpesvirinae
Roseolovirus
Capside icosaedrico
con involucro
Herpesvirus
umano 6
Herpesvirus
umano 7
Gammaherpesvirinae
Lymphocryptovirus
Rhadinovirus
EpsteinBarr
virus
Herpesvirus
umano 20
8
o del sarcoma
di Kaposi
QuickTime™ and a
GIF decompressor
are needed to see this picture.
Genoma di HSV-1
DNAds lineare
QuickT ime ™e un d ecomp res sor e TIFF (No n compr ess o) s
(150 Kb)
Sequenze Uniche = U (L e S)
and a
Sequenze RipetuteQuickTime™
=
R
(L
e
S), (T e
GIF decompressor
are needed to see this picture.
I)
21
Genoma del virus Herpes Simplex di Tipo 1
Il genoma lineare quando entra del nucleo della cellula ospite circolarizza per
la presenza di sequenze presenti alle due estremità TR
Il genoma viene trascritto ad opera della RNA polimerasi II cellulare
22
HSV-1:REPLICAZIONE DEL GENOMA
proteine b necessarie per la sintesi del DNA virale
ORI binding protein (UL9)
helicase/primase complex (UL5, UL8, UL52).
DNA polymerase (UL30),
DNA binding proteins (UL42, UL29, ICP8)
23
Modello di replicazione del DNA dei virus erpetici
Cerchio rotante
1. taglio della molecola circolare : 3’-OH utilizzato come primer
2. la sintesi della nuova elica provoca la dislocazione dell’elica
complementare
3 e 4. sintesi in continuo della forma
circolare e sintesi discontinua sull’elica
dislocata con formazione di
concatameri di dsDNA
Il taglio del DNA (sequenza terminale
ripetuta a) congiuntamente al
processo di incapsidamento del DNA
genera i nuovi genomi virali lineari
24
From Flint et al. Principles of Virology (2000), ASM Press
CICLO DI
REPLICAZIONE
cascata di
espressione genica
assemblaggio e
maturazione
25
Capside icosaedrico
non-rivestito
18-28 nm
:
1 molecola lineare DNAss
4.5 - 6 Kb
Parvovirus autonomi
Dependovirus o Virus adeno-associati (AAV)
26
Adeno-associati
AAV
QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per
Ad
Virus satelliti: Per replicare richiedono attiva
replicazione cellulare o la co-infezione con27 un
virus helper (adenovirus o herpesvirus)
DNA lineare ss (4.5 - 6 Kb)
PARVOVIRUS:
115nt
115nt
Qu ic kTi me™ a nd a
GIF d ec omp res so r
are n ee de d to s ee th is pi ctu re .
Sequenze terminali
Sequenze terminali palindromiche
filamento codificante: filamento(-)
28
PARVOVIRUS
Regioni separate per:
proteine non strutturali (gene NS1, gene rep)
proteine strutturali (geni cap)
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Photo - JPEG.
29
REPLICAZIONE DEL GENOMA DEI PARVOVIRUS
rolling hairpin model
REP78
reazione nicking necessaria per la
risoluzione dei terminali.
attività di elicasi essenziale per
srotolamento della regione ITR
+
30




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)





