Patologia molecolare
Per dire che un gene candidato è
responsabile di una patologia, bisogna
trovare in esso mutazioni nei soggetti affetti.
A che cosa ci si può trovare di fronte quando
si sequenzia il DNA dei geni-malattia in
soggetti affetti da una patologia genetica ?
Gene tipo
A
B
D
C
ATG
*
A BCD E
mRNA
F
AAAAAAA
*
ATG
5’UTR
E
CDS
3’ UTR
F
Riarrangiamenti genici
Delezioni
A
B
E
F
Inserzioni
B*
A
C
D
E
F
Inversioni
A
C
B
D
E
F
Solitamente queste alterazioni determinano pesanti conseguenze a
livello della proteina (completa inattivazione o produzione di molecole
tronche). Ne risulta perdita di funzione del gene.
Mutazioni puntiformi
Wild type
M
L
K
F
K
Y
G
V
L
N
ATG TTG AAG TTC AAG TAT GGT GTG CTG AAC
Missense
M
L
K
F
K
F
G
V
R
N
ATG TTG AAG TTC AAG TTT GGT GTG CGG AAC
Non sense
M
L
K
F
K
ATG TTG AAG TTC AAG TAG GGT GTG CTG AAC
Frame-shift
M
L
K
F
K
Y
K
V
C
ATG TTG AAG TTC AAG TAT AAG GTG TGC TGA
La corea di Huntington
• Autosomica dominante, gene
localizzato su una regione
ristretta del cromosoma 4
mediante classica analisi di
linkage.
•Il sequenziamento del DNA dei
pazienti non evidenzia
nessuna delle mutazioni
tradizionali
• L’unica differenza identificata
consisteva nella lunghezza di
un particolare segmento
genico, corrispondente ad uno
degli esoni di un gene della
regione critica.
La corea di Huntington
Soggetto normale
M
L
K
Q
Q
Q
Q
Q
Q
Q
Q
Q
Q
ATG TTG AAG CAG CAG CAG CAG CAG CAG CAG CAG CAG CAG
Q
Q
Q
Q
Q
Q
F
K
Y
G
V
L
N
CAG CAG CAG CAG CAG CAG TTC AAG TAT GGT GTG CTG AAC
Paziente
M
ATG
Q
CAG
Q
CAG
Q
CAG
L
TTG
Q
CAG
Q
CAG
Q
CAG
K
AAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
Q
CAG
F
TTC
Q
CAG
Q
CAG
Q
CAG
K
AAG
Q
CAG
Q
CAG
Q
CAG
Y
TAT
Q
CAG
Q
CAG
Q
CAG
G
GGT
Q
CAG
Q
CAG
Q
CAG
V
GTG
Q
CAG
Q
CAG
Q
CAG
L
CTG
Q
CAG
Q
CAG
Q
CAG
N
AAC
Meccanismo della malattia
• La cornice di lettura non è alterata.
• I livelli di proteina si mantengono costanti
• L’espansione del tratto di triplette CAG all’interno della
sequenza codificante determina la produzione di
proteine caratterizzate da tratti di poliglutamina molto
lunghi.
• I tratti di poliglutamina sono tossici per i neuroni,
perché formano aggregati nucleari che portano, con
meccanismi ancora da chiarire, alla morte delle cellule.
Malattie da espansione di CAG nella
sequenza codificante
• Corea di Huntington
• Malattia di Kennedy
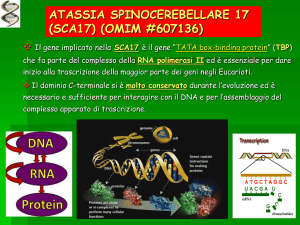
• Atassia spino-cerebellare 1
• Atassia spino-cerebellare 2
• Atassia spino-cerebellare 3
• Atassia spino-cerebellare 6
• Atassia spino-cerebellare 7
• Atrofia dentatorubro-pallidoluysiana
Distrofia miotonica
Autosomica dominante, espansione di triplette CGG all’interno della
regione 3’ non tradotta del gene codificante per una protein-chinasi.
Ancora non si conosce il meccanismo con cui questa mutazione
determina la patologia.
Altre patologie da espansione di
sequenze ripetute instabili
• Sindrome dell’X fragile, gene A (X linked recessiva, triplette
CGG nel 5’ UTR del gene FRAXA, ridotta espressione)
• Sindrome dell’X fragile, gene E (X linked recessiva, triplette
CCG nel promotore del gene FRAXE, ridotta espressione)
• Atassia di Friederiech (autosomica recessiva, triplette GAA
nel I introne ridotta espressione)
• Atassia spino-cerebellare 8 (dominante, triplette CTG in un
RNA non tradotto, mecanismo ignoto)
• Epilessia mioclonica giovanile (autosomica recessiva,
ripetizione CCCCGCCCCGCG nel promotore, ridotta
espressione)
Dominante o recessivo? lo strano caso di p53
• Inizialmente venne isolata come proto-oncogene, perché alcuni
suoi mutanti funzionavano da oncogeni, con un’azione
trasformante dominante
• In seguito si è capito che in realtà è un gene oncosoppressore
perché è il gene più frequentemente inattivato nei tumori (anche
da virus oncògeni).
• Come si spiega
l’effetto dominante di
alcuni mutanti
puntiformi?
Effetto dominante
negativo
Mutazioni caratteristiche delle patologie recessive
In genere le patologie recessive sono causate da mutazioni
che determinano perdita di funzione del gene interessato
(loss of function). Quando sono in eterozigosi, la quantità di
proteina residua, prodotta dall’allele normale è sufficiente a
mantenere un fenotipo normale.
Possono essere causate da tutti i tipi di mutazione conosciuti:
• Riarrangiamenti genici
• Mutazioni puntiformi (possono colpire la trascrizione, lo
splicing, la traduzione o determinare la comparsa di codoni
di stop precoci)
• Espansione di sequenze ripetute
Mutazioni caratteristiche delle patologie dominanti
• La proteina continua a svolegere la sua funzione, ma lo fa
in maniera eccessiva o non regolata. Le mutazioni alla base
di questo effetto sono in genere mutazioni puntiformi
missense o mutazioni troncanti che determinano la delezione
di domini autoinibitori della proteina
Ras
Notch
WT
Proteolisi
Mutante
Nucleo
Mutazioni caratteristiche delle patologie dominanti
• La proteina acquisisce una nuova caratteristica, che non ha
niente a che fare con il fisiologico meccanismo d’azione. Le
mutazioni responsabili sono in genere di tipo puntiforme
missense o troncanti. Tuttavia uno dei casi più tipici è quello
della espansione di triplette CAG codificanti poliglutamina.
• La proteina mutata interferisce con quella non mutata
eliminandone la funzione (meccanismo dominante negativo).
Questo si verifica di solito quando la funzione della proteina
in questione dipende dalla formazione di multimeri.
Mutazioni puntiformi missense o troncanti . Altro esempio è
quello delle patologie del collagene.
Osteogenesi imperfetta
Mutazioni caratteristiche delle patologie dominanti
• Raramente può succedere che, nel caso di una mutazione
inattivante di un allele, quello rimasto non sia sufficiente a
compensare (fenomeno dell’aploinsufficienza). In questo caso
comunque molto spesso gli omozigoti hanno un fenotipo
molto più grave degli eterozigoti.
Attività
Omozigote
normale
Eterzigote
Eterzigote
compensato aploinsufficienza
100%
50%
Fenotipo
normale
Fenotipo
normale
50%
Fenotipo
patologico
A volte mutazioni diverse a carico di uno stesso gene
possono determinare fenotipi completamente diversi
Gene PAX3
• Fattore trascrizionale che regola lo sviluppo dei melanociti
• Le mutazioni con perdita di funzione determinano la
sindrome di Waardenburg (difetti di pigmentazione cutanea +
sordità)
•Le mutazioni con acquisizione di funzione (traslocazione e
produzione di nuovi fattori trascrizionali ibridi) sono
oncogenesi (rabdomiosarcoma)
Ricordare sempre che tutti i fenotipi patologici
possono essere pesantemente influenzati dalla
contemporanea presenza di varianti di altri geni, detti
geni modificatori. I geni modificatori sono alla base
dei fenomeni di penetranza ed espressività variabile.
Chinasi
A
P-A
Fosfatasi
Funzione