
Clonaggio del DNA
a che cosa serve avere dei frammenti di DNA specifici:
-per poterlo analizzare piu’ facilmente isolatamente e non insieme a tutto
il resto del DNA genomico della specie su cui si vuole lavorare
-per avere delle sonde con cui andare ad isolare altri frammenti di DNA
limitrofo non ancora noto
-per poter poi fare delle mappe di restrizione, ossia delle mappe
genetiche fisiche in cui c’e’ corrispondenza tra lunghezza dei
frammenti e mappa, diversamente dalle mappe genetiche basate
sulle frequenze di ricombinazione ottenute con gli incroci
Dalle mappe genetiche in centimorgan alle mappe fisiche, i marcatori
genetici comunque sono stati prima mappati e poi siè giunti ad una
analisi sempre piu’ fine. La trasduzione è un sistema naturale con cui
sono stati isolati frammenti di DNA batterico come in un clonaggio
naturale. Negli organismi eucariotici e’ stata trovata presto l’omologia tra
geni di specie diverse e cosi’ clonati geni delle specie anche lontane.
Clonaggio del DNA
per poter clonare un frammento di DNA se ne devono conoscere alcune
caratteristiche
-si possono clonare frammenti che sfruttano l’omologia con dei
frammenti gia’ clonati
-oppure che hanno delle parti in comune con altre regioni di DNA note
-il metodo piu’ingenuo e’ quello di partire da frammenti ignoti e da
mappe cromosomiche trovarne la corrispondenza per poter avere
dei marcatori molecolari.
Nei procarioti e’ stato molto piu’ semplice andare ad identificare le
regioni genetiche a cui corrispondono dei frammenti di DNA. La
coniugazione batterica permette di avere dei merozigoti con cui si
possono ottenere facilmente dei ricombinanti con i marcatori voluti, con
l’aiuto anche di mappe di delezione si riescono ad individuare le regioni
corrispondenti ai marcatori. Il fenomeno della trasduzione e’ il metodo
naturale fisiologico del clonaggio di frammenti di DNA.
L’uso di enzimi di restrizione e’ stato il passo successivo per arrivare alle
mappe fisiche del DNA ed al clonaggio di frammenti di DNA.
Materiale necessario per clonare
Enzimi di restrizione e ligasi (eventualmente DNAsi, terminal transferasi
DNA polimerasi)
Vettori per il clonaggio: plasmidi, fagi
Tecniche di purificazione ed estrazione del DNA (inizialmente dopo la
lisi cellulare si usava centrifugazione su gradienti di Cesio o altro)
-ricordarsi l’esp. della semiconservativita’ della duplicaz. del DNA
di Meselson e StahlCosa significa clonare, da dove deriva la parola clone: in Microbiologia
alla parola clone corrisponde una colonia batterica o un placca virale o
fagica. Una colonia isolata su piastra Petri su terreno solido (agarizzato)
deriva da una singola cellula e quindi omogenea dal punto di vista
genetico, ogni cellula e’ geneticamente uguale alle altre.
In una cultura liquida ci possono essere dei mutanti che si mescolano tra
le altre cellule e quindi non essere perfettamente omogenea.
- ricordarsi del test di fluttuazione di S.Luria e Max Delbrück L’eccezione e’ data dal mosaicismo, evento raro.
Vettori: Plasmidi, Virus, Batteriofagi, Cromosomi artificiali
plasmidi, sono i vettori piu’ semplici ed i primi ad esser stati utilizzati
perche’ si replicano nei batteri e si purificano facilmente, sono presenti in
molte copie nei batteri, la maggior parte deriva da colEI (plasmide di
E.coli)
- Inizialmente si usavano per il clonaggio i siti di restrizione naturali del
plasmide, fino a quando sono state inserite delle mutazioni artificiali in
una regione del plasmide che non provocava danni dal punto di vista
funzionale del plasmide, i plasmidi possono portare la resistenza ad
antibiotici (data da un enzima che li metabolizza come la b-lactamasi)
che risultano utili per la selezione delle cellule trasformate col plasmide.
- Introdurre i plasmidi in cellule batteriche si dice trasformare e cio’
avviene in cellule che hanno la capacita’ di assorbire DNA attraverso la
membrana, impropriamente dette competenti,dopo trattamento con CaCl2
oppure con altri metodi come l’elettroporazione o shock termico.
Nei plasmidi per utilita’ sono state introdotte varie modifiche: piu’
resistenze ad antibiotici, un sito multiplo di clonaggio e altre funzioni.
Strutture necessarie ed utili per un plasmide
- origine di replicazione
- resistenza ad un antibiotico o due (ampicillina, tetraciclina,
canamicina, eritromicina, gentamicina, cloramfenicolo)
- resistenza ad un secondo antibiotico eucariootico di mammifero
- sito multiplo di clonaggio (multiple cloning site MCS) con la sequenza
taglio unica per gli enzimi di rstrizione piu’ comuni (non ve ne
sono altri in tutto il plasmide)
- gene reporter (b-gal) nel MCS indicatore dell’efficienza di clonaggio
per evidenziare colonie bianche e bleu (X-gal, IPTG)
Tipi di vettori: per clonaggio e sottoclonaggi, per librerie genomiche,
per cDNA, per espressione
Analisi con enzimi di restrizione:
Gli enzimi di rewstrizione tagliano qualunque DNA in corrispondenza di
sequenze specifiche. Per esempio: BamHI G’GATCC
Bgl II
A’GATCT
Eco RI
G’AATTC
Hind III
A’AGCTT
Kpn I
GGTAC’C
Pst I
CTGCA’G
Sma I
GGG’CCC
Hae IIII
GG’CC
Ecor RV
GAT’ATC
alcuni tagliano al centro della sequenza palindromica del sito di
restrizione (EcoRV, Hae,Sma) altri al 5’(Bam,B gl,EcoRI,Hind) ed altri
al 3’(Kpn, Pst)
Come conseguenza di questo le estremita’ del taglio sono pareggiate
quando il taglio e’ centrale, altrimenti sporgono al 5’ o al 3’.
Clonaggio costituito di varie fasi:
Estrazione del DNA (sia plasmidico che genomico da clonare) digestione
o amplificazione (per PCR)
Ligasi tra i DNA dell’ inserto
e del plasmide
Trasformazione batterica o di cellule eucariotiche o impacchettamento
dei fagi l o M13 (per estrusione) con helper
Selezione dei batteri per la resistenza all’antibiotico (ampicillina)
o altri per cellule eucariotiche, per i fagi selez. per vitalita’
Individuazione (screening) dei batteri con plasmide ricombinante
con gene indicatore (lac z: blue/binaco)
Estrazione del DNA plasmidico trasformante (mini-midi-maxi prep)
Tipi di clonaggio
La b-elica del DNA e’ doppia, ognuna orientata col verso 5’ 3’ (con la
numerazione degli atomi di carbonio del desossiribosio) che e’ il verso
della sintesi per opera delle polimerasi sia del DNA che dell’ RNA
5’ protruding (sporgenti) Hind III
3’ 5’
NNNA 3’
5’ AGCTTNNN
NNNT TCGA 5’
3’ ANNN
NNNA AGCTTNNN
NNNT TCGA ANNN
5’ 3’
3’ protruding Kpn I
3’ 5’
NNNGGTAC CNNN
NNNC CATGGNNN
NNNGGTAC 3’
5’CNNN
NNNC 5’
3’CATGNNN
5’ 3’
blunt ends (lisce, piatte) SmaI
NNNCCC 3’ 5’GGGNNN
NNNGGG 5’ 3’CCCNNN
Gli estremi dei frammenti di DNA ottenuti con degli enzimi di
restrizione possono quindi essere utilizzati per clonare ;
la digestione del DNA deve essere controllata, alcuni enzimi sono
sensibili e quindi inattivi se trovano delle basi metilate nella sequenza
specifica di taglio.
Si possono scegliere strategie diverse di clonaggio: lo stesso sito di
restrizione, uso di enzimi “isoschizomeri” che hanno lo stesso sito di
taglio o parte di esso come BamHI
e
Sau3A
nnnG GATCC nnn
nnn GATCnnnn
nnn CCTAG Gnnn
nnn CTAG nnnn
oppure siti di taglio ad estremita’ lisce (piatte) come SmaI, alle quali si
possono attaccare altre estremita’ lisce senza ricreare il sito di restrizione
oppure conservandolo
Un sito di restrizione “protruding” puo’ essere reso liscio con una
exonucleasi o con una polimerasi togliendo o aggiungendo le basi
mancanti.
Per clonare freammenti prodotti da PCR a seconda delle polimerasi si
possono avere estremita’ lisce o “protruding”
I vettori :
Vettori di clonaggio: plasmidi, cosmidi e fagi
- per sequenziamento: con elementi per avere singolo filamento di DNA
per clonare sonde ottenendo l’inserto a singolo filamento a DNAo
RNA
- i vettori di espressione in procarioti ed eucarioti (shuttle) recentemente
con possibilita’ di espressione controllata
- per studiare l’attivita’di promotori ed enhancers con geni reporter:
lac-z (b gal), CAT assay, luciferasi, GFP,
Con promotore costitutivo o inducibile (SV-40, CMV, HS Tk ecc.)
In fusione con un gene reporter al 5’ o al 3’
poly istidine (Invitrogen)
glutatione trasferasi (GST) pGEX
Sistema per studiare interazione tra proteine: il doppio ibrido, sorta di
complementazione, Two hybrid vectors system
(se ne parla a parte)
CORSO DI BIOLOGIA APPLICATA E PRINCIPI DI GENOMICA
A.A. 2007 BU
Prof. Domenico Frezza
Lezione N.2
A COSA SERVE il CLONAGGIO del DNA??
a creare ‘copie identiche’ (= CLONI) di un
frammento di DNA
DIFFERENZE con la PCR:
• è una reazione in vivo
sfrutta l’apparato di replicazione del DNA delle cellule batteriche
si fanno duplicare le cellule
• non necessita nessuna conoscenza a priori della sequenza del frammento
di DNA da clonare
non si usano primers
• è un processo più accurato
le copie di DNA ottenute sono esattamente identiche l’una all’altra
sistemi di ‘correzione degli errori’ cellulari
A CHE COSA SERVE AVERE DEI FRAMMENTI DI DNA SPECIFICI?
-per analizzarlo più facilmente isolatamente e non con tutto il resto
del DNA genomico della specie su cui si vuole lavorare;
-per avere delle sonde con cui andare ad isolare altri frammenti di
DNA limitrofo non ancora noto;
-per fare delle mappe di restrizione, ossia delle mappe genetiche
fisiche in cui c’è corrispondenza tra lunghezza dei frammenti e
mappa.
TRAMITE il CLONAGGIO
• individuare nuovi geni da sequenziare
librerie geniche
• stabilire quali geni sono espressi in ogni tipo cellulare
librerie di cDNA
• produzione di molecole ricombinanti
vettori di espressione (es. insulina umana)
COME FUNZIONA il CLONAGGIO del DNA?
PREMESSE:
1) I BATTERI
CELLULE PROCARIOTICHE:
assenza nucleo
singola molecola circolare di DNA (3-4 M bp) nel
citoplasma
possono acquisire facilmente piccole molecole di DNA esogeno
2) I PLASMIDI
piccole molecole circolari di DNA (3000-4000 bp)
che portano pochi geni
possono facilmente essere acquisiti dalle cellule
batteriche
COME FUNZIONA il CLONAGGIO del DNA?
PREMESSE:
3) GLI ENZIMI DI
RESTRIZIONE
ENDONUCLEASI che tagliano il
DNA in corrispondenza di
specifiche sequenze di basi
Estremità piatte o blunt
Estremità coesive o sticky
COME FUNZIONA il CLONAGGIO del DNA?
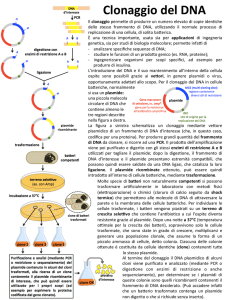
ENZIMA di RESTRIZIONE
1.
Il DNA da clonare viene
tagliato con un enzima di
restrizione
2.
inserito in un plasmide
tagliato con lo stesso enzima
3.
ad opera della LIGASI o
della TOPOISOMERASI
LIGASI TOPOISOMERASI
PLASMIDE
PLASMIDE RICOMBINANTE
CELLULA BATTERICA
4.
Il PLASMIDE RICOMBINATE
viene inserito in una cellula
batterica e al suo interno si
duplica
PUO’ SUCCEDERE CHE…
1)
La cellula non acquisisce il plasmide e quindi neanche il DNA da clonare
2)
Il plasmide si richiude su se stesso senza acquisire il DNA da clonare
la cellula acquisisce il plasmide ma NON il DNA da clonare
STRATEGIE per VERIFICARE che il CLONAGGIO è AVVENUTO:
1) Il plasmide contiene un gene che conferisce alla cellula la resistenza ad un antibiotico
le cellule vengono fatte crescere (e duplicare) in un terreno selettivo contente
quell’antibiotico: SOLO le CELLULE CONTENENTI il PLASMIDE POSSONO
DUPLICARSI nel terreno contenete l’antibiotico
2) Il DNA da clonare viene inserito all’interno di un gene che codifica per un enzima
(che compie una determinata reazione metabolica) impedendo la sintesi di tale
enzima
le CELLULE CONTENENTI il PRODOTTO della REAZIONE CATALIZZATA
dall’ENZIMA NON CONTENGONO il DNA CLONATO
UN ESEMPIO: il TOPO-TA Cloning
• il plasmide è tagliato in un sito che lascia
delle estremità coesive di Timina mentre al
frammento da clonare sono aggiunte delle
estremità coesive di Adenina
• il legame tra plasimde e DNA da clonare è
fatto dall’enzima Topoisomerasi
• il plasmide porta i geni per la
resistenza agli antibiotici ampicillina
e kanamicina
• il sito di inserzione del DNA da
clonare spezza la sequenza del gene
lacZ (b-galattosidasi)
La b-GALATTOSIDASI e l’X-gal
La b-galattosidasi è un enzima coivolto
nella prima tappa del metabolismo del
galattosio
le cellule in cui viene sintetizzata la b-galattosidasi
(il frammento da clonare non si è inserito)
appaiono di colore BLU
le cellule senza b-galattosidasi (il frammento da
clonare si è inserito) appaiono di colore BIANCO
L’ X-gal è un
omologo del
galattosio che
viene scisso dalla
b-galattosidasi in
due prodotti uno
dei quali è di colore
blu intenso
IN PRATICA:
Le FASI deL CLONAGGIO
1.
Preparazione del terreno di coltura e delle piastre di crescita
2. Preparazione del DNA da clonare
purificazione
creazione delle estremità coesive o “blunt”
3. Inserzione del DNA da clonare nel plasmide
4. Inserzione del plasmide nelle cellule batteriche
trasformazione
5. Piastramento delle cellule e crescita
6. Recupero degli inserti o ampliconi se si usa PCR



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)






![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)