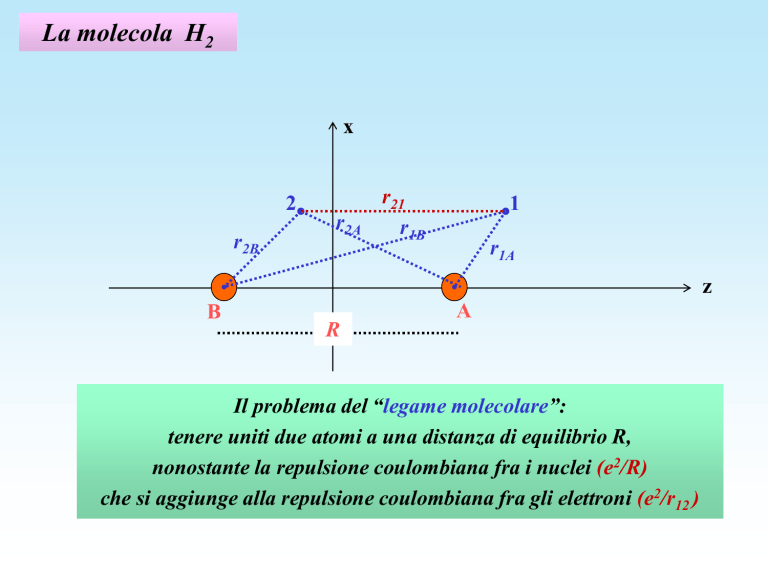
La molecola H2
x
2
r2B
r2A
r21
r1B
1
r1A
z
B
R
A
Il problema del “legame molecolare”:
tenere uniti due atomi a una distanza di equilibrio R,
nonostante la repulsione coulombiana fra i nuclei (e2/R)
che si aggiunge alla repulsione coulombiana fra gli elettroni (e2/r12 )
come si misurano
le dimensioni di una molecola
sorgente
R
un fenomeno analogo:
le “riflessioni” dei colori sul CD
i2
occhio
li3
la differenza fra i cammini
d2
d12= li2 - li1 + ld2- ld1
interferenza costruttiva per
d12=
k
ld4
li4
ld2
ld3
li1
h
li2
ld1
a2
d34= li4 - li3 + ld4- ld3
interferenza costruttiva per
d34=
a2+p
a4
A
a4+p
metodo della “somma sui cammini” di Feynman: contribuiscono tutti i cammini che non sono “proibiti”
(“QED, la strana teoria della luce”- per una versione didattica si veda http://www.iapht.unito.it/qm)
l i 2 li1
h 2 a 22 h 2 a 2 p 2
calcolo del passo p fra i solchi
h 2 a 22 h 2 a 22 2a 2 p
sorgente
a 2 p
l i 2 l i 2 1
sen i 2 p
2
li 2
l d 2 l d1 sen d 2 p
occhio
inserendo i dati della figura
per il blu ( 450 nm): k
d12= li2 - li1 + ld2- ld1
p |sen i2- sen d2| 0.3 p
i4 50o
i2 20o
d2 45o
d2
ld4
li3
15o
li4
ld2
ld3
h
li2
ld1
per il rosso ( 650 nm):
d34= li4 - li3 + ld4- ld3
a2
p |sen i4 - sen d4| 0.4 p
a2+p
a4
p 1,2 m
li1
A
a4+p
nel caso della molecola ...
d12= l2 - l1 R sen 2
l3
interferenza costruttiva per
d12= R sen 2
l4
R
sorgente
RÅ
l1
l2
Å
raggi X
2
rivelatore
l3
l4
l’3
l’4
R
l’2
l2
l1
l’1
tipico apparato
sperimentale
sorgente di
raggi X
campione da
esaminare
rivelatore
altre configurazioni
- asse della molecola non perpendicolare al fascio
d1
l1 = l + d1
l2 = l
R
l
l
d1 = R sin
fasci di elettroni o di neutroni
- energia cinetica di un elettrone con lunghezza d’onda 10-10 m :
2
1
2c
Ekin
2
2
2
2mc
2mc
2mc
( pc )2
7
( ck )2
2
12.56 10 eVm
1
2
10
eV
10
106 eV
10
m
- per un neutrone Ekin 2000 volte di meno 50 meV (energie “termiche”)
x
La molecola H2
2
r2B
r2
r12
r2A
1
r1B
r1
r1A
z
B
R
A
Hamiltoniana:
p2A
pB2
e2 p12
p22 e2 e2 e2 e2 e2
H ( r1, r2 , RA, RB )
2 M N 2 M N R 2me 2me r12 r1A r1B r2 A r2 B
termini che dipendono
solo dalle coordinate
dei nuclei
termini che dipendono
solo dalle coordinate
degli elettroni
termini che “mescolano” le
coordinate degli elettroni e
quelle dei nuclei
Equazione di Schrödinger:
La molecola H2
H ( r1, r2 , R A , RB ) ( r1, r2 , R A , RB ) E ( r1, r2 , R A , RB )
Approssimazione di Born-Oppenheimer:
data la grossa differenza fra la massa dell’elettrone e quella dei nuclei, è lecito
trascurare la variazione delle posizioni dei nuclei nella soluzione del moto degli
elettroni e risolvere l’equazione con una funzione d’onda prodotto della
funzione d’onda nucleare per una funzione d’onda elettronica con i nuclei
fermi a una distanza R.
funzione d’onda
R interviene come
funzione d’onda
elettronica con i nuclei
parametro e non come
nucleare
a distanza fissa R
variabile.
( r1, r2 , R A , RB ) R ( r1, r2 ) ( R A , RB )
energia degli elettroni con
i nuclei fissi a una
distanza R (non
necessariamente uguale a
quella di equilibrio)
R
H ( r1, r2 , RA , RB ) H el ( r1, r2 ) H nucl ( RA , RB )
R
R
H el ( r1, r2 ) R ( r1, r2 ) Eel R ( r1, r2 )
x
molecola ione-idrogeno
rB
2
2
2
p
e
e
H elR (r )
2me rA rB
B
potenziale coulombiano
repulsione fra i
due nuclei
20
10
0
energia (eV)
-10
-20
-30
elettrone-nuclei
-40
-50
-60
-70
-80
-8
-7
-6
-5
-4
-3
-2
-1
0
1
z (angstrom)
2
R
3
4
5
6
7
8
r rA
A
z
La funzione d’onda molecolare |g> è scritta come
sovrapposizione lineare di funzioni d’onda che risolvono
l’equazione di Schroedinger per l’atomo isolato:
funzioni
funzione
d’onda
1
d’onda
g
1s(rA ) 1s(rB )
atomiche
2
molecolare
Metodo LCAO:
Linear
Combination
of
Atomic
Orbitals
2
p 2 e2 e2
e
1s ( rA ) E1s 1s ( rA )
1s ( rA )
2m rA rB
rB
p 2 e2 e2
e2
1s ( rB ) E1s 1s( rB )
1s( rB )
2m rA rB
rA
2
2
p 2 e2 e2
e
e
1s( rA ) 1s( rB ) E1s 1s( rA ) 1s( rB )
1s( rA )
1s( rB )
2m rA rB
rB
rA
termine di
energia atomica
termini che si aggiungono
grazie all’attrazione da
parte “dell’altro nucleo”
1 x 2 y 2 ( z z A )2 / ao
x 2 y 2 ( z z B ) 2 / ao
1s
e
g (r )
e
2
tutte le funzioni sono mostrate
per valori
1s - sigma
geradefissi, diversi da 0, di x e y
5
x
1sg(z)
4
rB
funzione d'onda
4
1s(z-zA)
1s(z-zB)
3
B
R
r rA
A
3
2
molecola ioneidrogeno:
funzione d’onda
“gerade”
2
1
1
0
-7
-6
-5
-4
-3
-2
-1 0 1
2
z (angstrom)
3
4
5
6
7
z
H
g | H | g
energia della
funzione d’onda “gerade”
g | g
p 2 e2 e2
1
g H g 1s( rA ) 1s( rB )
1s( rA ) 1s( rB )
2
2m rA rB
p 2 e2 e2
1s( rA )
1s( rA ) 1s( rB ) E1s 1s( rA ) 1s( rA ) 1s( rB ) 1s( rA )
2m rA rB
e2
1s( rA ) 1s( rA )
rB
e2
1s( rB )
rA
p 2 e2 e2
e2
e2
1s( rB )
1s( rA ) 1s( rB ) E1s 1s( rB ) 1s( rA ) 1s( rB ) 1s( rB )
1s( rA ) 1s( rB )
1s( rB )
2m rA rB
rB
rA
p 2 e2 e2
1s(rA ) 1s(rB )
1s(rA ) 1s(rB ) E1s 1s(rA ) 1s (rB ) 1s (rA ) 1s(rB )
2m rA rB
energia
coulombiana
2
e2
e
1s (rA ) 1s (rA ) 1s(rB ) 1s(rB )
rB
rA
energia di
risonanza
2
e2
e
2 1s(rA ) 1s(rB ) 1s(rA ) 1s(rB )
rB
rA
energia della funzione d’onda “gerade”
termini coulombiani
C
g H g E1s g g
1
e2
e2
1s (rA )
1s (rA ) 1s (rB )
1s (rB )
2
rB
rA
2
2
e
e
1s (rB )
1s (rA ) 1s (rA )
1s (rB )
rB
rA
x
rB
Q
B
termini di “risonanza”
e2
C 1s( rA )
1s( rA )
rA
e
e2
Q 1s(rB ) 1s(rA ) e
rB
R
e2
2 x 2 y 2 ( z z A ) 2 / ao
2
2
x y ( z zB )
x 2 y 2 ( z z A ) 2 x 2 y 2 ( z z B ) 2 / ao
2
r rA
A
z
dx dy dz
e2
x 2 y 2 ( z zB )2
dx dy dz
energia della funzione d’onda “gerade”
Normalizzazione della
funzione d’onda:
termini di “sovrapposizione”
1
g g 1s( rA ) 1s( rB ) 1s( rA ) 1s( rB )
S
2
1
1
1s( rA ) 1s( rA ) 1s( rB ) 1s( rB ) 1s( rA ) 1s( r B) 1s( rB ) 1s( rA )
2
2
1
S 1s( rB ) 1s( rA )
<g| g> = 1 + S
x 2 y 2 ( z z A ) 2 x 2 y 2 ( z z B ) 2 / ao
e
dx dy dz
H
g
g H g
g g
C Q
E1s
1 S
da ricordare che tutti i termini (C, Q, S) sono determinati
per un certo valore della distanza interatomica R
molecola ione-idrogeno: energia della
funzione d’onda “gerade”
1sg(r)= (1s(rA)+1s(rB))/2
x
energie
10
rB
repulsione fra i
nuclei
8
somma
6
energia (eV)
4
B
0
z
R
distanza di
equilibrio,
Ro1,06 Å
2
r rA
A
livello energetico dell’atomo
isolato (R = infinito)
-2
attrazione
coulombiana da parte
alla distanza di equilibrio:
dell’altro nucleo
-4
-6
-8
energia di risonanza
H
-10
0,0
1,0
2,0
3,0
distanza fra nuclei (angstrom)
4,0
5,0
g
e2
E1s 2,8 eV
Ro
“legame
molecolare”
1su(r)= 1/2 (1s(rA)-1s(rB))
1 x 2 y 2 ( z z A )2 / ao
x 2 y 2 ( z z B )2 / ao
e
1s u ( r )
e
2
x
1s - sigma ungerade
rB
8
6
1s(rA)
B
Rint
4
funzione d'onda
r rA
A
1su(r)
2
0
-2
molecola
ioneidrogeno:
funzione
d’onda
“ungerade”
-1s(rB)
-4
-6
-8
-4
-3
-2
-1
0
1
z (angstrom)
2
3
4
z
molecola ione-idrogeno: energia della
funzione d’onda “ungerade”
1su(r)= 1/2 (1s(rA)-1s(rB))
energie
x
50
rB
40
B
energia (eV)
30
repulsione fra i
nuclei
somma
0
z
Rint
20
10
r rA
A
energia di
risonanza
attrazione da parte
dell’altro nucleo
-10
-20
0,0
1,0
2,0
3,0
distanza fra nuclei (angstrom)
4,0
H
<u| u> = 1 - S
u H u
C Q
E1s
u u
1 S
5,0
u
1s - sigma gerade
“orbitali” molecolari
-1s
5
4
funzione d'onda
4
3
3
2
2
1
1
0
-7
-6
-5
-4
-3
-2
-1
0
1
z (angstrom)
2
3
4
1s - sigma ungerade
8
6
funzione d'onda
4
2
0
-2
-4
-6
-8
-4
-3
-2
-1
0
1
z (angstrom)
2
3
4
5
6
7
x
r12
2
r2B
r2
La molecola H2
r2A
1
r1B
r1A
r1
z
B
A
R
2
2
2
2
2
2
2
p
p
e
e
e
e
e
p
p
e 2e
e
e
e
H 1
H 2m 2
m r
r r
r
r
m
r2 A r2 B r12
( r2
, r ) ( r )2
m
( r ) r
1
A
/ 2 r1B
2
1
1 2
2
2
g 1
2
2
2
2
2
1A
1B
2A
2B
12
g 2
1 2
1 2
funzione d’onda:
- antisimmetrica nello scambio delle funzioni di spin
dei due elettroni,
- simmetrica nello scambio delle funzioni spaziali
( r1, r2 ) g ( r1 ) g ( r2 ) 12 12 / 2
x La molecola H
2
p12 p22 e2 e2 e2
e2 e2
H
2m 2m r1A r1B r2 A r2 B r12
H
(o )
(o )
Hm
H
,1
m,2
p12 e2 e2
(o )
H m,1
2m r1A r1B
H m(o,1)
H m(o,)2
g
g
;
2
r2B
2
e
r12
p22 e2
e2
(o )
H m,2
2m r2 A r2 B
B
r2
r12
r2A
1
r1B
r1A
r1
R
z
A
(o)
g(r1 ) H m,1 g(r1 )
(o)
g(r2 ) H m, 2 g(r2 )
2
e
(o )
(o )
H g H m,1 H m,2 g( r1) g( r2 )
g( r1) g( r2 )
r12
g
g
alla distanza di equilibrio
Ro 0,7 Å
H
g
e2
2 E1s 4,7 eV
Ro
“legame molecolare”
primi livelli energetici
di molecole biatomiche
energia
orbitale molecolare
orbitale dell’atomo 1
2s
orbitale dell’atomo 2
(2)
2s u
2s
(2)
(2)
(2)
(molteplicità)
(2)
1s
(2)
(2)
2s
g
1s u
1s
g
(molteplicità)
1s
(2)
le molecole biatomiche
dall’idrogeno al berillio
legante
antilegante
l’energia di dissociazione è la
somma dell’energia elettronica,
della repulsione fra i nuclei e
dell’energia “zero vibrazionale”
2pz
“orbitali” atomici 2pz
0,15
funzione d'onda
0,10
0,05
0,00
-0,05
0
2 pz ( r ) R21 ( r )Y1 ( , )
-0,10
-0,15
-8 -7 -6 -5 -4 -3 -2 -1 0 1 2
z (angstrom)
3
4
5
6
7
8
Cre
r / ao
Cze r / ao
cos
“orbitali” molecolari 2pz
2pz - sigma gerade
0
g 2pz (r)
0
g 2 pz ( r )
0
C ' [( z z A )e rA / ao ( z zB )e rB / ao ]
0
0
0
2pz - sigma ungerade
0
0
0
u 2pz (r)
2pz (rA)
-2pz (rB)
0
0
0
-7
-6
-5
-4
-3
-2
-1 0
1
2
z (angstrom)
funzione d'onda
funzione d'onda
0
u 2 pz ( r )
C ' [( z z A )e
0
3
0
4
5
6
7
2pz (rB)
0
0
2pz (rA)
0
rA / ao
( z z B )e
0
rB / ao
0
-7
-6
u 2pz (r)
]
-5
-4
-3
-2
-1 0
1
2
z (angstrom)
3
4
5
6
7
“orbitali”
molecolari
-2pz
2pz - sigma gerade
0
g 2pz (r)
0
0
funzione d'onda
0
0
0
0
0
0
0
0
-7
-6
-5
-4
-3
-2
-1
0
1
z (angstrom)
2
3
4
5
6
2pz - sigma ungerade
0
u 2pz (r)
0
funzione d'onda
0
0
0
0
0
0
0
-7
-6
-5
-4
-3
-2
-1
0
1
z (angstrom)
2
3
4
5
6
7
7
“orbitali” atomici 2px
2 p ( r ) R21
( r )Y11( , ) Cre r / ao sen ei
2px
2 p ( r ) R21 ( r )Y11( , ) Cre r / ao sen e i
0,25
1
2 p ( r ) 2 p ( r ) C ' xe r / ao
2 px (r )
2
0,20
X
0,15
funzione d'onda
andamento0,10a x>0
0,05
0,00
-0,05
-0,10
-0,15
-0,20
-0,25
andamento
a x<0
-8 -7 -6 -5 -4 -3 -2 -1 0 1 2
z (angstrom)
2 p ( r ) R21 (r )Y11( , ) Cre r / ao sen ei
3
4
5
6
7
8
2 p ( r ) R21 ( r )Y11( , ) Cre r / ao sen ei
1
2 p (r ) 2 p (r ) C' xer / ao
2 px ( r )
2
“orbitali” molecolari 2px
u 2 px (r )
rA / ao
rB / ao
C
'
[
x
e
x
e
]
2px - pigreco ungerade
0.3
0.2
funzione d'onda
0.2
andamenti a x>0
0.1
0.1
2px (rA)
2px (rB)
0.0
2p xu(r)
-0.1
-0.1
-0.2
-0.2
andamenti a x<0
-0.3
-8 -7 -6
-5 -4 -3 -2 -1 0 1 2
z (angstrom)
3
4
5
6
7
8
“orbitali” molecolari 2px
2px - pigreco ungerade
0
0
0
funzione d'onda
0
0
andamento a x>0
0
0
0
0
0
0
-8
-7
-6
-5
-4
-3
andamento a x<0
-2
-1 0 1 2
z (angstrom)
3
u 2 px (r )
C ' [ x e rA / ao x e rB / ao ]
4
5
6
7
8
“orbitali” molecolari g 2px
2px - pigreco gerade
0
0
0
funzione d'onda
0
0
andamento a x>0
0
0
0
0
0
andamento
a x<0
0
-8
-7 -6
-5
-4 -3
-2 -1 0 1 2
z (angstrom)
3
4
g 2 px (r )
C'[ x e
rA / ao
xe
rB / ao
]
5
6
7
8
orbitale molecolare
energia
(2)
(4)
2p
(6)
(2)
Livelli
energetici
di molecole
biatomiche
2pzu
2p±
g
2pz
2p
(6)
g
(4)
2p±u
orbitale dell’atomo 1
2s
orbitale dell’atomo 2
(2)
2s u
2s
(2)
(2)
(2)
(2)
1s
(2)
(2)
2s
g
1s u
1s
g
1s
(2)
(molteplicità)
legante
legante
antilegante
molecole
biatomiche
molecole biatomiche
eteronucleari: legame ionico
attrazione fra gli ioni
repulsione fra i nuclei
e gli elettroni interni
2
e
b
E p
R R9
nel punto di equilibrio Ro:
dE p
e
2
9b
2 10 0
dR R
R
b
e
2
Ro8
9
2
energia nel punto di minimo:
8e
Em
9 Ro
e 2 e 2 Ro8
E p
R 9R 9
per Na Cl, Em -5,12 eV
(togliendo 1,42 eV di
ionizzazione si ottiene
-3,7 eV)
molecole biatomiche
eteronucleari
energia di dissociazione:
possibili meccanismi
radiazione:
2 πc 1260 eV nm
E
E
esempio: per O2 , E 5 eV, 240 nm
per N2 , E 7 eV, 170 nm
N2 e O2 sono “trasparenti” all’UV
UV A 380 nm > > 320 nm
UV B 320 nm > > 280 nm
UV C 280 nm > > 180 nm
UV C
UV oltre C
eccitazione non radiativa
(urti fra molecole):
distribuzione di Boltzmann:
energia di dissociazione:
possibili meccanismi
N E Eo e Eo / k BT
probabilità relativa di due sistemi di energie E1 ed E2:
es: N2 + O2
2 NO
(kB= costante
di Boltzmann
10-4 eV K-1)
P e ( E1 E2 ) / k BT
Ediss, N2 = -7,4 eV; Ediss, O2 = -5,1 eV
Ediss, NO = -5,3 eV; E1-E2 2 eV
a 300 K, kBT 0,03 eV P e-2/0,03 10-30
a 1500 K (motore Diesel), kBT 0,15 eV P e-2/0,15 10-6
da 1g di N2 qualche g di NO!










