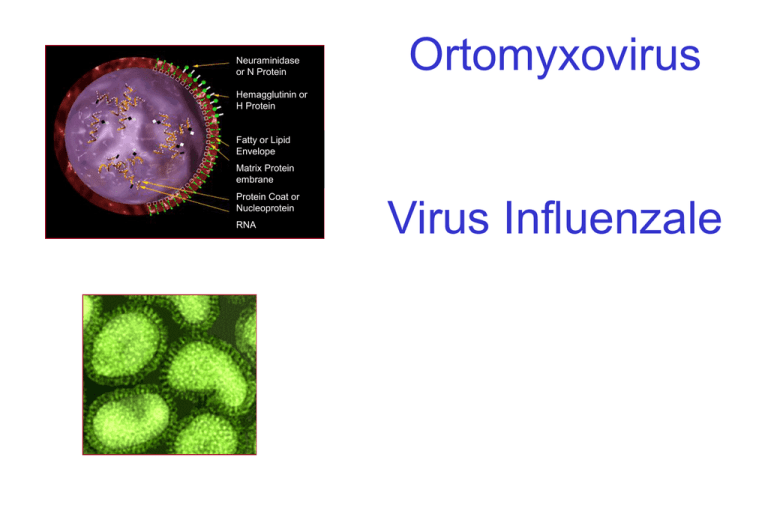
Neuraminidase
or N Protein
Ortomyxovirus
Hemagglutinin or
H Protein
Fatty or Lipid
Envelope
Matrix Protein
embrane
Protein Coat or
Nucleoprotein
RNA
Virus Influenzale
“INFLUENZA”
• influenza “vera”
– virus dell’influenza
• malattia dell’apparato respiratorio
associata a sintomi sistemici e causata da
una varietà di altri microrganismi spesso
chiamata “influenza”
ORTHOMYXOVIRUS
HA - emogglutinina
NA - neuraminidasi
nucleocapside elicoidale (RNA e
proteina NP)
lipid bilayer membrane
complesso della polimerasi
M1 protein
TIPO A, B, C : NP, M1 protein
SOTTO-TIPO: HA or NA protein
CICLO REPLICATIVO
ASSORBIMENTO
Processamento posttrascrizionale
packagin
Traduzione
ENDOCITOSI
mRNA
vRNA (-)
NUCLEO
Fusione e
uncoating
cRNA (+)
Principali antigeni virali
Gli antigeni INTERNI (proteine M1 e NP) sono le
proteine tipo-specifiche (A, B, C)
Gli antigeni ESTERNI (HA e NA) sono gli antigeni
sottotipo- e ceppo-specifici.
Nomenclatura
Nelle proteine esterne (HA e NA) si manifestano
le principali differenze fra i vari ceppi influenzali
Sono quindi utilizzate per indicare un
PARTICOLARE CEPPO DI INFLUENZA, secondo la
caratteristica denominazione “HxNy”
Sono le proteine che stimolano il nostro sistema
immunitario
Ab specifici
protezione
IL VIRUS INFLUENZALE NEL
TRATTO RESPIRATORIO
Il virus influenzale si
attacca (tramite
l’HA) alle cellule
epiteliali del tratto
respiratorio
Il virus replica
all’interno delle
cellule
La neuraminidasi virale
consente il rilascio
dei virioni
neoformati nella vie
aeree
COME SI TRASMETTE?
Turbinati nasali
Esofago
Trachea
Flusso d’aria
Bronchi
Bronchioli
Clearance mucociliare
Linfonodi
• Il virus viene trasmesso
tramite goccioline di saliva
starnutendo, tossendo o
semplicemente parlando
• La trasmissione è facilitata
dal contatto stretto
• È altamente contagioso e
trasmissibile
• I soggetti infetti sono
contagiosi da pochi giorni
prima e per i 5-7 giorni
successivi alla comparsa dei
sintomi
MANIFESTAZIONI CLINICHE
• Il periodo di incubazione può durare dalle 24 ore ai 5/6 giorni.
• L’inizio della malattia è improvviso, con comparsa di febbre, brividi,
mal di testa, gola secca, mialgia, malessere inappetenza e mal di
gola.
• Il sintomo più rilevante è la febbre , che in assenza di trattamento
antipiretico si innalza bruscamente nel corso di 24-48 ore fino ai
41°C.
• Lo spettro delle manifestazioni cliniche varia notevolmente per
gravità, da manifestazioni asintomatiche a complicazioni minori
quali rinite e/o faringite fino alla polmonite virale con esito
potenzialmente fatale
COMPLICAZIONI DELL’INFLUENZA
Respiratorie
• Polmonite virale
• Polmonite batterica
– da stafilococco
– da streptococco
Non-respiratorie
Miosite
Miocardite, pericardite
Mioglobinuria
depressione midollare
CNS
sindrome di Guillain-Barré
mielite trasversa
sindrome di Reye syndrome
Encefalite
INFLUENZA: LE COMPLICAZIONI
RESPIRATORIE
•
URTI
•
LRTI
- otite media
- sinusite
- esacerbazione di asma/broncopatia
- croup (un'ostruzione laringea
sottoglottica), bronchiolite
- polmonite virale primaria (rara*)
- polmonite batterica secondaria
* 5-10% delle polmoniti come complicanze
dell’influenza
INFLUENZA AL DI FUORI DEL TRATTO
RESPIRATORIO
• ? dati disponibili su modelli animali
• Influenza come causa riconosciuta di miocardite nei
cavalli
• Dati molto limitati sull’uomo
• La PCR potrà aiutare a riconoscere la presenza di
virus influenzale al di fuori del tratto respiratorio
PREVALENZA DEI SINTOMI
Tosse
Malessere
Sensazione di febbre
Esordio improvviso
Brividi
Cefalea
Anoressia
Congestione nasale
Mialgia
Mal di gola
Catarro
Vertigini
Abbassamento di voce
Dolore retrosternale
Vomito
Diarrea
Dolore addominale
Febbre >37.8C
0 10 20 30 40 50 60 70 80 90 100
Percentuale dei casi
Nicholson, 1998
CARATTERISTICHE CLINICHE DELL’INFEZIONE INFLUENZALE
40
39
temperatura orale (*C)
38
insorgenza
dei sintomi
37
giorni dopo
l’insorgenza dei sintomi
0
1
2
3
4
5
6
7
8
congestione nasale /
secrezione
mal di gola
dolori muscolari
mal di testa
tosse
anoressia
malessere
infettività/
isolamento virale
titolo anticorpale serico
< 4
8
(dal 6o al
21o giorno
ANTICORPI NEUTRALIZZANTI (AN)
Ab anti-neuroaminidasi nel siero
Ab neutralizzanti nel siero
102
101
100
+ + + + + +
105
Virus
104
40
103
30
Interferon
102
20
101
10
0
2
4
6
8
Giorni post- infezione
10
14
21
28
IFN nell’aspirato nasofaringeo (U/ml)
Titolo anticorpale (Reciprocal Geom. Mean)
103
Titolo virale nell’aspirato nasofaringeo (TCID50/ML)
Sintomi influenzali
LOG10 titolo virale massimo nell’aspirato
nasofaringeo (RMK TCID50 /ml)
Correlazione tra risposta febbrile e titolo virale
8
6
4
2
0
0
1
2
3
febbre
4
5
6
PRODUZIONE DI CITOCHINE DURANTE L’INFEZIONE
DA INFLUENZA A ACQUISITA NATURALMENTE
Concentrazione
media di citochine
nel lavaggio
nasale (pg/ml)
Hayden et al, 1998
DIAGNOSI DI INFLUENZA
La disponibilità di dati epidemiologici provenienti da un
sistema locale di sorveglianza migliora le possibilità di una
diagnosi clinica attendibile
Riconoscimento dell’antigene
Isolamento virale
Sierologia
(RT-PCR)
}
• lavaggio/tampone nasale
• aspirato nasofaringeo
• tampone faringeo, saliva
• test di inibizione
dell’emagglutinazione
• fissazione del
complemento
EPIDEMIE STAGIONALI
• Ogni anno nel periodo invernale si presentano
epidemie influenzali
• Ogni anno le popolazioni a rischio devono essere
vaccinate
• Ogni anni si presenta un NUOVO CEPPO
influenzale in grado di causare EPIDEMIE
PANDEMIE
Dall’inizio del ‘900 ad oggi si sono verificate 3
grandi pandemie influenzali con elevata mortalità
•1918-19
Spagnola
•500,000 USA
•20,000,000 mondiale
•1957-58
Asiatica
•70,000 USA
•1968-69
Hong Kong
•34,000 USA
• Come e perché si originano i ceppi
epidemici?
• Come e perché si originano i ceppi
pandemici?
VARIAZIONE ANTIGENE
Il virus influenzale A è soggetto a 2 forme distinte di variazioni
che riguardano le sue proteine superficiali:
- “antigenic drift” (“drift” antigenico): riflette cambiamenti
minori sia dell’HA che della NA, o di entrambe
- “major antigenic shift” (cambiamento maggiore): avviene
raramente e riflette la comparsa di ceppi virali con proteine di
superficie solo lontanamente correlati a quelli dei ceppi
precedenti. Generalmente riguarda l’HA
Antigenic drift (1)
Variazioni antigeniche MINORI (“drift”)
derivano da MUTAZIONI nei geni dell’HA e
della NA
Antigenic drift (2)
• HA e NA accumulano mutazioni
– virus a RNA
• la risposta immunitaria non garantisce una
protezione completa
• epidemie limitate
Antigenic shift
Cambiamenti antigenici MAGGIORI (“shift”)
sono molto diversi, con ampia variazione nella
sequenza amminoacidica
Genoma
• Il virus influenzale presenta un’organizzazione
del materiale genetico molto caratteristica
• Il virus dell’influenza ha un genoma costituito da
molecole di RNA
• A differenza di altri virus questo e’ suddiviso in
vari SEGMENTATI
– 8 segmenti diversi codificano proteine diverse
– Tutti e gli 8 frammenti sono necessari perché la
particella sia infettiva
Come avviene l’antigenic shift
Human
Virus virus
A
Non-human
Virus B virus
Reassortant
virus
Nuovo
virus
Antigenic shift (2)
• Lo shift si verifica quando 2 virus con 2 HA
diverse infettano la stessa cellula (A e B)
• Il virus che viene prodotto può essere
identico ad A eccetto per la sua HA che
deriva da B
INFLUENZA A, B o C
Esistono 3 tipi di virus influenzali: A, B, C
Virus influenzali A, infettano uomo e numerose
specie animali (UCCELLI!)
Virus influenzali B e C sono virus UMANI, non
isolati in uccelli (C è isolato da maiali e cani)
SOLO L’INFLUENZA DI TIPO A PUO’
CAUSARE PANDEMIE
• Come e perché si originano i ceppi
epidemici?
• Come e perché si originano i ceppi
pandemici?
VARIAZIONE ANTIGENE
Il virus influenzale A è soggetto a 2 forme distinte di variazioni
che riguardano le sue proteine superficiali:
- “antigenic drift” (“drift” antigenico): riflette cambiamenti
minori sia dell’HA che della NA, o di entrambe
- “major antigenic shift” (cambiamento maggiore): avviene
raramente e riflette la comparsa di ceppi virali con proteine di
superficie solo lontanamente correlati a quelli dei ceppi
precedenti. Generalmente riguarda l’HA
Antigenic drift (1)
Variazioni antigeniche MINORI (“drift”)
derivano da MUTAZIONI nei geni dell’HA e
della NA
Antigenic drift (2)
• HA e NA accumulano mutazioni
– virus a RNA
• la risposta immunitaria non garantisce una
protezione completa
• epidemie limitate
Antigenic shift
Cambiamenti antigenici MAGGIORI (“shift”)
sono molto diversi, con ampia variazione nella
sequenza amminoacidica
Genoma
• Il virus influenzale presenta un’organizzazione
del materiale genetico molto caratteristica
• Il virus dell’influenza ha un genoma costituito da
molecole di RNA
• A differenza di altri virus questo e’ suddiviso in
vari SEGMENTATI
– 8 segmenti diversi codificano proteine diverse
– Tutti e gli 8 frammenti sono necessari perché la
particella sia infettiva
Come avviene l’antigenic shift
Human
Virus virus
A
Non-human
Virus B virus
Reassortant
virus
Nuovo
virus
Antigenic shift (2)
• Lo shift si verifica quando 2 virus con 2 HA
diverse infettano la stessa cellula (A e B)
• Il virus che viene prodotto può essere
identico ad A eccetto per la sua HA che
deriva da B
Diversità delle HA
• In natura sono note ben 16 HA diverse e 9 NA
diverse
• Fino al 1997 nell’uomo hanno circolato solo la
HA1, HA2 e la HA3
• I 3 ceppi pandemici del 1918, 1957, 1968,
rispettivamente H1N1, H2N2, H3N2 sono i ceppi
ancora in circolazione
“Ribbon diagram” di alcune delle 16
HA
Trimeri di H1,
H3, H7 e H9
Diverso
orientamento
del dominio di
legame al
recettore in 4
divese HA
Dove sono le rimanenti 13 HA ?
I 16 tipi di HA e i 9 tipi di NA
sono TUTTI presenti negli
uccelli acquatici e selvatici
Questi sono il SERBATOIO
naturale di TUTTI i virus
influenzali
Coevoluzione VIRUS-OSPITE
Gli uccelli acquatici sono la
fonte di TUTTI i ceppi
influenzali
Tutte le 16 HA e le 9
NA
Influenza aviaria
• Influenza A
• Gli uccelli selvatici funzionano da
serbatoio
• Gli uomini normalmente non vengono
infettati direttamente
• Gli uccelli domestici sono suscettibili
d’infezione
– HPAI
– LPAI
Influenza aviaria
Proprietà dei virus dell’influenza
aviaria
– Highly Pathogenic Avian Influenza (HPAI) causano
infezioni sistemiche LETALI (gli uccelli muoiono
nel giro di 1 settimana)
– Low Pathogenic Avian Influenza (LPAI) causano
infezioni blande
Suscettibilità
Molte specie di uccelli selvatici e domestici sono
SUSCETTIBILI all’infezione
Virus che sono altamente patogeni in una specie possono non
esserlo in un’altra
Fra le specie domestiche polli e tacchini sono più suscettibili ai
ceppi HPAI
I fattori dell’ospite che rendono un virus HPAI non sono noti
Sono noti i fattori virali (caratteristiche dell’HA)
Trasmissione
• I virus dell’influenza aviaria replicano non solo a
livello dell’apparato respiratorio, ma anche a livello
INTESTINALE
• Sono trasmessi attraverso la via ORO-FECALE
• Sono presenti nelle feci degli uccelli
• Questo vale anche per le infezioni asintomatiche
IN NATURA: il riassortimento di segmenti genici dopo la coinfezione di una cellula da parte di un virus influenzale UMANO e di
uno ANIMALE è responsabile della comparsa di nuovi ceppi
pandemici
TRASMISSIONE VIRALE INTERSPECIE
2 teorie principali, entrambe dimostrate
PASSAGGIO DA UCCELLO DOMESTICO A UOMO
PASSAGGIO ATTRAVERSO UN OSPITE INTERMEDIO (IL
MAIALE)
Riassortimento nell’uomo
Migratory
water birds
1918 H1N1, Tautenberg JK et al,
Nature, October 2005
Riassortimento nel maiale
Migratory
water birds
1957 H2N2, 1968 H3N2
Ceppi pandemici
• TUTTI i ceppi pandemici fino ad ora
analizzati (H1N1, H2N2, H3N2) presentano
un’HA di origine AVIARIA
• Inoltre, l’HA presentava una serie di
caratteristiche che la accomunavano alla
corrispondente HA di ceppi HPAI
Determinanti di paotogenicità nei
virus dell’influenza aviaria
Emoagglutinina (HA)
Meccanismo d’azione dell’HA
1. L’HA (che è una proteina TRIMERICA) media il
legamie del virus ai recettori cellulari che
contengono ACIDO SIALICO.
Dopo il legami il virus è internalizzato per
endocitosi
2. L’ambiente acido dell’endosoma provoca delle
variazioni conformazioniali nel trimero di HA,
mediando la fusione fra envelope virale e la
membrana degli endosomi, che consente il
rilascio della ribonucleoproteina virale al
citoplasma
L’acido sialico
a 2,3
a 2,6
• Zucchero a 9 atomi di C, derivato dalla reazione di acido
fosfenolpiruvico e N-acetil mannosammina
• Si ritrova in Gangliosidi e Glicolipidi della membrana cellulare
• Un composto molto simile è l’etere etilico dell’N-acetil
glucosammina ( ACIDO MURAMICO)
Riconoscimento del recettore
• Le HA dei virus aviari riconoscono
preferenzialmente i recettori contenenti residui di
acido sialico legati al galattosio mediante un legame
2,3 (NeuAc2,3Gal)
• Le HA dei virus umani riconoscono
preferenzialmente i recettori contenenti residui di
acido sialico legati al galattosio mediante un legame
2,6 (NeuAc2,6Gal)
HA e patogenicità
• Riconoscimento del recettore
• Attivazione proteolitica
Riconoscimento del recettore
• I virus aviari riconoscono preferenzialmente i
recettori contenenti residui di acido sialico legati al
galattosio mediante un legame 2,3 (NeuAc2,3Gal)
• I virus umani riconoscono preferenzialmente i
recettori contenenti residui di acido sialico legati al
galattosio mediante un legame 2,6 (NeuAc2,6Gal)
• Residui aa COINVOLTI nel legame sono DIVERSI
Complesso fra recettore
“umano” e HA di un virus
influenzale umano
Complesso fra recettore
“aviario” e HA di un virus
influenzale “aviario”
Attivazione proteolitica
Oltre al riconoscimento del recettore, l’attivazione
proteolitica è essenziale per l’infettività e la
disseminazione dell’infezione
La molecola di HA subisce un taglio post-traduzionale
in due subunità HA1 e HA2 dalle proteasi dell’ospite
il dominio fusogenico all’ N-termine
dell’HA2 media la fusione tra l’envelope virale e la
membrana endosomiale
Ruolo dell’attivazione proteolitica nella
patogenicità
L’Ha dei virus LPAI è tagliata da proteasi specifiche localizzate nel tratto
respiratorio e intestinale
infezioni localizzate e leggere
L’HA dei virus HPAI presenta una serie di aa basici a livello del sito di taglio
(RERRRKKR), che sono brsaglio di proteasi presenti in numerosi organi
infezioni disseminate e letali
L’attivazione proteolica dell’HA è uno dei principali determinanti del
tropismo tissutale dei virus dell’influenza aviaria
Differenze nella distribuzione tissutale delle proteasi e la diversa
suscettibilità dell’HA a questi enzimi può determinare l’outcome
dell’infezione.
CEPPI PANDEMICI
• Un ceppo pandemico si genera quando il virus con la nuova HA
è in grado di passare da uomo a uomo
• Serie di alterazioni che consentono al virus di adattarsi alle
cellule umane
• I recettori cellulari per i virus umani e aviari sono DIVERSI!
• Il maiale contiente nelle cellule della sua trachea entrambe i
recettori (NeuAc2,6Gal; NeuAc2,3Gal) perfetto serbatoio il
riassortimento
Adattamento dell’HA aviaria al
recettore umano
Durante l’adattamento al nuovo ospite il virus cambia in alcuni residui aa
che rendono la proteina aviaria adatta a riconoscere il recettore umano
138A
190E
194L
225G
226Q
228G
Evoluzione di una molecola di HA
(H3)
HA del Virus della Spagnola
Trimero
dell’HA
An expanded view
Punti di contatto
fraF
of the
subdomain
recettore e HA
indicating
differences
between H1 and
H5 subtypes of
HA
Imparando dalle passate
pandemie
Sia H2 che H3 presentavano le sostituzioni:
226Q L and 228G S.
appare essere la più critica
226Q L
H1 mostrava mutazioni alle posizioni 138, 190, 194 and 225
H5N1 isolato nel 1997 NON presentava alcuna di queste mutazioni
Trasmissione diretta all’uomo
La difficoltà dei ceppi di influenza aviaria di infettare
le cellule umane veniva considerata una barriera al
salto di specie
Tuttavia, dal 1997 una serie di virus aviari, a partire
dal ceppo H5N1 si sono trasmessi DIRETTAMENTE
dagli uccelli all’uomo
Anche H1N1 (ceppo pandemico della Spagnola) sembra
essere un virus aviario che ha saltato direttamente
la specie
Influenza aviari dagli uccelli
all’uomo
Migratory
water birds
Domestic birds
• Hong Kong 1997,
H5N1
• HK, China 1999,
H9N2
• Netherlands
2003, H7N7
• Hong Kong 2003,
H5N1
•Viet Nam and
Thailand, 2004
H5N1
Influenza aviaria nell’uomo
• 1997: Hong Kong
– A(H5N1) infected chickens and humans
– 18 hospitalized, 6 died
– 1.5 million chickens culled
• Direct bird to human transmission
• Person-to-person transmission noted, rare (??)
– Requires reassortment or adaptation
Avian Influenza
• 1999: Hong Kong
– A(H9N2)
• 2003: Netherlands
– A(H7N7)
• 2003: Hong Kong / China
– A(H9N2)
• 2003-2004: Thailand, Vietnam
– A(H5N1)
– 22 of 33 cases died
H7N7
•
H7N7, Netherlands, 2003: The Netherlands reported outbreaks of
influenza A (H7N7) in poultry on several farms. Later, infections were
reported among pigs and humans. In total, 89 people were confirmed to have
H7N7 influenza virus infection associated with this poultry outbreak. These
cases occurred mostly among poultry workers. H7N7-associated illness
included 78 cases of conjunctivitis (eye infections) only; 5 cases of
conjunctivitis and influenza-like illnesses with cough, fever, and muscle
aches; 2 cases of influenza-like illness only; and 4 cases that were classified
as “other.” There was one death among the 89 total cases. It occurred in a
veterinarian who visited one of the affected farms and developed acute
respiratory distress syndrome and complications related to H7N7 infection.
The majority of these cases occurred as a result of direct contact with
infected poultry; however, Dutch authorities reported three possible
instances of transmission from poultry workers to family members. Since
then, no other instances of H7N7 infection among humans have been
reported.
11 giugno 2009: è pandemia
• 11 giugno 2009: l’OMS porta a livello 6
H1N1v Marzo 2009: i fatti
• Viene descritto un nuovo sottotipo virale A/H1N1 che si
diffonde da uomo a uomo
• Il sequenziamento dei primi isolati rivela che questo virus
contiene segmenti genomici provenienti da 4 diversi virus
influenzali:
• North American swine
• North American avian
• North American human
• Eurasian swine
H1N1 nei suini: un po’ di storia
• 1930: H1N1 viene isolato per la prima volta nei
suini
stretta correlazione con H1N1
“umano” del 1918
• 1930 - anni ’90: H1N1 continua a circolare nei
suini rimanendo relativamente stabile dal
punto di vista antigenico
•
H1N1
nei
suini:
un
po’
di
storia
1998 (Nord America): si verifica un evento di TRIPLO
riassortimento: H1N1 suino + H3N2 umano + ceppo americano
di virus dell’influenza aviaria (sottotipo non conosciuto)
Triplo riassortante H3N2 (rH3N2) nella popolazione dei suini
del Nord America
• Ulteriore riassortimento con il ceppo H1N1 suino “classico”
Tripli riassortanti suini H1N1 e H1N2
(recentemente isolati anche in Asia)
H1N1 nell’uomo: un po’ di storia
• 1918-1957: H1N1 circola nella popolazione
umana (comparsa di H2N2)
– “antigenic drift” sostanziale
• 1977: H1N1 riemerge nella popolazione
umana
• 1977-2009: continua RAPIDA evoluzione
del virus
Considerazioni
• Gap significativo tra l’HA di H1N1 circolante nei suini e
nell’uomo
Suino come reservoir di virus H1 con potenziale pandemico
• Occasionali isolamenti di virus dell’influenza suina
dall’uomo
Trasmissione uomo-uomo sempre risultata SPORADICA
H1N1v: caratteristiche
Il virus passato all’uomo a Marzo del 2009 contiene una combinazione di
segmenti genici mai precedentemente descritta
• I segmenti genici NA e M derivano dal lineage Euroasiatico dei virus suini
(originariamente aviari)
• I segmenti HA, NP, e NS provengono la lineage classico dei virus suini
(originariamente aviari)
• I segmenti PB2 e PA derivano dal lineage dei virus triplo riassortanti suini
• Il segmento PB1 deriva anch’esso dai triplo riassortanti, ma ha origine
umana (originariamente virus aviaria)
L’ origine di H1N1v
Da Garten RJ and al., Science 10 July 2009: Vol. 325. no. 5937, pp. 197 - 201
Quali eventi di riassortimento abbiano portato
alla genesi dell’attuale H1N1v e dove sia
avvenuto tale riassortimento è al momento
ancora da chiarire
Prevenzione
Chemotherapy
Rimantadine and amantadine block virus entry across the endosome and also interfere with virus
release. They are good prophylactic agents for influenza A, but there are some problems in taking
them on a long term basis. They may be given as protective agents during an outbreak, especially to
those at severe risk and key personnel. They may also be given at the time of vaccination for a few
weeks, until the humoral response has time to develop.
Two neuraminidase inhibitors have recently been approved by the FDA (zanamivir [Relenza] and
oseltamivir). They are active against influenza A and influenza B. These drugs can reduce the
duration of uncomplicated influenza (by approximately 1day). Oseltamavir is approved for prophylaxis
as well as treatment. At the moment, Zanamivir is only approved for treatment but trials indicate it is
probably as effective as oseltamivir in prophylaxis.
As yet there are no clear data on the ability of any of the these drugs to reduce serious
complications when used to treat influenza (as contrasted with when they are used prophylactically).
.
Amantadine, Rimantadine
Uncoating of the influenza virus occurs in low pH endosome or lysosomes, as the result
of a pH-dependent fusogen
Amantadine, Rimantadine are lysosomotropic. They were originally thought to stop
acidification of the endocytic vesicles but it is now thought that they may act on
maturation of virus in trans-Golgi network which is also acidic
These drugs act on maturation of influenza HA glycoprotein so that progeny virus is
poorly infective.
These drugs good for oral prophylaxis against influenza A (but not influenza B). They
are a good alternative to vaccine in immunocompromised patients and the elderly.
Other than this, they are not used much in western countries.
Zanamivir e Tamiflu
The neuraminidase (sialidase) has several functions. It allows the virus to move through mucous
secretions in the respiratory tract so that it may infect new cells. Since sialic acid is the influenza
receptor, it is necessary to remove sialic acid from the surface of the infected cell and of the virus so
that viral particles may escape. The neuraminidase is therefore very important for the spread of the
virus from cell to cell.
Zanamivir (Relenza) is a potent inhibitor of the viral neuraminidase of types A and B influenza viruses.
The design of Zanamivir is based on the three-dimensional structure of the neuraminidase.
Treatment of community-acquired type A and B influenza with Zanamivir shortens the duration of
major symptoms by about one day in the study group as a whole and about three days in the sicker
patients if the drug is started early.
Another neuraminidase inhibitor, Tamiflu (generically called oseltamivir), a carbocyclic sialic acid
analogue can be given orally.
Resistenza ai farmaci
• Tutti gli H1N1v presentano resistenza agli
adamantani (S31N in M2)
• Sono stati descritti solo alcuni ceppi di H1N1v
resistenti all’osaltamivir (mutazioni a livello della
NA, H275Y e I223V )
• Tre ceppi resistenti sono stati identificati in
Italia (uno a Padova)
Scopi della riduzione a livello comunitario
della trasmissione dell’influenza —
mitigazione
•
•
•
•
Ritardare e appiattire il picco epidemico
Ridurre il picco del carico sul sistema sanitario e il pericolo
Ridurre sensibilmente il numero totale dei casi
Guadagnare tempo
Casi
Senza interventi
giornalieri
Con interventi
Giorni dal primo caso
Based on an original graph developed by the US CDC, Atlanta
Perché vaccinarsi contro il virus
influenzale
Per proteggersi se stessi dalla malattia e dalle sue
complicanze, riducendo i costi sociali connessi
Per proteggere la collettività, riducendo le fonti di
infezione e quindi la circolazione interumana del
virus
Campagna per la vaccinazione nei confronti dei virus influenzali
stagionali (2009-2010)
Composizione del vaccino
antinfluenzale
A/Brisbane/59/2007 (H1N1) (presente
anche nel vaccino 2008/09)
A/Brisbane/10/2007 (H3N2) (presente
anche nel vaccino 2008/09)
B/Brisbane/60/2008 (nuova variante)
Effetti collaterali alla vaccinazione
I vaccini antinfluenzali contengono solo VIRUS
INATTIVATI o parti di questi, pertanto NON causano
infezione da virus influenzale
Gli effetti collaterali consistono generalmente in reazioni
locali (dolore, eritema, gonfiore nel sito di iniezione) che
si risolvono in pochi giorni
Le reazioni sistemiche (malessere generale, febbre,
mialgie) sono più rare e durano 1-2 giorni
Sono segnalate reazioni allergiche
L’EMEA (Agenzia europea controllo farmaci)
ha autorizzato l’utilizzo di due vaccini contro
l’influenza A/H1N1 nuova variante (pandemica)
FOCETRIA (Novartis)
PANDEMRIX (GlaxoSmithKline)
Entrambi i vaccini contengono l’Ag HA
ottenuta su uova embrionate di pollo e sono
ADIUVATI
Programma di vaccinazione contro
H1N1v
• I fase
–
–
–
–
Personale sanitario
Copertura
del
40%
Personale dei servizi essenziali
popolazione
Donne neldella
II e III
trimestre di gravidanza
Persone a rischioitaliana
sanitario tra i 6 mesi e i 65
anni
• II fase
– Tutte le persone comprese tra i 6 mesi e i 27
anni di età
The seed virus was grown in Madin–Darby Canine Kidney (MDCK) cell culture by means of standard
processes similar to those used for the development of Optaflu vaccines against interpandemic influenza.
The vaccine was formulated and produced by Novartis (Marburg, Germany) as an inactivated surfaceantigen H1N1 vaccine, with MF59 adjuvant, and supplied in 0.5-ml prefilled single-dose syringes. Each
MF59-adjuvanted vaccine contained 7.5 μg of H1 hemagglutinin, 9.75 mg of the squalene MF59, 1.175 mg
of polysorbate 80, and 1.175 mg of sorbitan trioleate in buffer.
H5N1 virus
In May 1997, an H5N1 virus (A/Hong Kong/156/97) was isolated
from a 3-year-old boy in Hong Kong, who later died of extensive
influenza pneumonia complicated by REYE SYNDROME
By the end of 1997, a total of 18 people had been infected by this
virus, six of whom died
The clinical features of infection included onset of fever and upperrespiratory-tract infection, typical of classical influenza. Some
patients had severe complications, mainly pneumonia,
gastrointestinal manifestations, elevated liver enzymes and renal
failure
Epidemiological studies suggested direct transmission of the virus
from birds and serological evidence of human-to-human
transmission was limited to a few cases, indicating that the virus
had not become fully adapted to its human host
H5N1 virus
• The human H5N1 isolates were not reassortants like the 1957 and 1968
pandemic strains
• All of the viral genes originated from a Eurasian avian virus
• The gene that encodes HA was derived from a H5N1 virus first isolated
from a goose that died in Guangdong Province, China
(A/goose/Guangdong/1/96)
• From 1997 through 2001, H5N1 viruses with an HA of the same genetic
lineage continued to circulate in birds in southeastern China
• In 2002, another H5N1 virus showing ANTIGENIC DRIFT emerged in
Hong Kong and was highly pathogenic in ducks and other aquatic birds, a
property rarely associated with HPAI viruses
• In early 2003, an H5N1 virus infected a family in Hong Kong — the father
and son developed severe respiratory illness and the father died. The
daughter also died
Outbreak of H5N1 influenza
• The most devastating outbreak of influenza associated with H5N1
HPAI viruses occurred in 2003−2004 in Asian countries
• Although not officially reported, such viruses first appeared in July
2003 in poultry in Vietnam, Indonesia and Thailand
• Extensive phylogenetic analysis of the viruses isolated from poultry
revealed multiple genetic reassortants representing multiple
genotypes (A, B, C, D, E, V, W, X0−3, Y, Z and Z+), although each of
the reassortant viruses possessed an HA similar to that of the HA
from the A/goose/Guangdong/1/96 strain
• Similarly, nine different genotypes were identified in domestic-duck
isolates in mainland China (proposed definition: A−I), which were not
related to the genotypes that were described for poultry isolates.
Transmission to human
• Although outbreaks of HPAI viruses were confined to poultry (mainly
chickens) in most countries, there was substantial transmission to
humans, resulting in a total of 53 deaths in three countries
• The clinical presentations of fever, cough, diarrhoea, shortness of
breath, rapid respiratory rate, lymphopaenia and abnormalities on
chest radiography were similar to those noted during the 1997 H5N1
outbreak in Hong Kong
• The mortality rate in this outbreak was significantly higher than in
the 1997 outbreak (54.6% versus 33.3%)
• There was no compelling evidence of human-to-human transmission in
the more recent outbreak, with the exception of a few cases
Avian Influenza
Chickens
Pigs
Vegetable plots
Canal
Human Flu
Avian Flu
Novel Flu
Basi molecolari della virulenza di H5N1
Krug RM, Science 2006
NP
PA, PB1,
PB2
K in pos 627
Sito di taglio
PQ-RERRKKR-G
HA
M1
NA
M2
NS1
C-terminale
ESEWEPEV
Altri fattori di virulenza
?
?
H1N1: caratteristiche virologiche
Le passate pandemie ci hanno insegnato quali sono i
principali determinanti di patogenicità:
• HA con RERRRKKR a livello del sito di taglio proteolitico
• Espressione della proteina PB1-F2 (ORF + 1 in PB1)
• PB2 importante determinante di specie specificità (aa 627)
• Porzione C-terminale di NS1 con il motivo ES/PEV
HA di H1N1v
Priva delle regioni
polibasiche che
determinano la
suscettibilità ad
una ampio
spettro di
proteasi
HA di H1N1v
• Siti di legame al recettore tipici di molti virus suini
classici H1N1, recentemente isolati
– Mutazioni in siti non funzionalmente rilevanti
• Confronta tra H1 di H1N1v e l’H1 dei virus umani
stagionali: mutazioni a livello di putativi siti
antigenici
Da Garten RJ and al., Science 10 July 2009:
Vol. 325. no. 5937, pp. 197 - 201
PB1-F
in
H1N1v
La sequenza presenta un codone di STOP (dopo 12 aa) che
impedisce la traduzione della proteina di fusione
PB2
• Molti virus influenzali aviari sono in grado di
infettare cellule murine ma non sono in grado di
replicare
• aa 627 di PB2 è fondamentale
• Nei virus aviari è acido glutammico, nei virus dei
mammiferi è una lisina
• Importante per la specie-specificità
PB2 in H1N1v
• Tutti i virus H1N1v sequenziati finora
hanno un Glu in posizione 627
– INASPETTATO: tipico dei virus aviari
MERIKELRDLMSQSRTREILTKTTVDHMAIIKKYTSGRQEKNPALRMKWMMAMRYPITADKRIMDMIPER
NEQGQTLWSKTNDAGSDRVMVSPLAVTWWNRNGPTTSTVHYPKVYKTYFEKVERLKHGTFGPVHFRN
QVKIRRRVDTNPGHADLSAKEAQDVIMEVVFPNEVGARILTSESQLAITKEKKEELKDCKIAPLMVAYM
LERELVRKTRFLPVAGGTGSVYIEVLHLTQGTCWEQMYTPGGEVRNDDVDQSLIIAARNIVRRAAVSAD
PLASLLEMCHSTQIGGVRMVDILRQNPTEEQAVDICKAAIGLRISSSFSFGGFTFKRTSGSSVKKEEEVL
TGNLQTLKIRVHEGYEEFTMVGRRATAILRKATRRLIQLIVSGRDEQSIAEAIIVAMVFSQEDCMIKAV
RGDLNFVNRANQRLNPMHQLLRHFQKDAKVLFQNWGIESIDNVMGMIGILPDMTPSTEMSLRGIRVS
KMGVDEYSSTERVVVSIDRFLRVRDQRGNVLLSPEEVSETQGTEKLTITYSSSMMWEINGPESVLVNTY
QWIIRNWEIVKIQWSQDPTMLYNKMEFEPFQSLVPKATRSRYSGFVRTLFQQMRDVLGTFDTVQIIK
LLPFAAAPPEQSRMQFSSLTVNVRGSGLRILVRGNSPVFNYNKATKRLTVLGKDAGALTEDPDEGTSGM
ESAVLRGFLILGKEDKRYGPALSINELSNLAKGEKANVLIGQGDVVLVMKRKRDSSILTDSQTATKRIRM
AIN
NS1
• Manca la sequenza ES/PEV (Stop in
posizione 220)
MDSNTMSSFQVDCFLWHIRKRFADNGLDAPFLDRL
RRDQKSLKGRGNTLGLDIETATLVGKQIVEWILKE
ESSETLRMTIASVPTSRYLSDMTLEEMSRDWFM
LMPRQKIIGPLCVRLDQAVMEKNIVLKANFSVIFN
RLETLILLRAFTEEGAIVGEISPLPSLPGHTYEDVK
NAVGVLIGGLEWNGNTVRVSENIQRFAWRNCDE
NGRPSLPPEQK
Resistenza ai farmaci
• Tutti gli H1N1v presentano resistenza agli
adamantani (S31N in M2)
• Sono stati descritti solo alcuni ceppi di H1N1v
resistenti all’osaltamivir (mutazioni a livello della
NA, H275Y e I223V )
• Finora nessun ceppo resistente è stato
identificato in Italia
H1N1v: caratteristiche antigeniche
• Studi della letteratura dimostrano che H1N1v è
antigenicamente omogeneo e simile ai virus dell’influenza
suina classici e triplo riassortanti che hanno circolato
nella popolazione animale americana negli ultimi 10 anni
• LA VARIABILITA’ ANTIGENICA TRA I VIRUS H1N1v
CIRCOLANTI NELLA POPOLAZIONE UMANA E’ ALLO
STATO ATTUALE INFERIORE A QUELLA OSSERVATA
DURANTE UNA TIPICA STAGIONE INFLUENZALE
H1N1v: caratteristiche antigeniche
• Anticorpi specifici per H1N1v NON sembrano riconoscere H1N1
stagionale (Garten RJ et al. Science. 2009 Jul 10;325(5937):197-201)
• Questo tuttavia NON esclude la possibiltà di cross-protezione nel caso di
co-infezione con i due virus
• Tumpey et al. (Proc. Natl. Acad. Sci. U.S.A. 101, 3166 (2004))
dimostrarono una certa cross-reattività tra sieri ottenuti da individui
vaccinati con il ceppo di influenza umana New Caledonia/20/1999
A(H1N1) e il ceppo A/Swine/Iowa/1930 A(H1N1)
• Il livello di protezione nei confronti di H1N1v in seguito a
vaccinazione con il vaccino dell’nfluenza stagionale rimane
tuttavia da determinare
Conclusioni
• H1N1v possiede una HA antigenicamente e geneticamente diversa da
quella dei ceppi di influenza stagionale
• H1N1v presenta una composizione genetica mai descritta in precedenza
• H1N1v si trasmette efficientemente da uomo a uomo
• Allo stato attuale manca di determinanti di patogenicità tipici dei virus
delle passate pandemie
sintomi blandi
• Il monitoraggio costante delle proprietà atigeniche e genetiche di H1N1v
è mandatorio al fine di rilevare ogni cambiamento che possa avere un
impatto sulla patogenesi, sulla resistenza ai farmaci e sulla validità del
vaccino