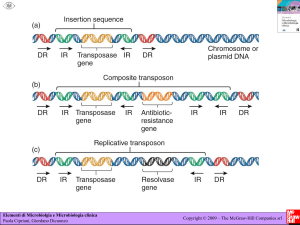
Meccanismi della trasposizione batterica
• per la trasposizione sono necessari gli elementi ripetuti invertititi
intatti alle estremità degli elementi (IR)
• Deve essere presente l’enzima TRASPOSASI
Per la trasposizione sono attivi 2 MECCANISMI
1. CONSERVATIVO : l’elemento è “TAGLIATO” dal
sito originale e si inserisce in un nuovo sito (es.
Tn5 -> elementi della classe I)
2. REPLICATIVO : l’elemento è replicato ed 1
copia rimane nel sito originale, la nuova copia si
integra in un nuovo sito (es. Tn3 ->elementi della
classe II)
Elementi trasponibili negli eucarioti
•Gli ELEMENTI TRASPONIBILI esistono in tutti i tipi di cellule. Quelli
del lievito, della Drosophila e del mais sono stati studiati in dettaglio,
come pure i retrovirus che si trovano nei mammiferi ad essi analoghi.
• Gli ELEMENTI TRASPONIBILI si possono muovere usando intermedi ad
RNA (RETROTRASPOSONI) oppure intermedi a DNA (come nei batteri)
•Alcuni elementi trasponibili possono essere usati come strumenti per il
clonaggio e la manipolazione dei geni (es. gli elementi P di Drosophila
possono essere usati per il trasferimento di geni nella linea germinale di
un moscerino; un altro esempio è dato dal T-DNA dei plasmidi Ti usato
per introdurre geni clonati in piante).
Elementi trasponibili in Drosophila melanogaster
In Drosophila sono presenti circa 30 differenti famiglie di elementi trasponibili,
ognuna presente con molte copie per genoma; costituiscono più del 20-25% del
genoma di Drosophila. Se si considerano anche le sequenze ripetute si arriva al
35% di tutto il genoma
Rubin e Young identificarono una classe di elementi di questo tipo che
chiamarono “copia ”
“ copia ” è una famiglia di elementi presenti in più di 30 copie in differenti
posizioni nel genoma di Drosophila
Alcune famiglie di elementi trasponibili sono simili a copia e quindi si definiscono
“ copia-like ”
Struttura dell’elemento copia
Elemento copia completo (5000 bp)
DTR (direct repeat) 267 bp
IR
(17bp)
IR
(17bp)
DTR (direct repeat) 267 bp
IR
(17bp)
IR
(17bp)
Le INVERTED REPEAT (IR) sono una caratteristica
peculiare di ogni tipo di elemento
La mutazione wapricot in Drosophila è dovuta all’inserimento
di un elemento copia nel gene white
Circa la metà delle mutazioni spontanee in Drosophila sono
causate da TE (Transposable Element)
La principale causa di mutazione spontanea è
l’inserzione di elementi trasponibili
Molte mutazioni spontanee del gene white di
Drosophila derivano dall ’ inserimento di elementi
trasponibili come copia, Doc, pogo, roo ecc. nella
sequenza del gene
Gene white
wch pogo (in Doc)
w1 Doc
wbfroo
whd copia
wa copia
wsp roo
w12 P
Esone 1
2
3
Direzione della trascrizione
4
5
Diversi tipi di elementi trasponibili in Drosophila
Esistono diverse categorie di elementi trasponibili
Elementi che traspongono con intermedi ad RNA denominati:
retrotrasposoni e/o retroposoni
LTR (con lunghi terminali ripetuti)
copia, aurora,mdg3,mdg 4 ed altri
NON-LTR (senza terminali ripetuti)
doc , I, G, F ed altri
Elementi che traspongono con intermedi a DNA
(metodo “cut and paste”) ->P, Bari-1, Mariner, TC1 ed altri
Elementi trasponibili nei Mammiferi
I cromosomi dei Mammiferi hanno una quota variabile tra
il 25-40% di TRASPOSONI che si sono accumulati nel
corso dell’EVOLUZIONE
La maggior parte dei TRASPOSONI dei Mammiferi usa
una trasposizione mediata da RNA (Retrotrasposoni)
Sono di due tipi : LTR (copia-like) e NON-LTR che
rappresenta la classe più numerosa nei Mammiferi
Molti elementi sono correlati ai RETROVIRUS
•I retrovirus sono virus a singolo filamento di RNA
•Il loro meccanismo di replicazione prevede che l’RNA sia copiato in DNA dalla
TRASCRITTASI INVERSA e, dopo la formazione di DNA a doppio filamento,
si integrano nel genoma dell’ospite e si chiamano provirus
Retrotrasposoni virali di specie diverse a confronto
retrovirus
gag
pol
int
env
int
env
pol
int
env
pol
Ty912 (lievito)
gag
copia (Drosophila)
gag
Struttura di classi differenti di elementi
trasponibili nei Mammiferi
Elementi della classe I
RETROTRASPOSONI di tipo LTR o copia-like
gag
pol
5’ LTR
3’ LTR
RETROTRASPOSONI di tipo Non-LTR
LINE (Long Interspersed NON-LTR Element)
ORF 2
ORF 1
AAAAAAA
SINE (Short Interspersed NON-LTR Element)
pol III
AAAAAA
Elementi della classe II
TRASPOSONI a DNA
Gli elementi della classe I (RETROPOSONI)
traspongono mediante un intermedio a RNA che
viene retrotrascritto mediante la
REVERSE
TRASCRITTASI prima di essere integrato in una
nuova posizione nel genoma.
Negli elementi LTR gag codifica una proteina
simile a quella dei capsidi e pol codifica una
“poliproteina” che ha attività di: proteasi, reverse
trascrittasi, RNAsiH e integrasi
Struttura di classi differenti di elementi
trasponibili nei Mammiferi
Elementi della classe I
RETROTRASPOSONI di tipo LTR o copia-like
gag
pol
5’ LTR
3’ LTR
RETROTRASPOSONI di tipo Non-LTR
LINE (Long Interspersed NON-LTR Element)
ORF 2
ORF 1
AAAAAAA
SINE (Short Interspersed NON-LTR Element)
pol III
AAAAAA
Elementi della classe II
TRASPOSONI a DNA
Gli elementi della classe I (RETROPOSONI)
traspongono mediante un intermedio a RNA che
viene retrotrascritto mediante la
REVERSE
TRASCRITTASI prima di essere integrato in una
nuova posizione nel genoma.
Negli elementi LTR gag codifica una proteina
simile a quella dei capsidi e pol codifica una
“poliproteina” che ha attività di: proteasi, reverse
trascrittasi, RNAsiH e integrasi
Gli elementi NON-LTR sono i LINE e SINE.
Gli elementi LINE (i più abbondanti), hanno un
promotore per la polimerasi II e codificano per due
ORF (una con capacità di legame all’RNA e l’altra con
attività di endonucleasi e reverse trascrittasi). Sono
AUTONOMI (L1,L2,L3). Nel genoma umano sono
presenti molte copie tronche di questi elementi,
soprattutto di L1.
Gli elementi SINE hanno un promotore per la
polimerasi III, e non codificano proteine ma dipendono
dall’attività dei LINE, sono quindi NON AUTONOMI.
Un classico esempio di SINE sono le sequenze Alu
(circa 300 bp). Ne sono presenti fino a 500000 copie
nel genoma umano (11%). Sono ripetizioni specifiche
dei Primati, pur se altri Mammiferi hanno sequenze
simili.
Un altro tipo di retrotrasposone NON-AUTONOMO,
che usa l’apparato di L1 per trasporsi nel genoma è
“SVA”:
un
elemento
ripetuto
composito
(SINE+VNTR+ALU)
Struttura di classi differenti di elementi
trasponibili nei Mammiferi
Elementi della classe I
RETROTRASPOSONI di tipo LTR o copia-like
gag
pol
5’ LTR
3’ LTR
RETROTRASPOSONI di tipo Non-LTR
LINE (Long Interspersed NON-LTR Element)
ORF 2
ORF 1
AAAAAAA
SINE (Short Interspersed NON-LTR Element)
pol III
AAAAAA
Elementi della classe II
TRASPOSONI a DNA
Gli elementi della classe I (RETROPOSONI)
traspongono mediante un intermedio a RNA che
viene retrotrascritto mediante la
REVERSE
TRASCRITTASI prima di essere integrato in una
nuova posizione nel genoma.
Negli elementi LTR gag codifica una proteina
simile a quella dei capsidi e pol codifica una
“poliproteina” che ha attività di: proteasi, reverse
trascrittasi, RNAsiH e integrasi.
Gli elementi NON-LTR sono i LINE e SINE.
Gli elementi LINE (i più abbondanti), hanno un
promotore per la polimerasi II e codificano per
due ORF (una con capacità di legame all’RNA e
l ’ altra con attività di endonucleasi e reverse
trascrittasi). Sono AUTONOMI (L1,L2,L3). Sono
presenti molte copie tronche soprattutto di L1.
Gli elementi SINE hanno un promotore per la
polimerasi III, e non codificano proteine ma
dipendono dall ’ attività dei LINE, NON
AUTONOMI.
Un classico esempio di SINE sono le sequenze Alu
(circa 300 bp). Ne sono presenti fino a 500000
copie nel genoma umano (11%). Sono ripetizioni
specifiche dei Primati, pur se altri Mammiferi
hanno sequenze simili.
Un altro tipo di retrotrasposone NONAUTONOMO, che usa l’apparato di L1 per
trasporsi nel genoma è “SVA”: un elemento
ripetuto composito (SINE+VNTR+ALU)
Gli elementi della classe II (TRASPOSONI) traspongono mediante un intermedio a DNA ed una TRASPOSASI e si
muovono con un meccanismo di “ taglia e incolla”
Retrotrasposoni virali dei Mammiferi
Trasposone LTR “copia-like”
LTR
5,8 kb
Sequenza codificante per proteine
LTR
Ripetizione diretta
lunga 5-10 bp
Trasposoni retrovirali NON-LTR sono:
SINE (Short Interspersed Non-LTR Element)
LINE (Long
Interspersed Non-LTR Element)
Elementi trasponibili che si trovano nel gene umano (HGO)
Il gene HGO, codifica per l’omogentisato 1,2 -diossigeneasi, la cui mancanza determina l’ALCAPTONURIA
10 kb
1
23
4
5
6 7 8 9 10
11
12
ESONI
NON-LTR
Sequenze LINE
Sequenze SINE
Sequenze Alu (ripetizioni di 300 bp)
LTR
13
14
Ruolo degli elementi trasponibili
•Elementi trasponibili specifici sono localizzati, in alcune specie, a
livello dei centromeri (grano, riso, ecc.)
•In alcune specie i centromeri sono costituiti da ripetizioni che possono
essersi originate da riarrangiamenti di elementi trasponibili come le
sequenze alfoidi nei centromeri umani, ma anche ripetizioni in
Arabidopsis, Drosophila, balena ecc.
•In Drosophila i telomeri sono costituiti da elementi trasponibili
(TART, HetA e TAHRE)
- TART è un elemento AUTONOMO di 10654 bp
- Het A è un elemento NON AUTONOMO di 6083 bp, è localizzato solo nell’eterocromatina
-TAHRE (Telomeric Autonomous HetA-Related Element) è un elemento autonomo di 10463 bp
Gli elementi trasponibili sono finemente regolati. Nel corso
dell ’ evoluzione si sono evoluti dei meccanismi di
SILENZIAMENTO di questi trasposoni. Se questi meccanismi di
silenziamento non funzionano ci possono essere effetti anche
gravi nell’organismo!
RNA interferenza