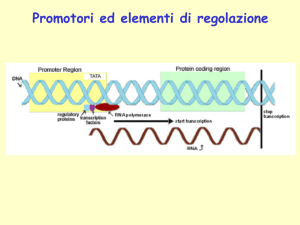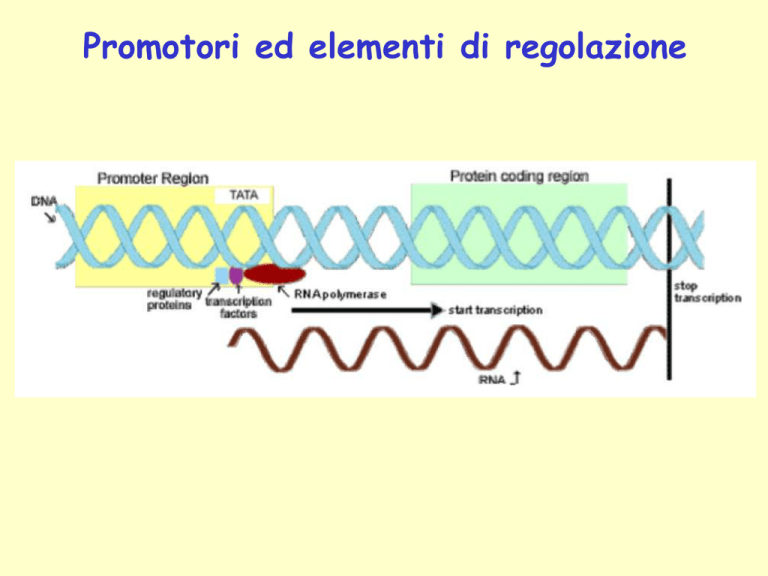
Promotori ed elementi di regolazione
Elementi cis- e trans- per la regolazione dell’espressione genica
Cis-acting elements – Sequenze di DNA nella vicinanza
della regione codificante di un gene che sono necessarie
per l’espressione genica
Trans-acting factors – fattori, di solito proteine, che
si legano agli elementi cis- per il controllo
dell’espressione genica
Struttura e organizzazione di un gene eucariotico
Elementi comuni presenti in quasi tutti i promotori:
CAAT box. Una sequenza consenso vicino a -80 bp dal punto di inizio della
trascrizione (+1). Aumenta la forza del promotore; sembra funzionare in
entrambi gli orientamenti. Nelle piante questo box è rimpiazzato da una
sequnza consenso chiamata AGGA box. E’ il sito di legame del fattore di
trascrizione CTF/NF1; Muller, et al. 1988. Eur. J. Biochem. 176:485
TATA box. Sequenza di solito localizzata intorno a 25 bp a monte del sito di
inizio della trascrizione. In genere tende ad essere circondata da sequenze
ricche in GC. E’ il sito di legame per l’RNA polimerasi II e una serie di fattori
di trascrizione (TFIIX, dove X è una letetra che indica uno specifico fattore
di trascrizione) che formano il complesso di inizio; il primo di questi è il
TFIID; Workman & Roeder, 1987. Cell 51:613
GC box. Un sequenza ricca in G e C, di solito è presente in copie multiple ,
normalmente intorno al TATA Box. E’ il sito di legame del fattore di
trascrizione SP1; Anderson & Freytag, 1991. Mol. Cel. Biol. 11:1935
CAP site. Sequenza di inizio della trascrizione definito +1, in cui il processo
di trascrizione inizia.
Struttura del
complesso di
trascrizione dell’RNA
polimerasi II e
sequenza di
assemblaggio
Le tre principali categorie dei fattori di trascrizione
Gli attivatori modulano a distanza la regolazione genica
Introni, attivatori (Enhancer) e regioni di attacco
(Attached regions)
Queste regioni contribuiscono a regolare l’espressione genica.
Possono costituire delle porzioni aggiuntive di DNA nel costrutto da
utilizzare nella trasformazione genetica di tessuti vegetali per
aumentare, modulare o stabilizzare l’espressione genica.
Ad esempio le regioni MAR o SAR (Sito di attacco alla matrice, Matrix
attachment regions o sito di attacco all’impalcatura, Scaffold
attachment Regions) vengono utilizzate per stabilizzare l’espressione
genica soprattutto nel caso di trasformazioni genetiche effettuate con
il metodo biolistico, dove il numero di copie può essere elevato.
MAR o SAR: Regione del DNA che si attacca alla matrice nucleare.
Matrice nucleare: Rete di fibre che circonda il nucleo e penetra al suo
interno.
Nuclear scaffold e MAR
Regioni MAR (regioni di attacco alla matrice) possono regolare
l’espressione genica
Identificazione e analisi funzionale degli
elementi cis- e dei promotori
Identificazione di elementi promotori funzionali
mediante analisi footprinting
Footprinting:
es. Determinazione
del sito di legame
del repressore lac
Espressione con i geni reporter
•GUS
•GFP
•Luciferasi
topo
foglia
Trasformazione stabile e trasformazione transiente
Trasformazione stabile
Trasformazione transiente
•Metodo Biologico
•Metodo Fisico
Saggio GUS (b-glucuronidasi)
•Si esegue 2-3 giorni dopo la
trasformazione del tessuto
•Il tessuto sottoposto a trasformazione
viene immerso per 16-24 ore in una
soluzione contenente 5-Bromo-4-Cloro-3indolil-b-D-acido glucuronico (X-gluc).
•L’eventuale attività enzimatica viene
evidenziata nel tessuto intatto come delle
macchie blu.
Vantaggi:
• Semplicità di esecuzione e non necessità di
apparecchiature particolari.
• Può essere anche quantitativo.
• In tal caso si quantifica l’attività GUS mediante saggio
fluorometrico usando come substrato il 4metilumbelliferil b-D-glucuronide (MUG). Questa analisi
implica l’estrazione dell’enzima e la successiva analisi
dell’attività GUS.
Comunque il vantaggio principale risiede nella semplicità
del saggio istochimico.
Svantaggi:
La colorazione istochimica causa la morte del tessuto.
Saggio GFP
E’ forse il gene reporter attualmente più
utilizzato nelle analisi transienti in pianta.
La GFP è una proteina di 238 amminoacidi della
medusa Aequorea victoria.
I componenti richiesti per la bioluminescenza dell’equorea
victoria includono:
•
Equorina, una fotoproteina attivata dal Ca++ che
emette luce blu-verde
•Green Fluorescent Protein (GFP), una proteina
fluorescente verde, che accetta energia
dall’equorina e la riemette come luce verde.
La fluorescenza intrinseca della GFP è dovuta a 3
amminoacidi (Ser-65-Tyr-66-Gly-67) che
ciclizzano formando un cromoforo che ha due
massimi di eccitazione:
•con luce UV (396 nm)
•luce blu (475 nm).
I massimi di emissione della
luce sono rispettivamente a
508 e 503 nm.
Sono state prodotte diverse versioni modificate
della GFP in modo da renderla più adatta come
reporter.
Una delle modificazioni più efficaci ha riguardato
la sostituzione della Ser-65 con una Thr.
La GFP’S65T’ possiede:
•un solo picco massimo di eccitazione alla luce blu
•un più forte segnale fluorescente rispetto alla
proteina nativa, rendendo più semplice il suo
rilevamento.
Cos-7 cell with GFP-tubulin (projection of 5 confocal
sections through the cell).
ALTRI GENI REPORTER
Saggio Luciferasi
•Utilizza la luciferina 4-monoossigenasi che ossidando
la luciferina produce una luce giallo-verde (560 nm)
Luciferina
Ossiluciferina
+ luce
•La reazione avviene in presenza di ATP e produce
AMP, biossido di carbonio e luce.
•La luce prodotta viene misurata utilizzando un luminometro
•Quando la luciferina è presente in eccesso, la quantità di
luce emessa è proporzionale alla quantità di enzima.
•Il gene della luciferasi deriva dalla lucciola (Photinus pyralis)
Luciferase gene expression
Firefly Luciferase is a nearly ideal reporter gene for plant cells. The first luciferase gene cloned was
from the North American firefly Photinus pyralis. The active enzyme is a single polypeptide with a
mass of 62kDa. The luciferase reaction emits yellow-green light (560nm) and requires only the
enzyme, ATP, Mg2+, O2 and the substrate luciferin. In fireflies, the luciferase reaction occurs in the
peroxisomes of a specialized light organ, but the reaction can also occur in bacteria, cell-free extracts
and in plant tissues. Most frequently the detection of the luciferase activity is performed in a photon
measuring device such as a luminometer or a scintillation counter. With DIANA III and LAS cooled
CCD camera systems, the luciferase expression can be detected and measured more elegantly in situ.
Saggio CAT
Questo saggio usa l’enzima batterico
cloramfenicolo acetiltrasferasi (Cat).
E’ particolarmente usato nei sistemi di
mammifero perché le cellule dei mammiferi non
possiedono questo enzima.
Un test usato per rilevare l’attività CAT consiste
nell’usare come substrato [ 14C]acetil-CoA.
Le forme acetilate vengono separate mediante
cromatografia su strato sottile e il segnale
identificato mediante autoradiogarfia.
Saggi transienti per l’espressione genica
Geni Reporter- sommario
I geni reporter sono geni che codificano per proteine
facilmente saggiabili.
Sono utilizzati da soli o in combinazione con altre regioni
codificanti il cui prodotto proteico è difficile da saggiare
I più comuni geni reporter sono:
GFP (green fluorescent protein): Emissione fluorescente
dopo eccitazione (rilevamento in situ mediante i sistemi
DIANA, LAS o FLA).
GUS (b-glucuronidasi): idrolizza glucuronodi incolori
producendo un prodotto colorato.
•LUC (luciferasi): Ossida la luciferina, emettendo fotoni a
560 nm. (rilevamento in situ mediante DIANA or LASsystems)
•CAT (cloramfenicolo acetiltransferasi): trasferisce il
gruppo acetile radioattivo al cloramfenicolo; rilevamento
mediante cromatografia su strato sottile e autoradiografia
(sistema Fuji BAS o FLA)
•GAL (b-galattosidasi): idrolizza galattosidi incolori
producendo un prodotto colorato
Utilizzo dei geni reporter per:
•Attività regolativa di una sequenza di DNA
-1
-2000
GUS
-1500
GUS
-800
GUS
-300
GUS
Utilizzo dei geni reporter per:
•Determinazione della ‘Forza’ dei Promotori
Attività di alcuni promotori in saggi transienti con CAT e GFP
Utilizzo dei geni reporter per:
•Determinazione della tessuto-specificità
Es: Espressione nello stigma e nel tessuto vascolare del gene Pvpgip1
Jones et al. 2006 Options mediterraneennes 81
Utilizzo dei geni reporter per:
•Scelta del tessuto per la trasformazione
•Embrioni immaturi
•Dischi fogliari
•Meristemi apicali
Utilizzo dei geni reporter per:
•Scelta delle condizioni per la trasformazione
Costrutti per verificare l’influenza dell’elemento a –160
sull’espressione temporale e spaziale del gene: uso dei geni
reporter GUS e GFP
Piante transgeniche di A. thaliana per verificare l’influenza
dell’elemento a –160 sull’espressione del gene GUS
Tipi di promotori
•Promotori costitutivi
•Promotori tessuto specifici o stadio di sviluppo specifici
•Promotori inducibili
•Promotori sintetici
Promotori costitutivi
Promotore 35S del Virus del Mosaico del Cavolfiore
(CaMV 35S)
•Subdomain A: -46 10 +1 corrisponde al promotore minimo
•Subdomain B: -208 to -46 corrisponde ad una regione enhancer
•Può essere usato nella combinazione 35S duplicato, per una
maggiore espressione
Promotore dell’Ubiquitina di mais (Ubi-1)
-214 -204
Promotori tessuto-specifici
Esempi di promotori tessuto specifici brevettati
Es: Dx5-Promotore delle glutenine ad alto peso molecolare
Promotori inducibili
Sono indotti in seguito alla presenza o assenza di specifici
fattori biotici o abiotici
I geni sotto il controllo di questi promotori possono essere
attivati o disattivati a specifici stadi di sviluppo o in
specifici tessuti
Esempio di promotori indotti da fattori chimici o fisici
•Promotori regolati da fattori chimici: alcool,
tetraciclina, steroidi, metalli ed altri composti
•Promotori regolati da fattori fisici: luce e
temperatura
Physically-regulated promoters
Chemically-regulated promoters
Caratteristiche ideali di un sistema chimico-inducibile in pianta
• Livello di espressione costitutivo basso
• Alta inducibilità
• Elevata specificità rispetto all’induttore
• Elevata variabilità di risposta rispetto alle concentrazioni dell’induttore
• Risposta rapida dopo l’induzione
• Rapido silenziamento in seguito alla rimozione dell’induttore
• Induttore non tossico e privo di effetti fisiologici sulla pianta
• Induttore assente nelle piante bersaglio
Sistema inducibile basato sul desametasone
Sistema di espressione indotto dall’etanolo
Pathogenesis-related gene promoters
Promotori sintetici
Esempio di applicazione di specifici promotori
Tecnologia ‘Terminator
Questa tecnologia consiste nell’uso di una serie di
‘interrutori’ molecolari che possono essere attivati nelle
piante transgeniche per uccidere l’embrione e impedire
così la germinazione dei semi transgenici.
Molecole coinvolte
Gene I: Ricombinasi
Enzima che riconosce un tratto specifico di DNA lo
rimuove e riunisce le due estremità di taglio
Gene II: Inibitore della ricombinasi sotto il controllo di
un promotore reprimibile
Gene III: Tossina
La proteina codificata da questo gene è letale per
l’embrione ed è sotto il controllo di un promotore tardivo
(late promoter, LP) che è attivo soltanto nelle fasi finali
dello sviluppo dell’embrione
‘Blocker’: segmento di DNA posto tra il promotore e
la regione codificante della tossina composto da
sequenze riconosciute dalla ricombinasi.
La presenza del ‘blocker’ interferisce con la capacità
del promotore di regolare l’espressione del gene per
la tossina
Induttore: Sostanza con la quale la Ditta Sementiera
tratta i semi transgenici
Gene Ricombinasi
Gene Inibitore
Promotore reprimibile
Promotore tardivo
Blocker
Gene Letale
Meccanismo di funzionamento
Gene Ricombinasi
Promotore trardivo
Gene Ricombinasi
Gene Inibitore
Blocker
Gene Letale
Nei semi
non
trattati
con
l’induttore
Gene Inibitore
Gene Letale
Tossina
Nei semi
trattatti
con
l’induttore
Promotori maggiormente usati
nella trasformazione di piante
•Promotore 35S dal virus del mosaico del cavolfiore
(CaMV)
•nos 5’: regione regolatrice del gene della nopalina
sintetasi (NOS) dal T-DNA di Agrobacterium
tumefaciens
•Promotore dell’ ubiquitina di mais (Ubi )
•Promotore endosperma specifico: gluteline riso e
glutenine frumento