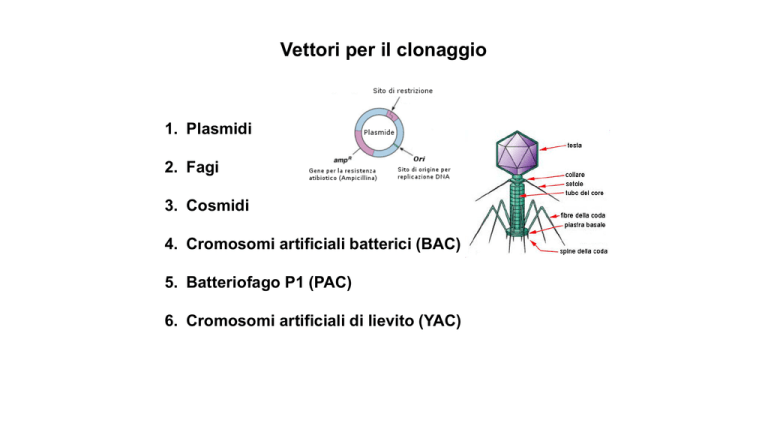
Vettori per il clonaggio
1. Plasmidi
2. Fagi
3. Cosmidi
4. Cromosomi artificiali batterici (BAC)
5. Batteriofago P1 (PAC)
6. Cromosomi artificiali di lievito (YAC)
Vettori e dimensioni degli inserti clonabili
Plasmidi
FUNZIONI ASSOCIATE
AI GENI PLASMIDICI
o Rappresentano in natura una forma di DNA parassita
RESISTENZA AGLI ANTIBIOTICI
E. coli; Salmonella; Sigella
o Talvolta possono passare da una cellula batterica all’altra (per mitosi e
coniugazione batterica)
RESISTENZA AI METALLI PESANTI
E. coli; Staphylococcus aureus; Pseudomonas
o Sono in grado di replicarsi all’interno dell’ospite
o La resistenza agli antibiotici non è l‘unica proprietà che conferiscono ai
batteri
• la resistenza a farmaci, ai metalli pesanti, ai raggi UV
• l'utilizzo di fonti di carbonio insolite o la fissazione di azoto
inorganico nel suolo
• la produzione di proteine in grado di uccidere gli altri batteri
(tossine)
• la virulenza (plasmide T di Agrobacterium tumefaciens)
o Sono presenti in copie multiple nella cellula batterica (cellula ospite)
o EcolE1: presente nel numero di circa 15 copie nel batterio E. coli wildtype
o molte centinaia di copie sono invece generate per i vettori
ingegnerizzati
METABOLIZZAZIONE DI LATTOSIO,
SACCAROSIO
Enterobatteri
PRODUZIONE DI BATTERIOCINE
E. coli; Batteri lattici
FATTORI DI VIRULENZA
Staphylococcus aureus
PRODUZIONE DI ANTIBIOTICI
Streptomyces
FISSAZIONE AZOTO
Rhyzobium
PRODUZIONE DI ENTEROTOSSINE
E. coli; Enterotossici
SINTESI DI DESTRANO
Streptococcus mutans
INDUZIONE DI TUMORI NELLE PANTE
Agrobacterium tumefaciens
DEGRADAZIONE DI IDROCARBURI
Pseudomonas
Plasmide ColE1
La maggior parte dei plasmidi più frequentemente usati
nelle tecnologie ricombinanti deriva dal plasmide ColE1,
presente naturalmente nei batteri di E. coli.
Il nome deriva dal fatto che contiene un gene per colicin E1
(gene CEA). Le colicine sono tossine che vengono rilasciati
nell'ambiente per ridurre la concorrenza di altri ceppi batterici.
Il plasmide possiede anche informazioni genetiche per
l'immunità derivanti dal gene imm.
Inoltre, ha una serie di geni per la mobilità (mob).
Le sue dimensioni e la presenza di un singolo sito di
riconoscimento EcoRI, lo hanno candidato ad essere
considerato un vettore per il clonaggio.
Tuttavia possiede un limitato numero di marcatori genetici per
la selezione dei cloni trasformati.
pBR322 il capostipite dei vettori plasmidici
pBR322: derivato dai plasmidi RSF2124 e pSC101 (AmpR e TetR)
e pMB1 (ori)
Sequenziamento e caratterizzazione di tutti i siti di restrizione
(Watson 1988): 6 nel gene AmpR e 13 nel gene TetR (clonaggio e
piastramento in replica)
AmpS TetR
AmpR TetR
Tet+
PstI ~CTGCAG~
~GACGTC~
Utile sito di RE per
clonaggio di code dG-dC
con Terminal
Deoxynucleotidyl
Transferase
pBR322, non solo clonaggio:
analisi della funzione di promotore di frammenti di DNA
HindIII è posizionato nel promotore della resistenza alla
Tetraciclina:
Se le sequenze clonate hanno attività promotrice-simile,
non si avrà inattivazione inserzionale del gene tet, per la
sintesi dell’enzima che produce resistenza alla tetraciclina.
+amp
+tet
Clonaggio frammenti troppo grandi di DNA: instabilità
plasmidica
Widera et al. 1978
HindIII
Vettori derivati dal pBR322
EcoRI
Inserzione del gene
per la resistenza al
Cloramfenicolo CmR
Gene reporter cloramfenicolo acetil transferasi (cat)
-15
-50
Il gene cat è posto sotto l’espressione di un promotore
minimale (TATAbox) per l’analisi di sequenze regolative
della trascrizione.
Cat conferisce resistenza all’antibiotico cloramfenicolo
(marcato con C14), che, aggiunto agli estratti cellulari,
viene modificato dall’enzima cloranfenicolo acetil
transferasi per acetilazione coi gruppi acetilici
dell’acetil-CoA (un cofattore essenziale della reazione).
Le molecole mono- e di-acetilate derivate dall’attività di
cat sono analizzate per cromatografia su strato sottile
(lastre TLC di silice) esposta a lastra autoradiografica.
di-acetylated
chloramphenicol
mono-acetylated
chloramphenicol
unacetylated
chloramphenicol
pl. Upstream sequences
Reazione della cloramfenicolo acetil transferasi
acetil-CoA + cloramfenicolo14 ⇄ CoA + cloramfenicolo 3-acetato
+
⇄
cloramfenicolo
+ 3-acetato
Vettori con il gene reporter Luciferasi
per lo studio di sequenze ad attività promotrice
Vettori sviluppati con MCS: i pUC
Batteriofago λ
E’ un virus che infetta E. coli.
Il suo genoma è una molecola di DNA lineare a
doppio filamento di 48.5 kb.
le estremità presentano un’estensione a singolo
filamento di 12 bp complementari (sito cos), che ne
consentono la circolarizzazione una volta entrato
nell’ospite.
Le sequenze responsabili della Ricombinazione (att)
Geni funzionalmente correlati sono raggruppati in
regioni distinte cI (regolatore negativo), N e Q
(regolatori positivi):
Regolanti il passaggio dal ciclo lisogenico al
ciclo litico N, cl (cII e cIII) e cro
Sintesi del DNA (O, P)
e i geni che codificano per proteine della Testa (Head)
e Coda (Tail):
Funzioni tardive (Q)
Lisi dell’ospite (S, R)
Ciclo lisogenico e ciclo litico del fago λ
Il DNA circolare del fago λ viene
duplicato in questa fase come
plasmidi (replicazione theta). Poi
passa ad un modello di rolling
circle e, infine, avviene il
packaging
1
2
8
3
Convivenza più o meno
stabile con l’ospite, DNA
fago λ duplicato insieme al
DNA cromosomico batterico;
altri bateriofagi non si
integrano come il P1 e in
questa fase si duplicano
come plasmidi
7
6
5
4
Replicazione del fago λ
Quando il fago λ infetta il batterio le estremità coesive si appaiano formando una struttura circolare (il sito cos è sensibile alla temperatura, che le
denatura) e i nicks sono riparati dalla ligasi batterica.
L'integrazione del genoma fagico all'interno di quello batterico avviene presso una speciale regione. La sequenza precisa sul genoma di E. coli è
chiamata attB (dall'inglese Bacterial attachment, sito di attacco batterico) ed è composta essenzialmente di tre segmenti, detti B-O-B'. La
sequenza omologa sul genoma del fago è attP (dall'inglese Phagic attachment, sito di attacco fagico) ed è composta delle regioni P-O-P'.
Il sito di attacco sul DNA del fago λ (attP) possiede solo 15 bp completamente omologhe al sito batterico (attB). La reazione operata dalla
integrasi (INT, ricombinasi di λ) genera due nuovi siti di attacco, che fiancheggiano il DNA fagico inegrato (attR e attL).
La proteina XIS (codificata dal fago) insieme alle proteine batteriche FIS e IHF procedono invece alla rimozione del DNA del fago λ.
Replicazione del fago λ
Il DNA circolare del fago λ viene duplicato in questa
fase come plasmidi (replicazione theta).
Poi passa ad un modello di rolling circle generando
una molecola di DNA lineare contenente un gran
numero di copie genomiche continue. In questa
modalità il fago si trova nel ciclo litico.
Replicazione theta del fago λ
https://www.youtube.com/watch?v=2
THQtKHkNhQ
Replicazione rolling circle del fago λ
https://www.youtube.com/watch?v=gmEj0ugnVWA
Packaging del fago λ
Il DNA lineare contenente un gran numero di copie genomiche
continue del fago λ viene tagliato producendo le estremità cos.
Le proteine sono assemblate in due strutture separate: testa (vuota,
dove sarà poi inserito il DNA) e coda.
Quindi il DNA viene impacchettato nella testa e poi viene inserita la
coda.
Clonando un DNA esogeno nel fago λ (49 kb) le sue dimensioni
aumenterebbero, poiché la testa può contenere massimo 51 kb, sarà
quindi necessario eliminare del DNA fagico, es. i geni del ciclo
lisogenico (25%), ma per mantenerne la stabilità non si possono
eliminare tutti i geni. Il 75% del DNA fagico dovrà essere conservato,
altrimenti non saranno prodotte le placche di lisi.
Importante: nel clonaggio considerare bene i limiti del packaging!!
Packaging in vitro: molto efficiente
Efficienza di trasformazione del
DNA di λ con un inserto clonato:
104-103 placche/μg di DNA
→Impaccamento in
placche/μg di DNA
vitro
106
Fagi λ mutanti in ceppi batterici adatti producono teste vuote
Fagi λ mutanti in ceppi
batterici adatti producono solo
proteine della coda e del
packaging
Clonaggio in fago λ
(geni del ciclo lisogenico)
Crescita del fago λ
Trasformazione batterica con plasmidi vs. trasfezione con fago λ
Colonie
su
agar selettivo
Placche di lisi
su
tappeto batterico cresciuto su soft agar
Processo meno efficiente, ma consente il clonaggio di
frammenti di dimensioni maggiori
Vettore λ gt10
braccio sinistro
braccio destro
estremità cos
Ha
dimensioni
ridotte
(43,3
kb)
estremità cos
consentendo l’inserimento di 7,6 kb
Estremità cos appaiabili a 37°C
Sito unico di taglio EcoRI
Clonando
in
EcoRI
si
produce
un’inattivazione inserzionale sul gene cI,
generando
funzionante,
quindi
un
repressore
non
si
avrà
il
non
ciclo
lisogenico, ma subito quello litico e la
produzione di placche di lisi trasparenti
(mentre i fagi senza inserto produrranno
placche torbide)
Vettore λ gt11
Dimensioni vettore 37 kb (51 kb-37 kb), clonabili 14 kb
Contiene il gene per la β-galattosidasi (lacZ)
Sito di taglio EcoRI e clonaggio in questo caso è al 3’ della βgalattosidasi (mentre nel plasmide pUC18 era al 5’): clonando in
EcoRI si producono placche di lisi bianche su terreno X-gal.
Inoltre, se il gene esogeno sarà inserito nello stesso frame di
lettura, produrrà una proteina di fusione contenente la βgalattosidasi fusa col prodotto clonato ed utilizzabile come target di
anticorpi anti-β-galattosidasi per lo screening delle placche
ricombinanti).
Vettori λ di sostituzione EMBL4
Sono stati sviluppati vettori di sostituzione:
In questo vettore si possono eliminare tutti i geni
non essenziali, ma buona parte del DNA fagico
dovrà essere conservato nelle sue dimensioni
totali per la stabilità virale e la produzione delle
placche di lisi,
Usato un enzima di restrizione con due siti di
taglio (BamHI), che taglia un frammento stuffer
(14 kb). DNA digerito, va corso su gel e purificati i
due bracci digeriti BamHI ciascuno contenente le
estremità fagiche cos.
estremità cos
estremità cos
Vettori λ di sostituzione EMBL4
Dopo il clonaggio in BamHI, il vettore è ricircolarizzabile alle
estremità cos.
Dimensioni braccia 29 kb, clonabili 22 Kb.
Senza inserto, il vettore sarà troppo piccolo per il
packaging, non produrrà placche di lisi, in quelle
prodotte ci saranno solo fagi ricombinanti. Non sarà
necessario defosforilare il DNA fagico prima della
reazione di ligasi
l’inserto di DNA da clonare viene invece defosforilando
si eviteranno i multimeri e si otterranno così solo fagi
con 1 copia del gene (ottimale per la produzione di
librerie geniche).
Inoltre, il frammento stuffer può essere sostituito non
necessariamente solo con DNA esogeno (da clonare), ma
si può associare anche all’inserzione di gene reporter (βgalattosidasi;
cloramfenicoloacetil
luciferasi, Green Fluorescent Protein).
transferasi,
BamHI
Vettori derivati dal fago λ: batteriofagi filamentosi M13
M13 è costituito da DNA a singolo filamento ed è maschio-specifico perché penetra nella cellula attraverso il pilus dell’ospite.
Usato in passato per produrre cloni di DNA a singolo filamento per il sequenziamento sfruttando il ciclo di replicazione di questo virus
DNA
fagico
a
singolo
Replicazione
prima
del
doppio
filamento entra nel batterio
filamento, poi con la trascrizione e
dal pilo F
produzione
delle
proteine
fagiche,
l’aumento della proteina del capside
(legante il DNA) inibisce
dell’emi-elica
la sintesi
complementare,
replicazione
diviene
asimmetrica a singolo filamento
1000 fagi rilasciati ad ogni ciclo replicativo, fuoriescono dalla membrana batterica
la
così
Vantaggi dei vettori a singolo filamento
Clonaggio non limitato dall’impaccamento, aumento delle dimensioni dell’inserto (teoricamente, fino a 6
volte quelle dell’M13 wild type)
Nella forma replicativa a doppio filamento può essere manipolato come un plasmide
Il vettore nella sua forma a singolo filamento può essere usato nel clonaggio per:
Sequenziamento diretto sul singolo filamento
Produzione di DNA mutato: mutagenesi sito-specifica mediante oligonucleotidi
Produzione di sonde d’ibridazione a singola elica
Vettori derivati dal fago λ: batteriofagi filamentosi M13
Impiegati nel phage display, esibizione di una proteina clonata in M13
Esponendola sulla superficie del batteriofago, facile screening dei ricombinanti.
Fab
Fab
Fab (fragment antigen binding) library
Phage display è una tecnica di laboratorio per lo studio delle
interazioni proteina–proteina, proteina–peptide e proteina–
DNA che usa dei batteriofagi per collegare le proteine con le
informazioni geniche che codificano per esse. Con questa
tecnica il gene codificante per la proteina d'interesse è
inserito nel gene di una proteina di rivestimento del fago
causando l'esposizione della proteina sull'esterno del
fago, instaurando una connessione tra genotipo e fenotipo.
Questi fagi recanti la proteina d'interesse possono quindi
essere sottoposti a screening con altre proteine (es.
anticorpi), peptidi o sequenze di DNA con l'obiettivo di
individuarne possibili interazioni. In questo modo una vasta
quantità di proteine può essere saggiata e amplificata in un
processo di selezione in vitro analogo alla selezione
naturale.
I batteriofagi più usati per il phage display sono il fago M13 e
il fago filamentoso fd, sebbene siano stati usati anche fagi
T4 e lambda.
Fab
Geni per gli anticorpi

![Lezione 15 Virus [modalità compatibilità]](http://s1.studylibit.com/store/data/000771737_1-84b1cca561c5813066d1b76125338a98-300x300.png)


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)





![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)
