PERCHE’ I BATTERI HANNO
SUCCESSO
• VERSATILITA’ METABOLICA
• VELOCITA’ DI ADATTAMENTO ALLE VARIAZIONI
AMBIENTALI
Livia Leoni
Università Roma Tre
Dipartimento Biologia
Laboratorio di Biotecnologie Microbiche
Stanza 2.3
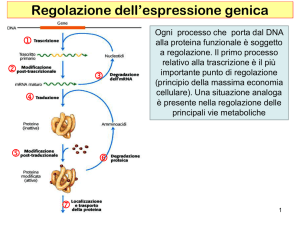
Regolazione dell’espressione genica
trascrizione
DNA
Regolazione a
livello della sintesi
di RNA
traduzione
RNA
Regolazione Posttrascrizionale
(stabilità dell’mRNA e delle
proteine)
PROTEINE
Regolazione
della attività
enzimatica
INDUZIONE
di una via catabolica
REPRESSIONE
di una via anabolica
Regolazione dell’espressione genica
trascrizione
DNA
Regolazione a
livello della sintesi
di RNA
traduzione
RNA
PROTEINE
Regolazione Posttrascrizionale
(stabilità dell’mRNA e delle
proteine)
Inizio della trascrizione
σ
Core RNAP
a) Legame RNA polimerasi
a)
b) Riconoscimento promotore
b)
c)
c) Formazione complesso
aperto
d)
d) Inizi abortivi
σ
e)
mRNA
e) Elongazione
L’RNA puo’ formare strutture secondarie che possono avere varie
funzioni
TERMINAZIONE DELLA TRASCRIZIONE rho-indipendente
Livello 1 : Il fattore sigma determina il riconoscimento specifico del
promotore. Il fattore sigma 70 serve a trascrivere la maggioranza dei geni,
ma esistono anche fattori sigma alternativi che servono ad esprimere
determinati gruppi di geni in risposta a specifici stimoli ambientali
LIVELLO 2:
Regolazione dell’espressione
attraverso proteine
regolatrici
I regolatori
trascrizionali
riconoscono e
legano il DNA a
livello di specifiche
sequenze. Spesso
sono proteine
omodimeriche che
riconoscono
sequenze
palindromiche
Nei procarioti, la maggioranza
dei fattori trascrizionali
presentano un dominio helixturn-helix di legame al DNA
LIVELLO 2:
Regolazione dell’espressione attraverso proteine regolatrici
Caso 1A:
Repressione mediata da induttore
La molecola induttore si lega al repressore e questo
provoca una diminuzione dell’affinità di legame del
repressore al DNA (modificazione allosterica)
Esempio: lac operone (operone del lattosio) !!!
LIVELLO 2:
Regolazione dell’espressione attraverso proteine regolatrici
Caso 1B:
Repressione mediata da co-repressore
La molecola co-repressore si lega al repressore e questo
provoca un aumento dell’affinità di legame del repressore
al DNA (modificazione allosterica)
Esempio: trp operone (operone del triptofano)!!!
LIVELLO 2:
Regolazione dell’espressione attraverso proteine regolatrici
Caso 2:
Attivazione
Caso 2A
Caso 2B
ALCUNI REGOLATORI POSSONO COMPORTARSI SIA
DA ATTIVATORI CHE DA REPRESSORI
AraC
Ara C
-Ara
+Ara
PBAD
reprime PBAD
induce PBAD
AraC
RNA pol
PBAD
I SITI DI LEGAME PER GLI ATTIVATORI POSSONO TROVARSI
DISTANTI DAL PROMOTORE
PROTEINE ACCESSORIE POSSONO CONTRIBUIRE
ALLA REGOLAZIONE DI UN PROMOTORE
DNA-bending protein
(e.g. IHF)
Specific binding site
SISTEMI A DUE COMPONENTI
Stimolo
INPUT
HK
Sensore
His
ATP
P
ADP
P
Asp
REC
Regolatore
BD
Risposta
trascrizionale
Quorum sensing: comunicazione tra cellule.
regolazione di geni in funzione della densità cellulare
Molecola segnale
Attivatore
trascrizionale
Gene bersaglio
Bassa densità cellulare
Alta densità cellulare
CURVA DI CRESCITA BATTERICA
Repressione da catabolita: curva di crescita diauxica
INDUZIONE di una via catabolica
ASSENZA DI LATTOSIO
La presenza di lattosio non basta ad
attivare l’espressione dei geni lac
La curva diauxica mostra che, se è
presente glucosio, il lattosio non
viene utilizzato
PRESENZA DI LATTOSIO
FENOMENO DELLA
REPRESSIONE DA CATABOLITA
Il promotore dell’operone lac
Sito di legame
del repressore
Sito di legame
dell’attivatore
Sito di legame
della RNA
polimerasi
In assenza di
glucosio aumenta
l’attività
dell’adenilato
ciclasi e quindi i
livelli di AMPc
PRESENZA DI SOLO GLUCOSIO = PIENA REPRESSIONE
LacI
Sito di legame
dell’attivatore
Sito di legame
del repressore
Sito di legame
della RNA
polimerasi
PRESENZA DI SOLO LATTOSIO = PIENA INDUZIONE
AMPc
CAP
LacI
Sito di legame
del repressore
Lattosio
Sito di legame
dell’attivatore
Sito di legame
della RNA
polimerasi
PRESENZA DI LATTOSIO E GLUCOSIO=MANCATA ATTIVAZIONE
AMPc
CAP
LacI
Sito di legame
del repressore
Lattosio
Sito di legame
dell’attivatore
Sito di legame
della RNA
polimerasi
REPRESSIONE DA CATABOLITA
•I livelli cellulari di AMP ciclico aumentano in assenza di glucosio
•AMPc è il co-induttore dell’attivatore CAP
•In presenza di lattosio + glucosio i geni lac non sono indotti perché
CAP non puo’ attivare
•L’operone è trascritto solo in presenza di lattosio ed assenza di
glucosio
LA PROTEINA CAP E’ UN REGOLATORE GLOBALE
REPRESSIONE di una via anabolica
L’RNA puo’ formare strutture secondarie che possono avere varie
funzioni
TERMINAZIONE DELLA TRASCRIZIONE rho-indipendente
Nell’mRNA del peptide leader ci sono delle
regioni che possono formare una forcina di terminazione
della trascrizione.
Il ribosoma inizia a tradurre il peptide leader,
Se c’è Trp sufficiente, il ribosoma prosegue.
Nel frattempo la RNAP sintetizza l’mRNA e
si forma una forcina di terminazione tra la
regione 3 e 4 dell’ mRNA del peptide leader
Il ribosoma inizia a tradurre il peptide leader,
Se NON c’è Trp sufficiente, il ribosoma
rallenda e si ferma sulla regione 1. Nel
frattempo la RNAP sintetizza l’mRNA e si
forma una forcina tra la regione 2 e 3 dell’
mRNA del peptide leader, QUESTO
IMPEDISCE LA FORMAZIONE DELLA
FORCINA DI TERMINAZIONE TRA LA
REGIONE 3 E LA 4
LA TRASCRIZIONE CONTINUA
ATTENUAZIONE
Lo stesso regolatore trascrizionale può agire sia
da repressore che da attivatore
Alta osmolarità
OmpC
Bassa osmolarità
EnvZ
OmpF
Poro piccolo
Poro grande
OmpR
OmpR lega il DNA solo quando è fosforilata e funziona da attivatore o da
repressore a seconda di quali siti occupa sul DNA.
La quantità di OmpR fosforilata è determinata dalla percezione della
pressione osmotica da parte della kinasi EnvZ.
La quantità della proteina OmpR fosforilata nella cellula determina quale
proteina tra OmpF e OmpC viene prevalentemente espressa.
BASSA OSMOLARITA’ (ambiente)
EnvZ non è stimolata
OmpC
PREVALE
OmpF
EnvZ
Fosforilazione
OmpR a livello basale
OmpR
OmpC
ATTIVAZIONE
OmpF
Sito di legame a bassa affinità
Sito di legame ad alta affinità
ALTA OSMOLARITA’ (intestino)
PREVALE
OmpC
EnvZ stimolata
OmpF
Fosforilazione
OmpR
ATTIVAZIONE
OmpC
REPRESSIONE
OmpF
Sito di legame ad alta affinità
Sito di legame a bassa affinità
REGOLAZIONE MEDIATA DA RNA ANTISENSO
Alta osmolarità (intestino)
EnvZ
OmpC
Fosforilazione
OmpR
Temperature elevate
Stress ossidativo/Tossico
sintesi di OmpC
174 b
mRNA per OmpF
micF RNA
UNITA’ TRASCRIZIONALI
SINGOLO GENE
OPERONE
REGULONE/MODULONE
REGOLAZIONE GLOBALE
Il regulone
Il regulone è l’insieme di più geni o operoni sotto il controllo della stessa
proteina regolatrice. I geni che lo costituiscono sono implicati nello stesso
pathway
Il regulone per la biosintesi dell’arginina
Gene o operone
arg A
arg B-C-E-H
arg D
arg F
arg G
localizzazione
60 min
88 min
72.5 min
6 min
68 min
Esempio di regulone sottoposto a controllo negativo: tutti i geni per la
catena biosintetica dell’arginina sono regolati da un repressore
Sistemi di controllo globale dell’espressione genica
Un batterio può regolare molti geni diversi simultaneamente in risposta a
cambiamenti ambientali. Spesso più operoni o reguloni diversi possono essere
attivati o disattivati
Il regulone è l’insieme di più geni o operoni sotto il controllo della stessa
proteina regolatrice. I geni che lo costituiscono sono implicati nello stesso
pathway
Se geni e operoni appartengono a pathway differenti si parla di moduloni
ESEMPI:
regolazione mediante fattori sigma alternativi
risposta SOS
repressione da catabolita
QUORUM SENSING
RETI REGOLATIVE
The cyanide operon is an example of complex regulation
Other QS regulators
GacS
GacA
LasRI
RsaL
RhlIR
RsmZ/Y
ANR
RsmA
hcnABCD
DNA
hcnABCD
mRNA