Prof. Maria Nicola GADALETA
E-mail: [email protected]
Facoltà di Scienze Biotecnologiche
Corso di Laurea in
Biotecnologie Sanitarie e Farmaceutiche
Biochimica e Tecnologie Biochimiche
DISPENSA N. 15
ENZIMI III: Cinetica, Parametri cinetici, Effetto di T
e pH, Reazioni a doppio substrato
La formazione del complesso ES è essenziale per
l’attività catalitica dell’Enzima.
Esso è stato il punto di partenza per:
a)l’elaborazione matematica che definisce il comportamento
cinetico delle reazioni catalizzate dagli Enzimi;
b)la descrizione teorica del meccanismo d’azione degli Enzimi.
M.N. Gadaleta
1. CINETICA ENZIMATICA
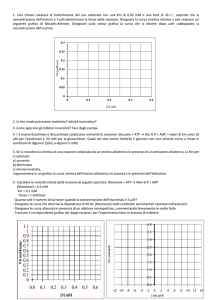
La cinetica enzimatica studia la velocità della reazione enzimatica in funzione della
concentrazione di substrato.
Caratteristica delle reazioni enzimatiche è il fenomeno della saturazione.
6.11 (Nelson & Cox IV ed.)
Nel 1903 Henry per spiegare l’andamento di tali reazioni aveva proposto la formazione
di complessi instabili ES, EP.
E+S
ES
EP
E+P
M.N. Gadaleta
Modello cinetico di Michaelis e Menten
Nel 1913 Michaelis e Menten proposero un’elaborazione matematica di tale
cinetica che portò alla formulazione dell’equazione che va sotto il nome di
“Equazione di Michaelis e Menten” e che mette in relazione la velocità di catalisi
con [S].
V0 = V max [S]
Km + [S]
dove:
V0 = velocità iniziale della reazione
V max = velocità massima
Km = costante di Michaelis = (k-1 + k2)/k1
[S] = concentrazione del substrato libero
M.N. Gadaleta
Modello MM: la formulazione di MM si basa su un modello di reazione in cui,
per definizione, è coinvolta una sola molecola di S:
k1
E+S
k-1
k2
ES
E+P
e fa le seguenti assunzioni sulle condizioni di reazione:
V0 = velocità iniziale della reazione quando P non si è ancora formato e
quindi è P = 0
stadio lento della reazione è il secondo per cui V0 = k2·[ES] (7.10)
[S] è 4-5 ordini di grandezza maggiore di [E] per cui [ES] può considerarsi
costante nei primi 60 sec.
[Et] = [enzima totale] = [E] + [ES]
[E] = [enzima libero] = [Et] – [ES]
Vmax = k2 [Et]
M.N. Gadaleta
La velocità di formazione di [ES] è la prima tappa veloce e reversibile della reazione
k1
E+S
k-1
ES
mentre la tappa di demolizione di [ES] è più lenta
k2
ES
Tappa 1.
E+P
Velocità di formazione di ES = k1 [E] [S]
Velocità di demolizione di ES = k-1 [ES] + k2 [ES]
Tappa 2.
In condizioni di stato stazionario (ipotesi dello stato stazionario di
Brigs e Haldane), cioè quando [E] viene mescolato con un grande
eccesso di S, ES rimane costante perché la sua velocità di
formazione è uguale a quella della sua demolizione, per cui:
k1 [E] [S] = k-1 [ES] + k2 [ES]
Possiamo sostituire ([Et] – [ES]) a [E]
k1 ([Et] – [ES]) [S] = k-1 [ES] + k2 [ES] (7.13)
M.N. Gadaleta
da: Nelson & Cox
M.N. Gadaleta
da: Nelson & Cox
V0 = k2·[ES]
V max = k2 [Et]
Questa è l’Equazione di Michaelis e Menten che è l’equazione della velocità
di una reazione a singolo substrato catalizzata da un enzima. Essa esprime
la relazione quantitativa tra velocità iniziale (V0), velocità massima iniziale
(V max) e la concentrazione iniziale di S [S] tra loro correlate da Km.
M.N. Gadaleta
V0 = V max [S]
Km + [S]
6.12 (Nelson & Cox IV ed.)
Vmax = [S] scomparso o [P] formato nell’unità di tempo = misura
la velocità della reazione enzimatica e l’attività dell’enzima in
concentrazioni di S saturanti
M.N. Gadaleta
V0 = Vmax [S]
Km + [S]
L’equazione di Michaelis e Menten
descrive l’equazione di una iperbole
equilatera.
Notare nella figura l’ordine della velocità
di reazione rispetto a [S]: a [S] bassissima,
[S] al denominatore può essere trascurata
rispetto a Km: V0 = K[S] (reazione di
primo ordine rispetto a [S], dipende solo
da [S]).
A [S] molto elevate rispetto a Km, Km può
essere trascurata rispetto a [S]: V0 = Vmax
(reazione di ordine 0 rispetto a [S], non
dipende da [S]).
Nel tratto intermedio della curva
l’equazione è di ordine misto rispetto a
[S].
M.N. Gadaleta
da: Champe et al.
Parametri cinetici che caratterizzano un enzima
nel modello di MM:
• Km (affinità per il substrato)
• Vmax (attività catalitica)
• Kcat/Km (potere catalitico)
1)Km= affinità di E per S
La specificità o affinità di un enzima
per il substrato è espressa dalla Km
Quando V0 = ½ Vmax sostituendo si ha:
1 Vmax = Vmax
[S]
2
[S] + Km
1 =
[S]
2
[S] + Km
[S] + Km = 2 [S]
1
Km = [S] quando V =
2
Vmax
Km ha le dimensioni di mmoli·l-1 o mM
Il valore di Km riflette l’affinità di
un enzima per il substrato:
più alta è la Km, più bassa è l’affinità
dell’enzima per S.
M.N. Gadaleta
da: Nelson & Cox, IV ed.
M.N. Gadaleta
2) Vmax = attività catalitica
Vmax esprime l’attività catalitica dell’enzima
• la Vmax rivela il numero di reazioni catalizzate da una
molecola di E nell’unità di tempo
In condizioni di saturazione di [E], cioè quando [S] è maggiore di 10·Km,
attraverso la Vmax si misura l’attività dell’enzima:
Vmax = k2 [Et]
infatti la velocità massima è direttamente proporzionale alla quantità
dell’enzima totale
• V
max
= k2 [Et]; significa che per raddoppiare la velocità di una reazione
catalizzata da un enzima è necessaria una quantità doppia di enzima totale
M.N. Gadaleta
• La misura della Vmax è importante per
misurare la quantità di E presente in
una liquido o in un estratto biologico,
perchè la Vmax si può misurare in
laboratorio al contrario di [Et].
• Una unità internazionale (U.I.) di E è la
quantità di E capace di trasformare una
µmole di S in 1 min.
Nella diagnostica enzimatica
Es. Vmax di un enzima nel siero di un paziente = 10 Vmax di quell’enzima
nell’individuo normale. Ciò vuol dire che nel siero del paziente l’enzima è
presente in quantità 10 volte superiore a quella di un individuo normale (es.
transaminasi, GOT, GPT)
Nella purificazione di un enzima
Es. attività specifica degli enzimi = Unità di E/mg prot
L’attività specifica durante la purificazione di un E deve aumentare fino a
quando l’E diventa puro: la strategia di purificazione, infatti, mira a scartare
tutte le proteine dell’estratto proteico ad eccezione della proteina che si vuole
purificare.
M.N. Gadaleta
ci dà una retta
KM
1
1
---- x ---- = - -----Vmax
[S]
Vmax
da: Nelson & Cox (modificato)
1
1
Vmax
1
---- = - ------ x -----= - ---[S]
Vmax
KM
KM
y= ax + b
Questa equazione è
1
per x = 0 ---- = 0
[S]
1
---V0
1
= -----Vmax
1
per y = 0 ---- = 0
V0
M.N. Gadaleta
da: Kleinsmith
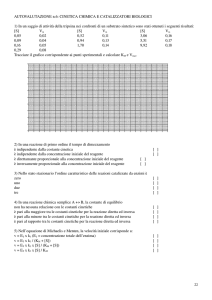
Km e Vmax sono i parametri cinetici che caratterizzano un enzima che segue il modello MM:
possono essere misurati sperimentalmente.
[E] fissa
La velocità di una reazione
si misura dalla quantità
di substrato (S)
consumato o di
prodotto (P)
formato nell’unità
di tempo
in condizioni di saturazione
ossia di Vmax.
Le unità di misura dell’attività enzimatica possono essere arbitrariamente fissate dallo
sperimentatore.
M.N. Gadaleta
Kcat (costante catalitica)
Nel meccanismo a 2 tappe proposto da M e M,
Vmax = k2 [Et]
k2 è la costante della reazione che limita la velocità.
Per reazioni a 3 tappe:
k1
E+S
k2
EP
ES
k-1
k3
E+P
k-2
K3 è la costante della tappa che limita la velocità di tutta la reazione.
In questo caso
Vmax = k3 [Et].
Viene definita Kcat la K della tappa che limita la velocità della reazione
globale. Per cui, in condizioni di saturazione di S ,si ha
Vmax = Kcat [Et].
M.N. Gadaleta
La Kcat è una costante di prim’ordine espressa in sec.-1 detta
anche numero di turnover.
Kcat = numero di turnover = numero di molecole di substrato che
vengono convertite in prodotto nell’unità di tempo da una singola molecola
enzimatica quando è saturata dal substrato (cioè quando [S] è maggiore di
10·Km).
Kcat esprime l’attività catalitica dell’enzima (è come Vmax ma espressa in
termini più generali).
Per gli enzimi che seguono il modello di M e M la Kcat = k2
M.N. Gadaleta
da: Nelson & Cox, IV ed.
M.N. Gadaleta
3) Kcat/Km (potere catalitico)
Kcat/Km, detta anche costante di specificità, esprime il potere catalitico di un
enzima, ossia di quante volte l’enzima aumenta la velocità della reazione non
catalizzata.
E’ un altro parametro che caratterizza l’enzima e che permette di meglio
confrontare il potere catalitico di più enzimi.
A [S] molto bassa Km può essere diversa che a [S] più elevate. Pertanto quando S
è <<< Km (vedi condizioni fisiologiche) è necessario tenere conto sia della
Kcat che della Km. Poiché [S] è trascurabile e Vmax è uguale a Kcat [Et]
sostituendo in
V0 = Vmax [S]
Km + [S]
si ottiene
V0 = Kcat [Et] [S]
Km
reazione di secondo ordine: la velocità dipende da [Et] ed
[S]; si esprime in M-1 sec-1.
M.N. Gadaleta
Il limite superiore del valore di questo rapporto è imposto dalla
velocità con cui i reagenti diffondono uno verso l’altro in soluzione
acquosa: questo limite dipende dalla velocità di diffusione che varia
da 108 a 109 M-1 sec-1.
Perfezione cinetica degli enzimi: catalizzano una reazione ogni volta
che incontrano S: per es. anidrasi carbonica, triosofosfatoisomerasi.
M.N. Gadaleta
da: Voete & Voet, II ed.
M.N. Gadaleta
Ogni reazione enzimatica è caratterizzata da un optimum di
temperatura
La velocità delle reazioni enzimatiche oltre che dal substrato dipende anche dalla
temperatura ambientale
L’optimum di temperatura è la risultante di due fenomeni
descritta da una curva a campana:
M.N. Gadaleta
La velocità delle reazioni enzimatiche è influenzata
dalla temperatura
• La velocità di una reazione enzimatica dipende dalla temperatura (vedi
costante di Boltzmann)
Q10 = parametro che indica di quante volte viene attivato un enzima
se la temperatura del sistema aumenta di 10°C
Q10 ~ 2,0
• Il Q10 varia da un enzima all’altro in quanto dipende dall’energia di
attivazione della reazione (∆
∆G‡)
• Di solito oltre i 55-60°C tutti gli enzimi si denaturano. Fanno
eccezione gli enzimi dei batteri termofili che resistono a
temperature superiori a 85°C (vedi Taq polimerasi)
M.N. Gadaleta
Ogni reazione enzimatica è caratterizzata da un optimum di pH
da: Nelson & Cox
La velocità di una reazioni enzimatica oltre che dal substrato e dalla temperatura
dipende anche dal pH ambientale
L’optimum di pH è anch’esso la risultante di
fenomeni di attivazione o di denaturazione
della proteina enzimatica in funzione del grado
di dissociazione degli amminoacidi importanti
per la struttura e la funzione della proteina
stessa.
M.N. Gadaleta
La velocità delle reazioni enzimatiche è influenzata dal pH
Curva a campana = risultante di due titolazioni che si sovrappongono
Le modificaizoni di pH influenzano le proprietà ioniche (acido-basiche) di
substrati (S) ed enzimi (E) e anche la conformazione di E
La curva a campana varia con [S] per cui la Km degli E cambia con il pH
Le curve più significative sono quelle che si ottengono quando l’E è saturato
da S a tutti i valori di pH
Il pH ottimale di un E non coincide necessariamente con il pH del suo
normale ambiente cellulare ma può essere più alto o più basso.
VANTAGGIO: il pH ambientale può regolare l’attività di E
M.N. Gadaleta
Non tutte le reazioni seguono il modello di MM: molte reazioni
cellulari sono a più substrati con cinetiche complesse.
Nella cellula le più comuni sono le reazioni a 2 substrati che vengono
classificate in
• reazioni a spostamento singolo con formazione di un complesso ternario
• reazioni a spostamento doppio senza formazioni di un complesso ternario
da: Nelson & Cox, IV ed.
M.N. Gadaleta
Reazioni enzimatiche a 2 substrati:
a) Reazioni a spostamento singolo con meccanismo ordinato: i substrati si
legano in modo sequenziale prima uno poi l’altro in ordine preciso. Es.
deidrogenasi piridiniche:
acido malico +
S2
NAD+
MDH
acido ossalacetico + NADH + H+
S1
P1
P2
Reazioni a spostamento singolo con meccanismo casuale: i substrati si
legano senza un ordine preciso. Es. fosfotransferasi:
glucosio + ATP
S1 o S2
S1 o S2
esochinasi
glucosio 6-P + ADP
P1 o P2
P1 o P2
M.N. Gadaleta
b) Reazioni a spostamento doppio: meccanismo a ping-pong. L’enzima lega il primo
substrato, lo modifica e rilascia il primo prodotto. Successivamente l’enzima (a sua volta
modificato) lega il secondo substrato e rilascia, poi, il secondo prodotto (es.
amminotransferasi).
Amminotransferasi o transaminasi
Enzimi che trasferiscono un –NH2 da un AA a un α cheto acido trasformandoli nell’α
αchetoacido e nell’α
α-AA corrispondenti.
GOT glutammico-ossalacetico transaminasi = aspartatoamminotransferasi
Gruppo prostetico: piridossalfosfato-piridossaminfosfato
α-chetoglut. (S2)
Aspartato (S1)
E
E-Aspartato
E-NH2-Ac. ossalacetico
E-NH2
Ac. ossalacetico (P1)
E-NH2-α-chetoglutarato
E-Glutammato
E
Glutammato (P2)
E = enzima-piridossalfosfato
E-NH2 = enzima-piridossamminafosfato
M.N. Gadaleta
da: Nelson & Cox, IV ed.
M.N. Gadaleta