Microbiologia
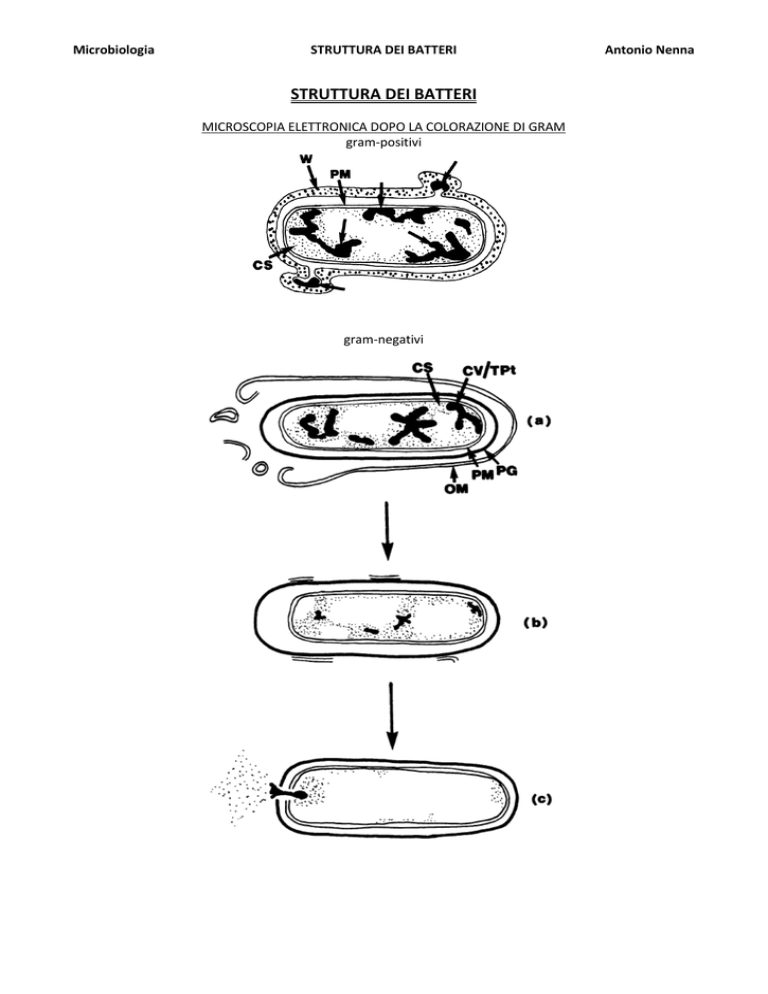
STRUTTURA DEI BATTERI
STRUTTURA DEI BATTERI
MICROSCOPIA ELETTRONICA DOPO LA COLORAZIONE DI GRAM
gram-positivi
gram-negativi
Antonio Nenna
Microbiologia
STRUTTURA DEI BATTERI
Antonio Nenna
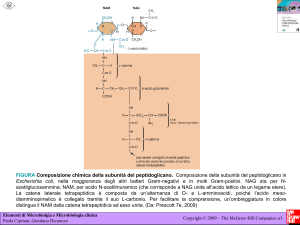
PEPTIDOGLICANO
Il peptidoglicano è un componente unico ed essenziale dei batteri, ed è formato da catene saccaridiche
unite da ponti peptidici; c’è molta differenza nella struttura tra le diverse specie batteriche, e inoltre varia
con le condizioni di crescita. E’ importante per preservare l’integrità del batterio, infatti ogni inibizione della
sintesi causa la lisi cellulare. Inoltre serve a mantenere la struttura della cellula e viene usato come zattera
per ancorare le proteine più esterne, come gli acidi teicoici. E’ quindi direttamente coinvolto nei processi di
crescita e divisione cellulare. E’ assente in Micoplasma, Rickettsia e Chlamidia.
Le catene di glicano sono formate da dimeri di N-Ac-glucosamina (NAG) e N-Ac-muramina (NAM) uniti da
legame β1→4. Ogni NAM ha un residuo sostituito da un peptide “stem” formato spesso da
L-ala / D-glu / ac. meso-diaminopimelico [mesoA2pm] (o L-lys) / D-ala / D-ala
Le catene di glicano sono formate per oligomerizzazione di unità disaccaridiche (NAG-NAM) attraverso
reazioni di transglicosilazione. Nei gram-positivi le catene possono contenere NAG o NAM all’estremità
riducente, mentre nei gram-negativi le catene non contengono NAG o NAM all’estremità riducente, ma con
molecole con legami ciclici interni (C1-C6-NAM).
I legami crociati nel peptidoglicano avvengono tra il gruppo carbossile della D-ala in posizione 4 e il gruppo
amminico di A2pm, direttamente o tramite un piccolo peptide ponte.
Il peptidoglicano quindi generalmente protegge il batterio dalla lisi osmotica. Inoltre nei gram-positivi è
usato anche come zattera per molti fattori di virulenza, mentre nei gram-negativi è una ancora per la
membrana esterna. Comunque, contiene pori per il passaggio di molecole diffusibili e nutrienti.
PEPTIDE STEM
POSIZIONE 1. Il primo aminoacido è aggiunto dalla MurC ligasi, ed è quasi sempre L-ala; in rari casi, è gly o
L-ser. Per i Mycobacterium c’è una particolarità: M. tuberculosis e M. leprae hanno gli stessi enzimi, ma il
primo aggiunge L-ala, mentre il secondo aggiunge gly; questo è dovuto alle differenti condizioni di crescita.
POSIZIONE 2. Il secondo aminoacido è aggiunto dalla MurD ligasi, ed è sempre D-glu, poichè tutte le
modifiche in questa posizione sono successive all’aggiunta.
POSIZIONE 3. Il terzo aminoacido varia notevolmente, ed è aggiunto dalla MurE ligasi. Eì generalmente un
acido di-aminico, come mesoA2pm (per i gram-negativi) o L-lys (per i gram-positivi); si trovano anche L-orn,
lantionina, D-lys, omoserina. Le modifiche sono successive all’aggiunta. L’enzima MurE è specifico.
POSIZIONE 4 e POSIZIONE 5. Il quarto e il quindi aminoacido sono aggiunti come un dipeptide D-ala/D-ala.
La sintesi del dipeptide è fatta dall’enzima Ddl, e l’incorporazione sul peptide stem è data dalla MurF ligasi.
Nei batteri resistenti alla vancomicina, il quinto aminoacido è D-lattato o D-ser.
Il peptide stem è il punto di aggancio per la lipoproteina di Braun, tra il peptidoglicano e la membrana
esterna. Questa proteina si lega al gruppo carbossile di A2pm tramite un residuo di lys.
CROSS LINKING
Il cross linking permette di aumentare la stabilità del peptidoglicano, e viene effettuato in due modi:
1) cross-linkage 3→4: dal gruppo amminico della catena laterale in posizione3 (accettore) al gruppo
carbossilico della D-ala in posizione4 (donatore); questo è il tipo più comune; è diretto (gram-negativi) o
tramite un ponte interpeptidico (gram-positivi)
2) cross-linkage 2→4: soprattutto nei corinobatteri, dal gruppo carbossilico del D-glu in posizione2
(accettore) al gruppo carbossilico della D-ala in posizione4 (donatore); necessita di un ponte interpeptidico
con un diamino-acido (lys), ed è catalizzata dal dominio transpeptidasico delle penicillin-binding-protein.
la lunghezza del ponte interpeptidico varia tra uno e sette aminoacidi nei differenti batteri
a: E. coli
b: S. aureus
c: C. pointsettie
Microbiologia
STRUTTURA DEI BATTERI
Antonio Nenna
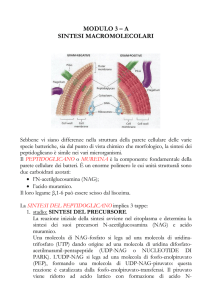
sintesi del peptidoglicano
Il punto interrogativo indica una flippasi (MviN – MurJ) che trasloca i
precursori ancorati al lipide attraverso la membrana citoplasmatica prima
della polimerizzazione nel peptidoglicano maturo.
La maturazione del peptidoglicano prevede:
- assemblaggio e allungamento delle catene di glicano attraverso glicosiltrasferasi;
- cross-linking delle catene peptidiche attraverso transpeptidasi;
- rimozione della D-ala terminale dal pentapeptide stem attraverso la DD-carbossipeptidasi.
Il peptidoglicano di nuova sintesi possiede meno cross-linking e ha meno lipoproteine con legame
covalente, ma è ricco in mureina con la catena pentapeptidica (potenziale donatore per reazioni di
transpeptidazione), ed è formato da unità di glicano più lunghe.
L’inserzione di nuove catene di glicano è problematica poichè il peptidoglicano è sempre sotto lo stress
della pressione intracellulare. Nei gram-positivi le nuove subunità sono attaccate agli strati di glicano più
vicini al citoplasma e sono spinti verso l’esterno per il continuo arrivare di nuove subunità, fino alla
degradazione che avviene sulla superficie esterna. Nei gram-negativi, invece, il peptidoglicano è più sottile
ed è formato da un unico strato di glicano; l’integrità del peptidoglicano è sempre minacciata dalla forza
necessaria ad inserire nuovi dimeri nella struttura; l’attività delle idrolasi quindi è attentamente regolata.
Considerando le variazioni della struttura del peptidoglicano (che possiede implicazioni tassonomiche), il
peptidoglicano di ogni batterio si classifica in base a un sistema a tre varianti:
- il primo carattere è una lettera romana che indica la modalità di cross linking (A per 3-4, B per 2-4)
- il secondo carattere è un numero che indica la lunghezza del ponte interpeptide
- il terzo carattere è una lettera greca che indica l’aminoacido in posizione3 sul peptide stem
Microbiologia
STRUTTURA DEI BATTERI
Antonio Nenna
peptidoglicano nei gram-positivi (a) e nei gram-negativi (b)
Le funzioni del peptidoglicano sono quattro:
- resistenza alla pressione osmotica
- forma cellulare
- divisione cellulare
- zattera per l’ancoraggio di polimeri di superficie (lipoproteine di Braun per i gram-negativi, acidi teicoici
per i gram-positivi, capsula)
Microbiologia
STRUTTURA DEI BATTERI
Antonio Nenna
FORMA E MORFOGENESI NEI BATTERI
Il peptidoglicano è importante per mantenere la forma cellulare e proteggere contro la pressione osmotica.
La distruzione del cell wall dei bastoncelli (Bacillus, Escherichia) con lisozima o penicillina genera cellula
rotonde e osmoticamente instabili (sferoplasti).
La forma a bastoncello è dovuta ai geni MreBCD e RodA, le cui proteine sono omologhi strutturali
dell’actina degli eucarioti, e formano strutture elicoidali lungo l’asse maggiore della cellula, al di sotto della
membrana citoplasmatica. MreB, la più importante, è quindi importante per l’apparato morfogenico.
Inoltre, alcuni batteri come E. coli e B. subtilis sintetizzano nuovo peptidoglicano lungo la parete laterale
durante la loro crescita, per mantenere la forma a bastoncello. La composizione del peptidoglicano di H.
pilori cambia quando la cellula muta da un bastoncello ricurvo a un cocco, in coltura. Quindi una parte della
morfologia cellulare è dovuta al peptidoglicano e al suo apparato di sintesi; quindi anche le PBP sono
importanti nella forma dei batteri, poichè questi enzimi catalizzano le reazioni di sintesi del peptidoglicano.
PENICILLIN BINDING PROTEIN
Le PBP si classificano in base al peso molecolare, alla sequenza e alla funzione enzimatica nella cellula. La
sintesi di nuovo peptidoglicano in posizioni specifiche avviene per il reclutamento di PBP differenti. Ad
esempio, nella formazione del setto di divisione del divisoma, FtsZ recluta FtsI (PBP3) nel piano di divisione.
Inoltre, nel bacillo C. crescentus, MreB recluta PBP2 per la formazione dell’elongasoma; MreB si localizza in
modo FtsZ-dipendente nel piano di divisione cellulare, indicando che MreB agisce da interruttore tra
l’elongazione della parete (elongasoma) e la sintesi del cell wall nel sito di divisione (divisoma).
L’ELONGASOMA, per l’allungamento della cellula, richiede PBP2 (PbpA) e RodA.
Il DIVISOMA, per la formazione del setto di divisione, richiede PBP3 (FtsI) e FtsW.
RodA e FtsW sono simili alla proteina SpoVE di B. subtilis, che funziona nel processo di sporulazione; queste
tre proteine sono prototipi dei SEDS (shape, elongation, division, sporulation), presenti in tutti i batteri.
Microbiologia
STRUTTURA DEI BATTERI
divisoma
elongasoma
Antonio Nenna
Microbiologia
STRUTTURA DEI BATTERI
Antonio Nenna
ELEMENTI GENETICI MOBILI
Gli elementi genetici mobili (MGE) e il trasferimento genico orizzontale (HGT) sono importanti nel
conformare il cromosoma batterico, determinando differenziazione dei ceppi, produzione di tossine,
virulenza, fitness, resistenza agli antibiotici e interferenza con i fattori di difesa dell’ospite.
ELEMENTI GENETICI MOBILI.
- plasmidi: DNA extracromosomiale lineare o circolare che si replica indipendentemente dalla cellula ospite.
I plasmidi coniugativi sono trasferiti dalla cellula donatrice alla ricevente, e necessitano di contatto fisico tra
le cellule mediante il pilus.
- batteriofagi: virus dei batteri che usano i macchinari dell’ospite per replicare; il DNA del fago entra la
cellula ospite e si integra nel genoma batterico, e viene passivamente ereditato nelle generazioni
successive, fino alla riattivazione litica del batterio.
- trasposoni: segmenti di DNA limitati da ripetizioni invertite, codificanti proteine che facilitano il
movimento tra regioni cromosomiche.
MECCANISMI DI TRASFERIMENTO DI DNA
- trasformazione: tramite l’uptake di DNA libero extracellulare
- coniugazione: tramite ICE (integrative conjugative elements), richiede il contatto cellulare
- trasduzione: tramite batteriofagi
- trasposizione: tramite trasposoni

![Lezione 3 La cellula procariotica [modalità compatibilità]](http://s1.studylibit.com/store/data/005508193_1-fc5aabf7dca9419233c48b9f769fd1e4-300x300.png)