Progetto “I LOVE RESEARCH”:
Progettazione di un database per la raccolta e la
gestione di dati Clinici e Sperimentali.
Tutor: Carmelo Laudanna, Gianluca Santamaria
Il progetto fa parte dell’iniziativa “I Love Research” promossa dalla Fondazione Univesità “Magna
Graecia” di Catanzaro in collaborazione con l’Istituto di Istruzione Superiore "Malafarina" di
Soverato (CZ). Cinque studenti del quinto anno sono stati selezionati e seguiti da un gruppo di tutor
(assegnisti e/o dottorandi) dell’Università “Magna Graecia”, per essere coinvolti nello sviluppo di
un progetto di ricerca.
Il progetto ha come scopo lo sviluppo di un database web-based per la raccolta, la gestione e
l’analisi bioinformatica di dati clinici e sperimentali generati dai diversi dipartimenti afferenti
all’università “Magna Graecia” di Catanzaro.
BACKGROUND
L'importanza delle informazioni è dovuta dal fatto che esse possono essere archiviate in modo
permanente e ritrovate quando devono essere utilizzate. Il vantaggio che se ne trae è quello di poter
ottenere, dall'elaborazione dei dati ritrovati, informazioni che potrebbero rivelarsi indispensabili o
strategiche nelle decisioni. Anche se potrebbero sembrare due sinonimi, i termini dato e
informazione hanno un significato diverso:
“dato” è un valore astratto, per esempio un numero, che per essere utilizzato deve essere
interpretato;
“informazione” è il significato contenuto nel dato che viene alla luce quando il dato viene
interpretato.
È molto importante decidere il modo in cui i dati vengono conservati perché ciò determinerà
successivamente anche il modo in cui i dati verranno estratti; ad esempio, potrebbe capitare che non
preoccupandosi per tempo dell'organizzazione dei dati durante l'archiviazione , si abbiano difficoltà
o complicazioni durante la fase del loro recupero.
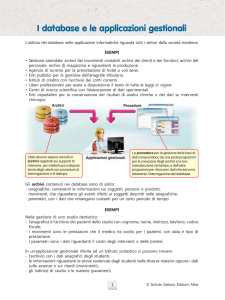
Mentre una volta gli archivi potevano essere su supporto cartaceo, oggi quasi tutti gli archivi si
possono trovare in forma digitale su supporto elettronico, e ciò rende possibile la loro elaborazione
automatizzata. Per esempio, un archivio può avere la forma di un file di dati opportunamente
organizzati.
Utilizzare i singoli file per creare archivi di dati può essere pratico per piccole applicazioni, ma in
generale, questo approccio, può dar luogo anche a diversi problemi. Per esempio: se un’Azienda
Ospedaliera utilizza un file per conservare i dati dei propri pazienti e un file per conservare i dati
delle relative cartelle cliniche, bisogna preoccuparsi di mantenere «sincronizzati» i dati tra i due
archivi. Se l’Azienda Ospedaliera permette l'aggiornamento degli archivi da parte di più
Reparti/Dipartimenti, i problemi aumentano.
I database nascono proprio per superare i limiti e gli inevitabili problemi che si potrebbero
incontrare usando gli archivi tradizionali. Tra i classici problemi vanno ricordati la ridondanza dei
dati e l'inconsistenza degli archivi. Il primo caso si verifica quando in diversi archivi si trovano
memorizzati gli stessi tipi di dati (il problema è quello di dover fare gli aggiornamenti in tutti gli
archivi interessati).
Il secondo caso si verifica come conseguenza della ridondanza, quando questi aggiornamenti non
vengono fatti in tutti gli archivi in cui si dovrebbe, e nascono così incongruenze tra dati nuovi e dati
vecchi.
BASE DI PARTENZA SCIENTIFICA
Nell’ultimo decennio in ambito clinico e biologico è diventato necessario lo sviluppo di sistemi per
l’archiviazione, gestione e analisi dei dati, proprio per un crescendo in ordine esponenziale
dell’ammontare dei dati prodotti. Soprattutto con l’avvento della “Next Generation Sequencing”
(NGS) il bisogno di strutture in grado di gestire grosse quantità di dati è una condizione essenziale.
Alcuni degli esempi più importanti sono soluzioni proposte dalle stesse aziende produttrici dei
sequenziatori di nuova generazione come “BaseSpace” dell’Illumina
(http://www.illumina.com/informatics/research/sequencing-data-analysismanagement/basespace.html), un sistema cloud-based di genomica che permette non solo di
archiviare dati ma anche di analizzarli. Altre soluzioni possibili sono quelle adottate da centri di
ricerca internazionali come l’European Molecular Biology Laboratory (EMBL) attraverso
l’European Bioinformatics Institute (EBI).
L’EBI è stato creato dall’EMBL ed ospita database biologici fra i più importanti, includendo quello
delle sequenze di DNA o sequenze nucleotidiche (ENA), delle sequenze di proteine (UniProt), dei
genomi animali (Ensembl), delle strutture tridimensionali (la Protein Databank), dei dati di
espressione genica (ArrayExpress), delle interazioni proteina-proteina (IntAct) e delle vie di
“signaling” (Reactome), (www.ebi.ac.uk/information).
METODOLOGIA
Gli studenti hanno utilizzato un ambiente di sviluppo di un database , un linguaggio informatico
descrittivo ed eseguito le seguenti attivita’:
•
•
•
•
•
progettare insieme ai tutor lo schema concettuale del database
implementare i costrutti Access necessari alla creazione ed alla generazione delle instanze
del database
progettare la componente strutturale del database
implementare un’interfaccia grafica
documentare l’intero progetto
Il software database utilizzato per il progetto è basato su Microsoft Access, un noto DBMS (Sistemi
per la Gestione di Database). Questo tipo di DBMS permette affidabilità e privatezza garantendo
resistenza a malfunzionamenti hardware e software e controllo degli accessi, e un buon livello di
efficienza ed efficacia. Permette, inoltre, la possibilità di poter traslare le istanze generate per dei
software DBMS più performanti come MySql (open-source). Infine tramite HTML è stata generata
ed integrata la struttura grafica del database.
CONCLUSIONI
Lo sviluppo del database potrebbe permettere un miglior utilizzo di dati tra diversi dipartimenti,
rendendo molto più veloce e sicura la condivisione dei dati stessi. Ogni Dipartimento, in questo
modo, può essere inteso come unità organizzativa che può essere divisa in settori o comunque può
svolgere diverse attività. A ciascun settore o attività corrisponde un sotto-sistema informativo
(privato o porzione di un sistema più grande).
Il database è in grado di favorire un recupero dei dati in modo semplice e istantaneo, come semplice
e istantanea è l’associazione dati-paziente. Questa procedura è utile anche ai fini di ricerca in quanto
permette un utilizzo dei dati, nel rispetto della privicy, per un’analisi a valle sempre più integrata tra
la componente clinica e quella di ricerca.