Indicatori
microbiologici e molecolari
Indicatori microbiologici
Dosaggio della carica microbica
Rappresenta il numero di microrganismi, appartenenti ad
un gruppo fisio-tassonomico generale (lieviti, funghi,
batteri, actinobacteri, protozoi), oppure ad uno
specifico gruppo fisiologico o funzionale (azotofissatori,
proteolitici, ammonificanti, nitrificanti, denitrificanti,
cellulosolitici, solfo-ossidanti, etc.), normalmente stimati
in relazione al grammo di suolo.
Si può stimare la
dimensione (quantità), o la
composizione (qualità) della microflora tellurica.
Può essere determinata per via
diretta (osservazione al microscopio)
indiretta (colturale)
La carica microbica per via diretta
Si utilizzano dei coloranti per marcare cromaticamente le cellule
microbiche da osservare successivamente al microscopio
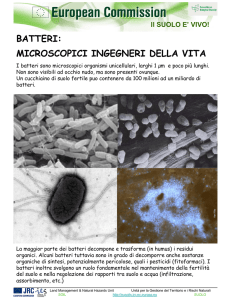
Batteri
Batteri
Funghi
La carica microbica per via indiretta
La carica microbica viene determinata utilizzando
terreni colturali agarizzati (in piastra) oppure
liquidi, validi per gruppi generici di microrganismi,
oppure “selettivi”
Schema generale di
estrazione dal suolo e
messa in coltura di
cellule microbiche
Dopo incubazione delle
piastre, le colonie
formate vengono
enumerate
Tuttavia…
Il 99% delle specie di batteri del suolo è
sconosciuto (Ritz et al., 2003).
Il 95% dei batteri conosciuti (1%) non è
coltivabile (Lynch, 2003).
Metodi di analisi molecolare
Permettono lo studio delle comunità microbiche del suolo
in relazione alla loro composizione, struttura, dinamica e
funzione
Analisi di steroli e di fosfolipidi di
membrana (FAME, PLFA)
Analisi di acidi nucleici (DNA e RNA)
Acidi grassi di fosfolipidi (PLFA)
per g soil: µg BC = 5.8·nmol total PLFA
Dosaggio dell’ATP
Dosaggio del DNA
per g soil: µg BC = 6.0·µg DNA
Metodi di indagine molecolare
Il 16S rDNA rappresenta la molecola di elezione per l’analisi
della diversità batterica ed è attualmente la biomolecola più
utilizzata nelle indagini di caratterizzazione biomolecolare per
lo studio delle comunità batteriche del suolo
Variabili misurate
- Proprietà fisiche
Sabbia, limo, argilla
- Proprietà chimiche
TOC,TN, pH, CEC, EC, TCa, ACa
- Proprietà biochimiche
Cmic, Nmic, Extr-C, Extr-N, SBR, qCO2
- Attività enzimatiche
fosfatasi acida (AcP), fosfatasi alcalina (AlkP),
arilsolfatasi (ArS), deidrogenasi (DH),
-glucosidasi (Glu), FDA-idrolasi (FDA)
- Struttura molecolare via PCR-DGGE
Variabili indagate: 22
Profili campionati: 11 (x 3 replicati)
Soil bacterial community analysis
Direct soil DNA extraction
and purification
PCR with bacterial primers
targeting the V6-V8
hypervariable regions
16S rDNA
Denaturing gradient gel electrophoresis
(DGGE) analysis of PCR-amplified 16S
rDNA fragments.
Fragments with the same length but
different
base
composition
are
separated along a denaturing chemical
(Urea + Formamide) gradient.
40% UF
58% UF
DGGE bacterial community fingerprinting
1-Ap
1-Bw
1-Bk
2-Oa
2-A
2-AB
N
DGGE bacterial community fingerprinting
BS 3-Ap
3-Bw
4-A
4-Bw
4-BC
N
Dendrogramma di similarità
Cluster 1
Cluster 2
Cluster 4
Cluster 3
Cluster 5
Cluster 6
I processi di pedogenesi determinano l'evoluzione del suolo con la
formazione di orizzonti diversi - caratterizzati da distinte proprietà
chimico-fisiche - che ospitano diversificate comunità microbiche
funzionalmente attive, le quali, a loro volta, sostengono il processo
pedogenetico dell’orizzonte e ne determinano l’evoluzione