1
Systems Biology: the 21st Century Science
Whole-istic biology
Systems biology is the study of an organism, viewed as an integrated and interacting
network of genes, proteins and biochemical reactions which give rise to life.
2
ANALOGY
If you wanted to study an
automobile, and focused on
identifying the engine, seat
belts, and tail lights, and
studied
their
specific
functions, you would have
no real understanding of
how
an
automobile
operates.
?
More important, you would have
no understanding of how to
effectively service the vehicle
when something malfunctions.
?
3
So too, a traditional approach to studying biology and human health has left us with
a limited understanding of how the human body operates, and how we can best
predict, prevent, or remedy potential health problems.
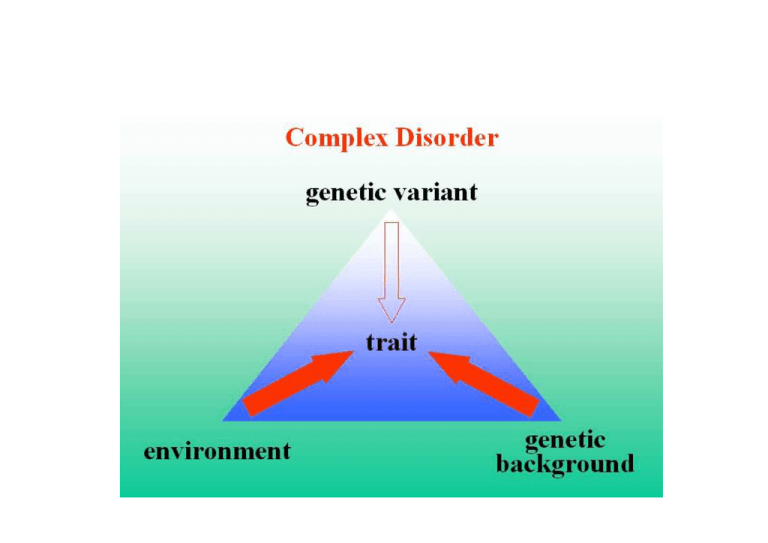
Researchers have had limited success in curing complex diseases such as
cancer, HIV, and diabetes because traditional biology generally looks at only
a few aspects of an organism at a time.
4
Genes and proteins almost never work alone. They interact with each other and
with other molecules in highly structured but incredibly complex ways, similar to the
complex interactions among the countless computers on the Internet.
Systems biology aims at explaining the properties and behavior of complex
biological systems such as the cell or its molecular machineries
5
6
Metodologie High-Throughput
(HT)
7
DNA microarrays consist of thousands of individual gene sequences bound to
closely spaced regions on the surface of a glass microscope slide.
20 - 50 µm
20 - 50 µm
Millions of identical
oligonucleotide
probes per feature
49 - 400
chips/wafer
1.28cm
up to ~ 400,000 features/chip
8
Esistono di fatto due tecnologie per la produzione di
microarrays: la prima denominata a spotting e la
seconda detta in situ.
9
Nella tecnologia spotting, le sonde da
ancorare al supporto solido, normalmente un
vetrino da microscopia, sono sintetizzate a
parte e quindi depositate sul supporto.
Tali sonde possono essere costituite da
molecole di cDNA lunghe alcune migliaia di
paia di basi le cui sequenze possono essere
ricavate da banche dati genomiche
(GenBank, dbEST o UniGene)
10
Selezionate le sequenze da studiare, il cDNA
relativo viene prodotto mediante PCR
ottenendo così sonde della dimensione da
600 a 2400 bp.
Più recentemente, le sonde che vengono
depositate sono rappresentate non tanto da
frammenti di materiale genomico ottenuto via
PCR, quanto piuttosto da sequenze sintetiche
di oligonucleotidi lunghe 50-70 paia di basi.
11
Una volta prodotte, le sonde vengono depositate sul supporto solido, in
genere costituito da un vetrino. La deposizione viene effettuata da
sistemi robotizzati che mediante l’utilizzo di pennini prelevano le sonde
direttamente dalle piastre utilizzate per la PCR e le depositano sul
vetrino formando spots di circa 100-150 µm di diametro, distanziati l’uno
dall’altro 200-250 µm.
Durante la deposizione, il sistema di controllo del robot registra
automaticamente tutte le informazioni necessarie alla caratterizzazione
ed alla completa identificazione di ciascun punto della matrice (identità
del cDNA, coordinate sul supporto, ecc.). Una volta sul vetrino, il probe
viene legato covalentemente ai gruppi amminici del supporto attraverso
una reazione innescata dall’irragiamento con luce ultravioletta, mentre il
cDNA in eccesso viene rimosso con semplici lavaggi dell’array. Infine, il
cDNA sul supporto viene reso a catena singola attraverso una
denaturazione termica o chimica.
12
L’altra tecnica utilizzata per la produzione di
microarrays è quella detta in situ che,
sviluppata da Affimetrix, è frutto
dell’interazione di due tecnologie particolari, la
fotolitografia e la sintesi diretta in fase solida di
oligonucleotidi.
La sintesi delle sonde avviene direttamente
sulla superficie del supporto solido
13
14
I targets, ovvero gli acidi nucleici da ibridizzare alle catene
di cDNA ancorate al supporto solido,
sono normalmente ottenuti dalla marcatura dell’mRNA
proveniente da un dato organismo per mezzo di molecole
fluorescenti.
Probes e targets vengono poi messi a contatto per fare
avvenire la reazione di ibridazione e dopo alcuni lavaggi per
rimuovere i prodotti aspecifici, l’array viene passato
attraverso uno scanner per la misura dei segnali
fluorescenti.
L’intensità dei pixel di ciascuna immagine è proporzionale al
numero di molecole di tracciante presenti sullo spot e quindi
al numero di targets che hanno ibridizzato le sonde
ancorate al supporto.
15
16
Schematically
Before labelling
Array
17
Schematically
Labelled but before hybridization
Array
18
Schematically
After hybridization
Array
19
Schematically
Quantification
4
2
0
Array
3
20
oligonucleotide arrays
cDNA clones
21
Tumor sample
Reference sample
RNA
RNA
cDNA
cDNA
Hybridize
22
excitation
red laser
green laser
emission
overlay images and normalise
23
Di fatto, livelli diversi di fluorescenza indicano livelli diversi di
ibridizzazione e quindi di espressione genica.
Il segnale rilevato dallo scanner viene poi sottoposto ad
algoritmi di filtrazione e di pulizia del segnale e convertito in
valori numerici .
In generale, quindi, un esperimento di analisi dei profili di
espressione fornisce come risultato una matrice di dati, in cui le
righe rappresentano i geni monitorati e le colonne corrispondono
alle diverse condizioni sperimentali, quali punti temporali,
condizioni fisiologiche, tessuti.
Ogni elemento della matrice rappresenta quindi il livello di
espressione di un particolare gene in uno specifico stato
fisiologico.
Ciascuna colonna è data da un vettore che ha tante dimensioni
quanti sono i geni o le sequenze immobilizzate sull’array.24
25
26
27
È necessario, quindi, avere a disposizione
tutta una serie di tecniche computazionali
capaci di gestire ed interpretare questi
enormi database nonché di interfacciarsi
con gli strumenti bioinformatici per l’analisi
funzionale (database mining).
28
Si definiscono tecniche di database mining tutta una serie di strumenti informatici per
l’esplorazione e l’analisi di grandi quantità di dati al fine di estrarre motivi caratteristici e
persistenti (patterns) e regole. Gli algoritmi che costituiscono il database mining derivano
da campi quali la statistica, la pattern recognition, l’intelligenza artificiale e l’analisi dei
segnali; essi sfruttano le informazioni ricavate direttamente dai dati per creare dei modelli
empirici in grado di descrivere il comportamento di un sistema complesso. Nel caso dei
profili di espressione genica, le tecniche di database mining rappresentano un utile
strumento per identificare ed isolare particolari pattern di espressione che di fatto
rappresentano delle vere e proprie impronte digitali genetiche di un determinato stato
fisiologico. L’analisi dei dati degli array di cDNA è normalmente basata sull’uso sinergico
di test di ipotesi (hypothesis testing) e di sistemi per l’estrazione della conoscenza
(knowledge discovery). I metodi di hypothesis testing sono sostanzialmente degli approcci
di tipo top-down con i quali si ricercano nei dati le conferme sperimentali ad ipotesi
precedentemente formulate. La knowledge discovery può essere intesa invece come un
approccio bottom-up nel quale sono i dati stessi che forniscono le indicazioni necessarie alla
formulazione di nuove ipotesi. Un aspetto cruciale dell’applicazione di queste procedure è
l’identificazione di tutti quei geni che manifestano un’elevata attività in un determinato
stato fisiologico. Questi geni attivi, e le loro relazioni, possono essere identificati attraverso
tecniche quali Mean Hypothesis Testing (MHT), Cluster Analysis (CA), Principal
Component Analysis (PCA) e Decision Tree (DT).
29
Microarrays may be used to assay gene expression within a single
sample or to compare gene expression in two different cell types or
tissues samples, such as in healthy and diseased tissue.
Follow population of (synchronized) cells over time, to see how expression
changes (vs. baseline)
Expose cells to different external stimuli and measure their response (vs.
baseline)
Take cancer cells (or other pathology) and compare to normal cells.
30
•Developmental stage-specific gene expression
31
•Gene expression during differentiation - investigation of how gene
expression patterns are altered during differentiation.
32
Gene expression during tumorigenesis - cells can be sampled at
different recognized stages during the progression to cancer
33
ADVANTAGES
Microarray analysis offers the advantage of profiling expression levels
of hundreds or thousands of genes simultaneously using a single RNA
preparation
Human ~ 25,000 genes
Mouse ~ 25,000 genes
Theorycally, all genes of an organism can be analyzed by a single array
Easy to use
Yeast ~ 6200 genes
E. coli ~ 4200 genes
High speed
Phage T4 ~ 20 genes
Influenza ~ 12 genes
34
This technology is a very dynamic one and is currently spawning
a variety of derivative technologies including the development of
protein and antibody microarrays and cell microarrays
35
LIMITS
Obviously, this is an expression-based technology, capable only
of monitoring cellular responses at the RNA level. Some critical
signaling changes may occur only at the protein or posttranslational level and therefore would not be detected with gene
arrays.
The correlation between the number of mRNA and protein molecules is
generally not strong enough to predict one value from the measurement
of the other
DNA
transcription
mRNA
translation
36
Protein
Regolazione dell’espressione genica
Organizzazione della cromatina
Punto 1
Inizio della trascrizione
37
38
LIMITS
High cost technology
No quantitative: 3 fold variations are experimental
Data interpretation represent a complex problem:
mRNA profiling data (as other applications) typically consist of many
thousands of measurements for each array
Needs verification by other approach (i.e., Real time PCR)
39
40
41
42
43
•screening of polymorphisms and mutation
•There is also huge potential for assaying for mutations in known disease
genes, as recently exemplified in the case of the breast cancer
susceptibility gene, BRCA1. In addition, there have been vigorous efforts
to identify and catalog human single nucleotide polymorphism (SNP)
44
markers
45
46
47
Comparative Genomic Hybridization
CGH
L’ibridazione genomica comparativa (Comparative Genomic
Hybridization,
CGH)
convenzionale
è
una
tecnica
di
rilevazione che consente di analizzare l’intero genoma del
soggetto che si vuole esaminare, grazie ad in un unico
esperimento in grado di identificare anomalie del corredo
genetico quali riarrangiamenti inter– ed intracromosomici,
regioni di amplificazione genica e regioni con delezioni.
48
Il principio della tecnica si basa su una competizione per il
legame di 2 DNA genomici, marcati con fluorocromi diversi, a
cromosomi in metafase, non marcati e provenienti da un
soggetto sano.
Il primo DNA è estratto dal paziente in esame mentre l'altro
costituisce il DNA genomico di riferimento proveniente da un
soggetto sano.
Tale
processo
prende
il
nome
di
ibridazione
in
situ
su
cromosomi e, grazie allo stato altamente condensato dei
cromosomi metafisici, la risoluzione di questa tecnica (vale a
dire la capacità di riconoscere alterazioni nelle sequenze del
genoma) è nell’ordine delle 5 Mb (1 Mb = 1 milione di basi).
49
Nelle immagini si vede la stessa preparazione di cromosomi (metafase) di un soggetto
normale, fatta ibridare con
(a) il DNA estratto da cellule tumorali marcate con uno specifico fluorocromo verde, e
(b) il DNA delle cellule normali di riferimento marcati con uno specifico fluorocromo
rosso.
(c) La sovrapposizione delle immagini dimostra l’intensità relativa della fluorescenza
verde e di quella rossa, riflettendo le variazioni del numero di copie di geni che si
riscontra nel genoma tumorale. Una perdita di geni in aree più o meno estese del
DNA tumorale conferisce al DNA di riferimento un vantaggio nella competizione per
il legame al DNA normale. Proprio per questo motivo in tali aree il microscopio rileva
un aumento della fluorescenza rossa.
Viceversa, duplicazioni ed amplificazioni di geni del DNA tumorale corrispondono al
prevalere della fluorescenza verde.
50
51
BANDEGGI
• Bandeggi generali:
bande
ande
identificazione di TUTTI i cromosomi per mezzo di una serie di b
lungo tutto l’
l’intero cromosoma, diverse da un cromosoma all’
all’altro
Bande G → varie tecniche, combinate a colorazione Giemsa
Bande Q → bande fluorescenti visibili con mostarda
quinacrina o composti simili
Bande R → dopo denaturazione al calore
• Bandeggi particolari:
limitati ad aree di ogni singolo cromosoma o gruppi di cromosomi
Bande T → regioni telomeriche
Bande C → regioni pericentromeriche
Bande AgNOR → regioni dell’
dell’organizzatore nucleolare
Bande DaDa-DAPI → tratto prossimale braccio corto n.15,
costrizioni secondarie n.1,9,16 e tratto distale Yq
52
53
54
CGHarray
Il principio della CGHarray è identico a quello della CGH
convenzionale.
La differenza sta nel fatto che i due DNA (test e reference)
vengono ibridati su un microarray di frammenti di DNA
genomico
ben
caratterizzati,
invece
che
su
un
vetrino
contenente metafasi.
55
Risoluzione
CGH convenzionale
CGH array
10 Mb
1 Mb
56
57
58
Identificazione delle mutazioni
Diagnosi
pre-impianto – prenatale – postnatale – eterozigoti
Terapia
malattia minima residua – terapia specifica –
disegno di nuovi farmaci
Prevenzione
screening eterozigoti – geni oncosoppressori SNPs
59



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)


![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)





