Applicazioni biotecnologiche in
systems biology
Lezione #2
Dr. Marco Galardini
AA 2012/2013
Contatti
Dr. Marco Galardini
Dip. Di Biologia
Via Madonna del Piano 6, Polo Scientifico
S. Fiorentino (c/o Incubatore delle idee)
Email: [email protected]
Tel: 0554574737
Systems biology
Principali tecnologie di
sequenziamento e controllo qualità
Lezione #2
Dr. Marco Galardini
AA 2012/2013
Sequenziamento del genoma
Genoma: intera sequenza nucleotidica di un organismo
Sequenziamento: determinazione di una sequenza nucleotidica
Le tecniche di sequenziamento
Generazione 0 – Sequenziamento chimico
Generazione 1 – Dye-terminator
Generazione 2 – NGS con pre-amplificazione
Generazione 3 – NGS su singola molecola
NGS: Next Generation Sequencing
Sequenziamento del genoma
Le tecniche di sequenziamento
Generazione 0 - Maxam & Gilbert
Generazione 1 - Sanger
Generazione 2 – Pyrosequencing (454), Solexa-Illumina, Solid, Ion Torrent
Generazione 3 –PACBIO RS, Nanopore
Evoluzione del sequenziamento
1977
Frederick Sanger
Prima tecnica di sequenziamento
1987
Applyed Biosystems
Prima macchina automatica per il
sequenziamento del DNA
1986
Leeroy E.Hoods
Prima macchina semiautomatica per il
sequenziamento del DNA
1996
Pål Nyrén
Nascita del pyrosequencing
1998
Phil Green and Brent Ewing
Viene pubblicato il softwere “Phred” per l’analisi di
sequenze di DNA
2000
Lynx Therapeutics
Lancio sul mercato del “MPSS” e inizio del
sequenziamento di nuova generazione (NGS)
Evoluzione del sequenziamento
1977
Frederick
Frederick Sanger
Sanger
Prima tecnica di sequenziamento
1987
Applyed Biosystems
Prima macchina automatica per il
sequenziamento del DNA
Qualità e controllo
dei dati ottenuti
1986
Leeroy E.Hoods
Prima macchina semiautomatica per il
sequenziamento del DNA
Riduzione del costo
Quantità di dati
1998
Green and Brent Ewing
nell’unità di Phil
tempo
1996
Pål Nyrén
Nascita del pyrosequencing
Viene pubblicato il softwere “Phred” per l’analisi di
sequenze di DNA
2000
Lynx Therapeutics
Lancio sul mercato del “MPSS” e inizio del
sequenziamento di nuova generazione (NGS)
Automatismi di sequenziamento
?
Automatismi di sequenziamento
Maggiore automazione, industrializzazione del sequenziamento
Concentrazione di sequenziatori presso centri genomici (economia di scala)
Riduzione del numero di singoli sequenziatori presso laboratori di ricerca
Utilizzo della genomica per applicazioni cliniche, farmaceutiche, industriali
Incremento della complessità
Sanger sequencing
Risorse per la lettura ed
immagazzinamento delle
sequenze
Quantità di basi lette
Nell’unità di tempo
NGS
Sequenziamento del genoma
Le tecniche di sequenziamento
Generazione 0 - Maxam & Gilbert
Generazione 1 - Sanger
Generazione 2 – Pyrosequencing (454), Solexa-Illumina, Solid, Ion Torrent
Generazione 3 –PACBIO RS, Nanopore
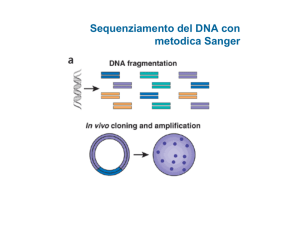
Generazione 1 - Sanger
ovvero l’idea geniale dei dideossi!
Sanger sequencing: Cycle Sequencing
Molecola segnale
La bassa percentuale di ddNTP
fa sì passi del tempo prima che
la sintesi venga interrotta
Animation @ dnalc
Sequenziamento manuale
• Di-deossi nucleotidi radioattivi
• Una lane per ogni nucleotide
• Esistono sequenziatori
automatici che leggono
automaticamente i gel di
poliacrilamide
Sequenziamento automatico
• Di-deossi nucleotidi fluorescenti
• Un colore per ogni nucleotide
• Una singola lane per i 4 nucleotidi
• Separazione attraverso elettroforesi
capillare
• Lettura nucleotidi tramite laser
• Macchinari multi-capillari per letture
in parallelo
Cromatogramma
Lunghezza dei frammenti raggiunta: fino a 1000 bp
Cromatogramma – possibili errori
Sovrapposizione
fra i picchi
Omonucleotidi
Difficile separazione fra i picchi
Sovrapposizione
fra i picchi
Necessario un approccio algoritmico per determinare la qualità dei
cromatogrammi ed individuare la giusta sequenza
Generazione 2 – NGS con pre-amplificazione
Pyrosequencing (454)
Solexa-Illumina
Solid
(Anche la tecnica Sanger prevede un’amplificazione del dna prima del
sequenziamento, con successiva ligazione di un primer)
NGS, seconda generazione
NGS, seconda generazione
Viene commercializzata
la prima macchina
automatica per il
pirosequenziamento
Prima pubblicazione
sul
pirosequenziamento
Primo
sequenziamento
automatico
Primo sequenziamento
semi-automatico
(Sanger dye-terminator)
NGS, seconda generazione
454 pyrosequencing
Viene commercializzata
la prima macchina
automatica per il
pirosequenziamento
Prima pubblicazione
sul
pirosequenziamento
Primo
sequenziamento
automatico
Primo sequenziamento
semi-automatico
(Sanger dye-terminator)
Illumina (Solexa)
SOLiD
Ion semiconductor
DNA nanoball
Helioscope
SMRT/RNAP
NGS, seconda generazione
454 pyrosequencing
Viene commercializzata
la prima macchina
automatica per il
pirosequenziamento
Prima pubblicazione
sul
pirosequenziamento
Primo
sequenziamento
automatico
Primo sequenziamento
semi-automatico
(Sanger dye-terminator)
Illumina (Solexa)
SOLiD
Ion semiconductor
DNA nanoball
Helioscope
SMRT/RNAP
Il Pyrosequencing (o 454)
La tecnologia 454
• Da 400 a 700 basi per read
• Circa la metà rispetto al Sanger
• 400-600 megabasi ogni 10 ore
• Più veloce del Sanger
• Costi medio alti
• Impossibilità di sequenziare
correttamente più di 8 basi
identiche consecutive
(omonucleotidi)
Il Pyrosequencing (o 454)
• Amplificazione tramite emulsion PCR (emPCR)
• Multiple reazioni di PCR nella stessa provetta
• Sospensioni acquose in “olio”
• Frantumazione DNA
• Primer di PCR legati a microsfere
• Intrappolamento microsfere in micropiastra
• Una microsfera per pozzetto
La tecnologia 454
Il Pyrosequencing (o 454)
Pirosequenziamento
• Inserimento di un nucleotide per volta
• Incorporazione di un nucleotide da parte della
DNA polimerasi comporta lo sviluppo di luce
• Rilevazione da parte di una videocamera
La tecnologia 454
Importante:
Cosa succede quando aggiungo ATP?
Importante:
Cosa succede quando aggiungo ATP?
Al posto dell’ATP si usa d-alpha-thio triphosphate (non utilizzata dalla luciferasi)
Il Solexa-Illumina
La tecnologia Solexa, cioè sequencing on-a-chip
•
•
•
•
Circa 100 basi per read
10 milioni di reads per spot
Ridotti costi di sequenziamento
Profondità di sequenziamento
elevata
Video
Overview
• Frammentazione + ligazione adattatori
• Annealing sul chip
• Amplificazione tramite bridge PCR
• Amplificazione tramite bridge PCR
• Formazione di cluster densi e separati
• Ogni cluster contiene una singola sequenza
• Aggiunta di nucleotidi fluorescenti
• Terminatori reversibili (il fluoroforo può essere clivato)
• Lettura di una base per volta, ma in parallelo su molti cluster
Lunghezza dei frammenti raggiunta: 100 basi (in pair-end)
Il SOliD – Sequencing by ligation
• No DNA polimerasi
• Utilizzo dell’enzima ligasi
•
•
•
•
•
Reads di 150 bp massimo (generalmente 35 bp)
Massima accuratezza
Vengono utilizzati sonde con due basi
Ogni base della sequenza viene «letta» due volte
Costi bassi
• Pre-amplificazione con emPCR su microsfere
• Ligazione delle microsfere su un vetrino
Sequencing by Ligation
1. Primers hybridize to the P1 adapter sequence on the templated beads.
2. A set of four fluorescently labeled di-base probes compete for ligation to the sequencing primer.
Specificity of the di-base probe is achieved by interrogating every 1st and 2nd base in each ligation
reaction.
3. Multiple cycles of ligation, detection and cleavage are performed with the number of cycles determining
the eventual read length.
4. Following a series of ligation cycles, the extension product is removed and the template is reset with a
primer complementary to the n-1 position for a second round of ligation cycles.
Lunghezza dei frammenti raggiunta: 35-75 basi
Video
Ion Torrent
•
•
•
•
•
Amplificazione tramite emPCR su microsfere (intrappolate in micropozzetti)
Utilizzo di normali dNTP
L’incorporazione di un dNTP rilascia un protone (H+)
Rilevazione della differenza di voltaggio (ΔV)
ΔV proporzionale al numero di nucleotidi incorporati
Lunghezza dei frammenti raggiunta: oltre 100 basi
Previsto nuovo CMOS chip da 400 basi entro dicembre 2011
Generazione 3
Oxford Nanopore
PacBio – Single Molecule Real Time sequencing (SMRT)
• Sequenziamento a singola molecola di DNA
• Amplificazione iniziale tramite PCR non necessaria
• Riduzione di bias dovuti all’amplificazione
(amplificazione differenziale di alcune parti della sequenza)
Oxford Nanopore
• Tecnologia ancora molto sperimentale
• Vari metodi per determinare i singoli nucleotidi
PacBio RS - SMRT Technology
Filmato SMRT
• Pozzetti con volume 10-21 litri (zeptolitri)
• Aggiunta sequenziale di dNTP marcati con fluorofori
• Determinazione incorporazione tramite ZMW (zero-mode waveguide)
Lunghezza dei frammenti raggiunta: 860-1100bp (esperimenti con circa 2.5 kb)
Qualità dei sequenziamenti
Dalla reazione alla sequenza
Qualità dei sequenziamenti
Sequence: ??????????????????????????????????
Qualità dei sequenziamenti
Come è possibile risalire alla sequenza di DNA sequenziata?
• Necessità di trovare un modo univoco per l’interpretazione dei
cromatogrammi.
E’ possibile ripetere il processo per milioni di volte?
• Necessità di sviluppare algoritmi in grado di assegnare ad ogni picco una
determinata base in modo automatico.
Qualità dei sequenziamenti
Qualità dei sequenziamenti
G
Qualità dei sequenziamenti
G C
Qualità dei sequenziamenti
G C A
Qualità dei sequenziamenti
G C A G
Qualità dei sequenziamenti
…………
G C A G
C
G
A
• Durante l’elettroforesi capillare frammenti di lunghezza diversa
possono non essere ben separati
• Lettura simultanea di basi diverse
• Man mano che il sequenziamento procede verso la fine del
segmento di DNA, la qualità andrà a diminuire (anche
all’inizio)
• Una bassa qualità può significare una sequenza diversa da
quella reale
Base-calling algorithm
Algoritmo che, data una serie di picchi
cromatografici, sia in grado di risalire alla corretta
sequenza di DNA che li ha generati ed assegnare ad
ogni base una certa «qualità»
Phred
E’ l’algoritmo più utilizzato. Ad oggi la
qualità delle sequenze di DNA viene
espressa utilizzando il suo score
Base-calling algorithm
Vengono presi in considerazione i
principali parametri relativi alla forma ed
all’ampiezza dei picchi; in base a questi
viene calcolato un certo valore di qualità
relativo ad ogni base.
Base-calling algorithm
Vengono presi in considerazione i
principali parametri relativi alla forma ed
all’ampiezza dei picchi; in base a questi
viene calcolato un certo valore di qualità
relativo ad ogni base.
La qualità così ottenuta è
strettamente legata alla probabilità
di ottenere quella determinata
base in quel punto del
cromatogramma
Base
A
C
Quality
40
30
Qualità e Probabilità
Q = Qualità (Phred Score)
P = Probabilità di errore
Q = -10log10(P)
P = 10-Q/10
BASES
A
ERROR
QUALITY 20
C
T
G
30
40
50
Qualità e Probabilità
Q = Qualità (Phred Score)
P = Probabilità di errore
Q = -10log10(P)
P = 10-Q/10
BASES
A
ERROR
1%
QUALITY 20
Ad esempio:
una qualità pari a 20 si traduce in una
probabilità di errore dell’1%, mentre una
qualità pari a 30 si traduce in una
probabilità dello 0.1%
C
0.1%
30
T
0.01%
40
G
0.001%
50
Qualità e Probabilità
BASES
A
ERROR
1%
QUALITY 20
Base scartata
C
0.1%
30
T
0.01%
40
G
0.001%
50
Qualità generalmente
accettata
Importante:
Una probabilità d’errore dell’1% può sembrare bassa
Bisogna tenere conto che vengono sequenziate milioni di basi
1% = 10’000 basi errate ogni Mb
Queste basi errate possono essere confuse per mutazioni -> implicazioni cliniche gravi!
Dal sequenziamento al genoma
Dalle reads alle sequenze (più o meno) complete
• Tutte le tecniche di sequenziamento
portano alla produzione di molte sequenze
corte (35-2000 bp)
• Il genoma batterico più corto è circa 1Mb
(500 volte la reads più lunga)
• Un gene batterico è lungo in media 300 bp
• Necessario unire insieme le varie reads per
ottenere un genoma
Trimming, Assembling, Scaffolding