E. Giordano
16/09/2010
1/41
GRUPPO NAZIONALE DI BIOINGEGNERIA
XXIX Scuola Annuale
BIOLOGIA SINTETICA
Bressanone 13 - 17 settembre 2010
COSTITUENTI MOLECOLARI DELLO CHASSIS CELLULARE
Emanuele GIORDANO
II Facoltà di Ingegneria
Dipartimento di Biochimica “G. Moruzzi”
Laboratorio di Ingegneria Cellulare e Molecolare
Università di Bologna – Campus di Cesena
2/41
Bressanone GNB 2010
1
E. Giordano
16/09/2010
3/41
Espressione genica nei batteri e negli eucarioti
Eucariote
Batterio
DNA
Nucleo
RNA polimerasi
RNA
precursore
RNA
polimerasi
Maturazione*
mRNA
polipeptide
ribosoma
Nei batteri, l’mRNA sintetizzato dalla
RNA polimerasi non deve subire una
maturazione prima di essere tradotto
dai ribosomi. Inoltre, dato che non c’è
una membrana nucleare, la traduzione
dell’mRNA può avere inizio mentre la
trascrizione è ancora in corso,
determinando l’accoppiamento della
trascrizione e della traduzione.
citoplasma
Negli eucarioti, il trascritto primario di RNA
è una molecola di mRNA precursore (premRNA), che viene processato nel nucleo
attraverso (*) l’aggiunta di un cappuccio
all’estremità 5’ e di una coda di poli(A)
all’estremità 3’, e la rimozione degli introni.
La traduzione può avvenire solo quando
l’mRNA maturo sia stato trasportato nel
citoplasma.
4/41
Bressanone GNB 2010
2
E. Giordano
16/09/2010
5/41
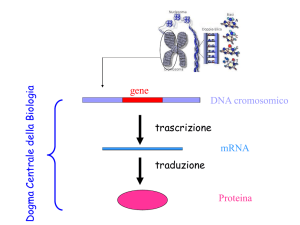
Il flusso dell’informazione contenuta nel DNA
6/41
Il flusso dell’informazione contenuta nel DNA
Bressanone GNB 2010
3
E. Giordano
16/09/2010
7/41
La trascrizione è il
processo di copia
in forma di RNA di
una specifica
sequenza di
interesse (gene) in
un filamento del
duplex di DNA.
8/41
In breve, le
caratteristiche della
molecola di RNA sono:
•singolo filamento;
•ribosio (non
desossiribosio);
•uracile (non timina).
Bressanone GNB 2010
4
E. Giordano
16/09/2010
9/41
Esistono quattro tipi principali di molecole di RNA, ciascuna codificata da un
gene specifico, ma solo uno di essi viene tradotto:
mRNA (RNA messaggero), che codifica per la sequenza di amminoacidi di
un polipeptide. Gli mRNA sono i trascritti dei geni che codificano per
proteine. La traduzione di un mRNA produce un polipeptide.
rRNA (RNA ribosomiale) che, insieme alle proteine ribosomiali, costituisce i
ribosomi – le strutture nelle quali viene tradotto l’mRNA.
tRNA (RNA di transferimento), che porta gli amminoacidi al ribosoma
durante la traduzione.
snRNA (small nuclear RNA; piccolo RNA nucleare) che, insieme a
proteine, forma complessi che vengono usati nella maturazione degli RNA
eucariotici per produrre mRNA funzionali.
10/41
• Diversamente dal DNA, l’mRNA è una molecola instabile, destinata a
un utilizzo temporaneo.
• Le dinamiche della sua sintesi/degradazione determinano la
presenza/assenza nella cellula del prodotto attivo di ogni specifico
gene.
• Il numero di copie di mRNA trascritte (e l’efficienza della loro
successiva traduzione) determina l’abbondanza di tale prodotto.
• La regolazione della trascrizione del DNA è una fase dell’espressione
genica.
Bressanone GNB 2010
5
E. Giordano
16/09/2010
11/41
L’espressione genica
12/41
Promotore, sequenza che codifica per l’RNA e regione del terminatore di un
gene. Il promotore si trova a monte della sequenza codificante, il terminatore a
valle. La sequenza codificante inizia con il nucleotide +1.
Gene
Promotore
Sequenza codificante per l’RNA
Terminatore
Filamento non-stampo
DNA
Filamento stampo
Sito di inizio della
trascrizione
A monte del
gene
Bressanone GNB 2010
Sito di terminazione
della trascrizione
A valle del
gene
6
E. Giordano
16/09/2010
13/41
Il processo di trascrizione.
La doppia elica del DNA
viene denaturata dall’RNA
polimerasi nei procarioti,
(e da altre proteine negli
eucarioti).
Sito di inizio
della trascrizione
RNA polimerasi
Direzione della trascrizione
Filamento non-stampo
Filamento
Promotore
Ibrido DNA-RNA
stampo
L’RNA polimerasi catalizza
quindi la sintesi di una
catena di RNA a singolo
filamento, a partire dal
punto di “inizio della
trascrizione”.
La catena di RNA viene
sintetizzata in direzione 5’3’, utilizzando solo un
filamento del DNA come
stampo che ne stabilisce
la sequenza di basi.
14/41
Il flusso dell’informazione contenuta nel DNA
Bressanone GNB 2010
7
E. Giordano
16/09/2010
15/41
Nei batteri è presente solo un tipo di RNA polimerasi (DNAdipendente), quindi tutte le classi di geni (geni che codificano per
proteine, geni per i tRNA, e geni per gli rRNA) vengono trascritte da
questo enzima. L’inizio della trascrizione di un gene richiede una
forma di RNA polimerasi chiamata oloenzima (o enzima completo).
L’oloenzima è formato dal nucleo enzimatico dell’RNA polimerasi,
costituito da due polipeptidi a, un polipeptide b e uno b’, legato a un
altro polipeptide chiamato fattore sigma (σ). Il fattore sigma assicura
che l’RNA polimerasi si leghi in maniera stabile solo ai promotori.
Direzione della trascrizione
16/41
Direzione della trascrizione
Nella maggior parte dei promotori dei geni di E. coli sono presenti due
sequenze di DNA fondamentali per specificare l’inizio della
trascrizione. Queste sequenze si trovano generalmente in posizione
–35 e –10, cioè 35 e 10 coppie di basi a monte rispetto al punto di
inizio della trascrizione, indicato con +1. La sequenza di consenso
(cioè la sequenza che si trova con frequenza maggiore in ciascuna
posizione) per la regione –35 (la box –35) è 5’-TTGACA-3’. La
sequenza consenso per la regione –10 (chiamata anche Pribnow box)
è 5’-TATAAT-3’.
Bressanone GNB 2010
8
E. Giordano
16/09/2010
17/41
L’RNA polimerasi inizia direttamente la trascrizione [non necessita
della presenza di un innesco (primer)] e polimerizza i nucleotidi in
direzione 5’
3’. Ciò vale qualsiasi sia il filamento stampo nel duplex
a DNA: l’RNA polimerasi puo adoperare come stampo l’uno o l’altro
dei filamenti.
18/41
La reazione chimica catalizzata dall’RNA polimerasi e coinvolta nella sintesi di
RNA utilizzando come stampo il filamento di DNA.
Catena di RNA in allungamento
Filamento di DNA stampo
RNA
polimerasi
Formazione del legame
fosfodiesterico
Ribonucleoside trifosfato
in ingresso
Direzione di
allungamento
(5’-3’)
della catena
Bressanone GNB 2010
Allungamento della catena
9
E. Giordano
16/09/2010
L’azione dell’RNA polimerasi di E. coli nelle fasi di inizio della trascrizione.
19/41
Nella fase di inizio, l’oloenzima dell’RNA polimerasi riconosce dapprima la regione –35 del promotore e quindi lo lega interamente.
Promotore
RNA
polimerasi
Sequenza codificante
Complesso del
promotore chiuso
Fattore σ
Mentre la fase di inizio procede, l’RNA polimerasi si lega più strettamente al promotore a livello della regione –10; lo srotolamento
localizzato del DNA in quella regione accompagna l’evento. A questo punto, l’RNA polimerasi è orientata nel modo corretto per
iniziare la trascrizione in posizione +1.
-35
-10
Nucleotide
d’inizio
Complesso del
promotore aperto
20/41
L’azione dell’RNA polimerasi di E. coli nella fase di allungamento della trascrizione.
Dopo la polimerizzazione di otto/nove nucleotidi, il fattore sigma si dissocia dal nucleo enzimatico.
Direzione della trascrizione
Filamento
stampo
Ibrido DNA-RNA
Fattore σ rilasciato
Mentre l’RNA polimerasi allunga la nuova catena di RNA, l’enzima
srotola il DNA a valle, mantenendo una bolla di trascrizione a
singolo filamento che comprende circa 25 paia di basi. Circa 9 basi
del nuovo RNA sono legate al DNA a singolo filamento nella bolla di
trascrizione, e il rimanente esce dall’enzima sotto forma di filamento
singolo.
RNA in allungamento
Promotore
Bressanone GNB 2010
Sequenza codificante
10
E. Giordano
16/09/2010
21/41
Promotore, sequenza che codifica per l’RNA e regione del terminatore di un
gene. Il promotore si trova a monte della sequenza codificante, il terminatore a
valle. La sequenza codificante inizia con il nucleotide +1.
Gene
Promotore
Sequenza codificante per l’RNA
Terminatore
Filamento non-stampo
DNA
Filamento stampo
Sito di terminazione
della trascrizione
Sito di inizio della
trascrizione
A monte del
gene
A valle del
gene
22/41
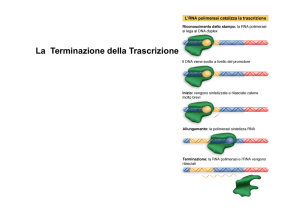
Sequenza di un terminatore (tipo I)
Simmetria bipartita
Stampo
(DNA)
Trascritto
(RNA)
Il trascritto si ripiega
formando la forcina di
terminazione
Bressanone GNB 2010
11
E. Giordano
16/09/2010
23/41
Negli eucarioti, la trascrizione dei geni per i quattro tipi principali di
RNA è effettuata da tre RNA polimerasi differenti.
L’RNA polimerasi I, localizzata nel nucleolo, catalizza la sintesi di tre
degli RNA che si trovano nei ribosomi: le molecole di rRNA 28S, 18S,
e 5,8S. (I valori S indicano il coefficiente di sedimentazione delle
molecole di rRNA durante la centrifugazione, e danno un’indicazione
molto approssimativa del peso molecolare.)
L’RNA polimerasi II, localizzata nel nucleoplasma, sintetizza gli RNA
messaggeri (mRNA) e alcuni piccoli RNA nucleari (snRNA).
L’RNA polimerasi III, che si trova anch’essa nel nucleoplasma,
sintetizza: (1) gli RNA di trasferimento (tRNA); (2) l’rRNA 5S, una
piccola molecola di rRNA presente in tutti i ribosomi; e (3) gli snRNA
non sintetizzati dall’RNA polimerasi II.
Tutte le RNA polimerasi eucariotiche sono costituite da più subunità.
Per esempio, l’RNA polimerasi II del lievito è formata da 12 subunità
24/41
Negli eucarioti inoltre,
il reclutamento
dell’RNA polimerasi
richiede
l’assemblaggio
coordinato e
sequenziale di
numerosi fattori
trascrizionali.
Bressanone GNB 2010
12
E. Giordano
16/09/2010
25/41
A seguito
dell’interazione tra un
fattore di trascrizione
(TF) e la sua
sequenza di consenso,
il TF determina una
distorsione della
topologia del duplex
che lo renderà
accessibile al
complesso
trascrizionale.
26/41
Negli eucarioti l’inizio
della trascrizione
richiede la presenza di
ulteriori, spesso
numerose, proteine
attivatrici.
Bressanone GNB 2010
13
E. Giordano
16/09/2010
27/41
Struttura generale dell’mRNA batterico e delle cellule eucariotiche.
Regione nontradotta in 5’
Regione nontradotta in 3’
Sequenza
codificante
Sito di inizio
della traduzione
Sito di fine della
traduzione
28/41
Successione delle fasi di maturazione dell’mRNA eucariotico (non tutte le fasi sono
necessarie per tutti gli mRNA)
Sequenza codificante
DNA
Promotore
CAP
Trascrizione da parte dell’RNA polimerasi II.
Il cappuccio in 5’ viene aggiunto quando sono
stati sintetizzati 20-30 nucleotidi del pre-mRNA.
Aggiunta della coda di poli(A) in 3’.
Esone
Introne Esone Introne
Esone
Coda di poli(A)
Pre-mRNA
Splicing:
rimozione
degli introni
mRNA
Traduzione (sintesi proteica)
Polipeptide
Bressanone GNB 2010
14
E. Giordano
16/09/2010
29/41
Successione delle fasi di maturazione dell’mRNA eucariotico (non tutte le fasi sono
necessarie per tutti gli mRNA)
Sequenza codificante
DNA
Promotore
CAP
Trascrizione da parte dell’RNA polimerasi II.
Il cappuccio in 5’ viene aggiunto quando sono
stati sintetizzati 20-30 nucleotidi del pre-mRNA.
Aggiunta della coda di poli(A) in 3’.
Esone
Introne Esone Introne
Esone
Coda di poli(A)
Pre-mRNA
Splicing:
rimozione
degli introni
mRNA
Traduzione (sintesi proteica)
Polipeptide
30/41
Successione delle fasi di maturazione dell’mRNA eucariotico (non tutte le fasi sono
necessarie per tutti gli mRNA)
Sequenza codificante
DNA
Promotore
CAP
Trascrizione da parte dell’RNA polimerasi II.
Il cappuccio in 5’ viene aggiunto quando sono
stati sintetizzati 20-30 nucleotidi del pre-mRNA.
Aggiunta della coda di poli(A) in 3’.
Esone
Introne Esone Introne
Esone
Coda di poli(A)
Pre-mRNA
Splicing:
rimozione
degli introni
mRNA
Traduzione (sintesi proteica)
Polipeptide
Bressanone GNB 2010
15
E. Giordano
16/09/2010
31/41
Successione delle fasi di maturazione dell’mRNA eucariotico (non tutte le fasi sono
necessarie per tutti gli mRNA)
Sequenza codificante
DNA
Promotore
CAP
Trascrizione da parte dell’RNA polimerasi II.
Il cappuccio in 5’ viene aggiunto quando sono
stati sintetizzati 20-30 nucleotidi del pre-mRNA.
Aggiunta della coda di poli(A) in 3’.
Esone
Introne Esone Introne
Esone
Coda di poli(A)
Pre-mRNA
Splicing:
rimozione
degli introni
mRNA
Traduzione (sintesi proteica)
Polipeptide
32/41
Alanina
Cisteina
Acido aspartico
Acido glutammico
Fenilalanina
Glicina
Istidina
Isoleucina
Lisina
Leucina
Metionina
Asparagina
Prolina
Glutammina
Arginina
Serina
Treonina
Valina
Triptofano
Tirosina
Bressanone GNB 2010
Ala
Cys
Asp
Glu
Phe
Gly
His
Ile
Lys
Leu
Met
Asn
Pro
Gln
Arg
Ser
Thr
Val
Trp
Tyr
A
C
D
E
F
G
H
I
K
L
M
N
P
Q
R
S
T
V
W
Y
Elenco dei 20
amminoacidi che
vengono utilizzati
durante la sintesi
proteica.
A sinistra il loro nome,
a destra la simbologia
corrispondente a 3 e 1
lettera.
16
E. Giordano
16/09/2010
33/41
Il codice genetico è costituito da 64
codoni. Una tripletta, codificante
per Met, è il segnale che indica
l’inizio del quadro di lettura della
sequenza di mRNA. Tre triplette
che non codificano per alcun
amminoacido
rappresentano
il
segnale di arresto. 60 rimanenti
triplette completano la codifica dei
restanti 19 amminoacidi. Come si
nota quindi, alcuni di essi vengono
specificati da numerose (fino a 6)
triplette distinte. Il codice è
universale, cioè condiviso da tutte
le specie viventi. Tuttavia non è
affatto detto che tutti i codoni per
uno
specifico
amminoacido
vengano utilizzati con la stessa
probabilità di occorrenza in ogni
specie.
34/41
I tRNA hanno il ruolo di far
corrispondere alle triplette (codoni)
dell’mRNA il corretto amminoacido,
mediante la corrispondenza con il
loro anticodone.
Bressanone GNB 2010
17
E. Giordano
16/09/2010
35/41
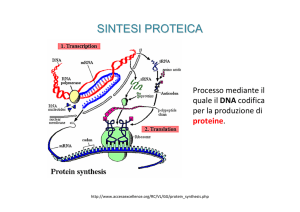
La sintesi dei polipeptidi
avviene nel ribosoma, un
organello che ogni cellula
possiede a migliaia.
Il ribosoma lega gli mRNA e
ne facilita l’interazione con i
tRNA per consentire la sintesi
di una catena polipeptidica.
36/41
Una caratteristica sequenza
della regione non-tradotta in 5’
dell’mRNA (Shine-Dalgarno,
RBS = ribosome binding site),
posizionata 8-12 nucleotidi
prima della tripletta (codone)
di inizio AUG, indirizza il
corretto legame della subunità
minore
del
ribosoma
all’mRNA.
Bressanone GNB 2010
18
E. Giordano
16/09/2010
37/41
38/41
Bressanone GNB 2010
19
E. Giordano
16/09/2010
39/41
La formazione del legame peptidico tra due amminoacidi viene
catalizzata dall’attività peptidiltransferasica ribosomiale
40/41
Bressanone GNB 2010
20
E. Giordano
16/09/2010
41/41
Bressanone GNB 2010
21







![Lezione 12 Trascrizione e maturazione RNA [modalità compatibilità]](http://s1.studylibit.com/store/data/004956814_1-03488078208fbae40d6f98c42ec7fd9f-300x300.png)