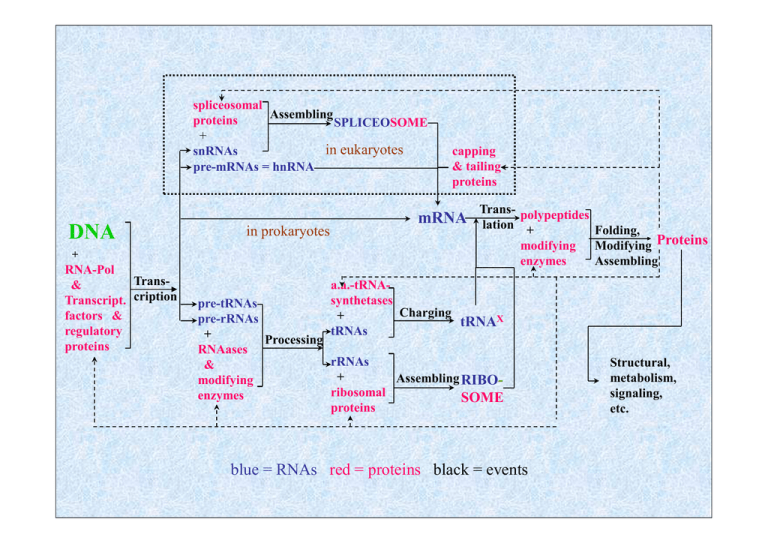
spliceosomal
Assembling
proteins
SPLICEOSOME
+
in eukaryotes
snRNAs
pre-mRNAs = hnRNA
DNA
+
RNA-Pol
Trans&
Transcript. cription
factors &
regulatory
proteins
in prokaryotes
capping
& tailing
proteins
mRNA
Trans- polypeptides
lation +
Folding,
modifying Modifying Proteins
Assembling
enzymes
a.a.-tRNAsynthetases
Charging
+
tRNAX
tRNAs
pre-tRNAs
pre-rRNAs
+
Processing
RNAases
rRNAs
&
+
Assembling RIBOmodifying
ribosomal
enzymes
SOME
proteins
blue = RNAs red = proteins black = events
Structural,
metabolism,
signaling,
etc.
La regolazione nei procarioti
I geni batterici sono tipicamente raggruppati in operoni, in cui più geni si
susseguono l’un l’altro preceduti da un unico promotore e trascritti insieme in un
unico RNA policistronico.
Il primo operone di cui è stata studiata la regolazione a livello molecolare è
l’operone del lattosio di Escherichia coli. Esso consiste di tre geni (LacZ, LacY,
LacA) posti di seguito, sotto il controllo di un unico promotore, i quali codificano tre
proteine coinvolte nel metabolismo del lattosio: la β-galattosidasi, una permeasi e
una transacetilasi. La permeasi facilita l’ingresso del lattosio nella cellula, la βgalattosidasi catalizza la scissione del lattosio in galattosio e glucosio, che poi
intraprendono la loro via metabolica, la transacetilasi ha una funzione a tutt’oggi
non chiara. I tre geni sono preceduti da un promotore inducibile (che cioè permette
la trascrizione dell’operone soltanto in risposta ad opportuni stimoli ambientali – in
questo caso la presenza del lattosio). Nella regolazione della trascrizione di questo
operone sono implicate due proteine regolatrici, il lac-repressor (codificato dal gene
LacI) e la cAMP-receptor-protein CRP (codificata dal gene crp) detta anche CAP
(catabolite activator protein).
β-galactosidase
H
+
Following the β-Galactosidase Reaction. The galactoside substrate X-Gal
produces a colored product on cleavage by β-galactosidase. The appearance of
this colored product provides a convenient means for monitoring the amount of the
enzyme both in vitro and in vivo.
I = lac-repressor
Z = β-Galactosidase
Y = Permease
A = Transacetylase
The DNA footprinting technique.
(A) This technique requires a DNA
molecule that has been labeled at one
end. The protein shown binds tightly to
a specific DNA sequence that is seven
nucleotides long, thereby protecting
these seven nucleotides from the
cleaving agent. If the same reaction
were performed without the DNAbinding protein, a complete ladder of
bands would be seen on the gel (not
shown). (B) An actual footprint used to
determine the binding site for a human
protein that stimulates the transcription
of specific eucaryotic genes. These
results locate the binding site about 60
nucleotides upstream from the start site
for RNA synthesis. The cleaving agent
was a small, iron-containing organic
molecule that normally cuts at every
phosphodiester bond with nearly equal
frequency.
Negative control of the lac operon
The i gene encodes a repressor which, in the absence of lactose (top),
binds to the operator (o) and blocks transcription of the three structural
genes (z, β-galactosidase; y, permease; and a, transacetylase).
Lactose induces expression of the operon by binding to the repressor
(bottom), which prevents the repressor from binding to the operator.
P = promoter; Pol = polymerase.
+1
The nearly palindromic operator sequence
M
Complex of both DNA fragments
with the repressor protein
Complex of the longer DNA fragment
with the repressor protein
Longer DNA fragment containing
the primary operator binding site
for the lac repressor
Shorter DNA fragment containing
the secondary operator binding
site for the lac repressor
Increasing amounts of repressor
Electrophoretic Mobility Shift Assay (EMSA)
M: corsia dei marcatori, vari
frammenti di DNA di lunghezze
note che servono da riferimento
per i frammenti allo studio
Positive control of the lac operon by glucose
Low levels of glucose activate adenylyl cyclase,
which converts ATP to cyclic AMP (cAMP).
Cyclic AMP then binds to the catabolite activator
protein (CAP) and stimulates its binding to
regulatory sequences of various operons concerned
with the metabolism of alternative sugars, such as
lactose. CAP interacts with the a subunit of RNA
polymerase to activate transcription.
L’operone del triptofano
La regolazione dell’operone del triptofano (che contiene i geni per cinque enzimi
necessari alla biosintesi di questo amminoacido) è basata su due meccanismi:
il primo implica la presenza di una proteina repressore che viene attivata solo dalla
presenza di triptofano, con cui interagisce in modo utile a legarsi alla sequenza
bersaglio sul promotore dell’operone bloccando la trascrizione,
Il secondo meccanismo (l’attenuazione) è esclusivo dei procarioti in quanto opera
in modo concertato tra trascrizione e traduzione (vedi oltre).
The binding of tryptophan to the tryptophan repressor protein changes the
conformation of the repressor.
The conformational change enables this gene regulatory protein to bind tightly to a
specific DNA sequence (the operator), thereby blocking transcription of the genes
encoding the enzymes required to produce tryptophan (the trp operon). The threedimensional structure of this bacterial helix-turn-helix protein, as determined by xray diffraction with and without tryptophan bound, is illustrated. Tryptophan binding
increases the distance between the two recognition helices in the homodimer,
allowing the repressor to fit snugly on the operator.
Mechanism of transcriptional attenuation The trp mRNA is translated while still being synthesized. In
the presence of high levels of tryptophan, the ribosomes proceed along the message slightly behind the site
of transcription. Under these conditions, the mRNA regions designated 3 and 4 hybridize to form a stemloop structure that signals the termination of transcription. In the presence of low levels of tryptophan,
however, the ribosomes stall at region 1 of the mRNA, which contains two adjacent codons for tryptophan.
In this case, since region 2 is not bound to a ribosome, it is free to form an alternative stem-loop structure
by hybridizing to region 3. This hybridization prevents formation of the 3 4 stem loop, and transcription is
able to continue past the attenuator sequence.
Leader peptide for attenuation of Trp operon
Leader peptides for attenuation of Thr, Phe and His operons
Il batteriofago λ
E’ un batteriofago che infetta Escherichia coli.
E’ costituito dal genoma di DNA lineare a doppio filamento lungo circa
48500 paia di basi avvolto dentro un capside proteico su cui è innestata
una coda, le cui proteine terminali servono a riconoscere la membrana
della cellula infettabile al cui interno inietta il suo DNA, che subito dopo
assume la forma circolare covalentemente chiusa.
Successivamente, a seconda delle condizioni della cellula infetta può adire
al ciclo litico, che porta alla produzione di numerose particelle fagiche
figlie che si liberano all’esterno provocando la lisi del batterio, oppure può
adire al ciclo lisogeno, in cui il suo genoma si integra in quello del batterio
e rimane quiescente per un numero indefinito di replicazioni batteriche, per
poi deintegrarsi in risposta a determinate condizioni ambientali e
riprendere il ciclo litico.
La doppia vita del
batteriofago λ
tR
tL
sib
About 10 less well known genes
Il primo evento dopo l’infezione,
ovvero l’iniezione del DNA fagico
nella cellula di E. coli, è la
circolarizzazione del genoma
mediante i terminali cos
(cohesive ends).
I primi geni trascritti sono N e cro. La proteina N è un antiterminatore che consente
all’RNA polimerasi batterica di ignorare i segnali di terminazione tl , tr1 e tr2, e di
esprimere anche le proteine CII, O, P, Q, CIII, xis e int.


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)







![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)
