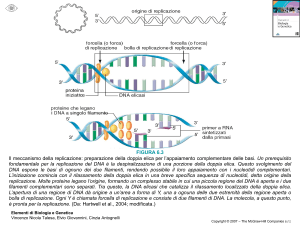
STRUTTURA DEL DNA
www.fisiokinesiterapia.biz
Watson e Crick
lastra fotografica
fonte di
raggi X
R. Franklin
M. Wilkins
DNA
Diffrazione con raggi X del DNA
•XWatson e Crick proposero il loro modello di struttura del
DNA in base ai risultati di Franklin and Wilkins sulla
diffrazione con raggi X del DNA ed a quelli di Chargaff - nelle
cellule quantità di adenina = timina e quantità di guanina =
citosina -
Composizione degli acidi nucleici
DNA (acido deossiribonucleico)
1)
Acido fosforico
2)
Deossiribosio
3)
Basi azotate
a)
Adenina
Purine
b)
Guanina
c)
Citosina
Pirimidine
d)
Timina
Zuccheri
OH
ribosio
RNA
H
2-deossiribosio
DNA
Basi azotate
PURINE
Adenina (A)
Guanina (G)
PIRIMIDINE
Uracile (U)
Timina (T)
Citosina (C)
Il DNA è un polinucleotide
che contiene A, G, T e C
L’ RNA è un polinucleotide
che contiene A, G, U e C
Nucleotidi
Nucleotidi purinici
fosfato
adenina
guanina
deossiribosio
Deossiadenosina 5’-monofosfato (dAMP)
Deossiguanina 5’-monofosfato (dGMP)
Nucleotidi pirimidinici
citosina
Deossicitosina 5’-monofosfato (dCMP)
timina
Deossitimina 5’-monofosfato (dTMP)
Singolo filamento
terminale 5’
legame
fosfodiesterico
terminale 3’
Legami covalenti fosfodiesterici
tra deossiribosi e gruppi fosfati
determinano la formazione del
singolo filamento
I terminali del filamento sono
differenti ( 5’ e 3’)
Accoppiamenti tra basi azotate
Base adenina-timina
(due legami idrogeno)
Timina
Adenina
deossiribosio
deossiribosio
Base guanina-citosina
(tre legami idrogeno)
Citosina
deossiribosio
Guanina
deossiribosio
Doppio filamento
I due filamenti
polinucleotidici sono
complementari e
antiparalleli
Le basi sono situate
perpendicolarmente
all’asse del doppio
filamento
Doppia elica
mattoni del DNA
fosfato
base
filamento di DNA
nucleotide
zucchero
doppio filamento di DNA
doppia elica di DNA
scheletro
zucchero-fosfato
basi appaiate
legami idrogeno
basi appaiate
legami idrogeno
continua
Doppia elica
asse elica
O
P
Misure della doppia elica:
H
basi appaiate
(C e N)
diametro 2 nm
giro elica 3.4 nm
base 0.34 nm
C
solco
maggiore
10 basi/1 giro elica
solco
minore
(1 nm =1 x 10-9 m)
modello molecolare
diagramma stilizzato
Caratteristiche principali della doppia elica
1.
Due catene polinucleotidiche avvolte in senso destrorso.
2.
Catene polinucleotidiche antiparallele:
5’ → 3’
3’ ← 5’
3.
Gli scheletri zucchero-fosfato sono all’esterno della doppia elica, le basi
sono orientate verso l’asse centrale.
4.
Le coppie di basi complementari sono legate insieme da legami deboli
idrogeno. A si appaia con T (2 legami H), G con C (3 legami H).
5’-TATTCCGA-3’
3’-ATAAGGCT-5’
5.
Una coppia di basi occupa 0.34 nm, un giro completo dell’elica occupa 3.4
nm (10 basi/giro).
6.
Gli scheletri zucchero-fosfato non sono egualmente spaziati, formando così
un solco maggiore ed uno minore.
doppia elica
doppia elica
“collana di perle”
nucleosoma
Compattamnto
della doppia elica
in cromosoma
fibra di 30 nm
domini ad ansa
cromosoma
metafasico
istone H1
istone ottamerico
Compattamento della doppia elica in
cromosoma
istone ottamerico
DNA
istone H1
DNA
istone H1
nucleosoma
istone ottamerico
cntinua
Compattamento della doppia elica in cromosoma