Dott. Antonio Sartini - 22/04/2011
La leucina ed i suoi metaboliti, ruolo nella sintesi e nella degradazione proteica, il
razionale d’uso nell’integrazione alimentare.
La perdita di massa magra corporea (Lean Body Mass - LBM) può inficiare sia la
funzionalità dell’organismo che la salute. In condizioni normali si può osservare perdita di
LMB con l’avanzare dell’età, con un regime alimentare inadeguato, con la mancanza di
attività fisica ed anche negli atleti in condizioni di sovrallenamento (over-training)1.
Condizioni patologiche come il cancro o l’HIV possono portare a perdita di LMB causata
da depauperamento muscolare, stessa cosa si osserva nella convalescenza postchirurgica. Interessante notare che l’allenamento alla forza e/o programmi e strategie di
integrazione nutrizionale hanno dimostrato di controbilanciare queste condizioni di
catabolismo muscolare.2 Quindi esiste un equilibrio dinamico tra segnali anabolici (di
sintesi) e segnali catabolici (di degradazione) nel processo che porta al guadagno o alla
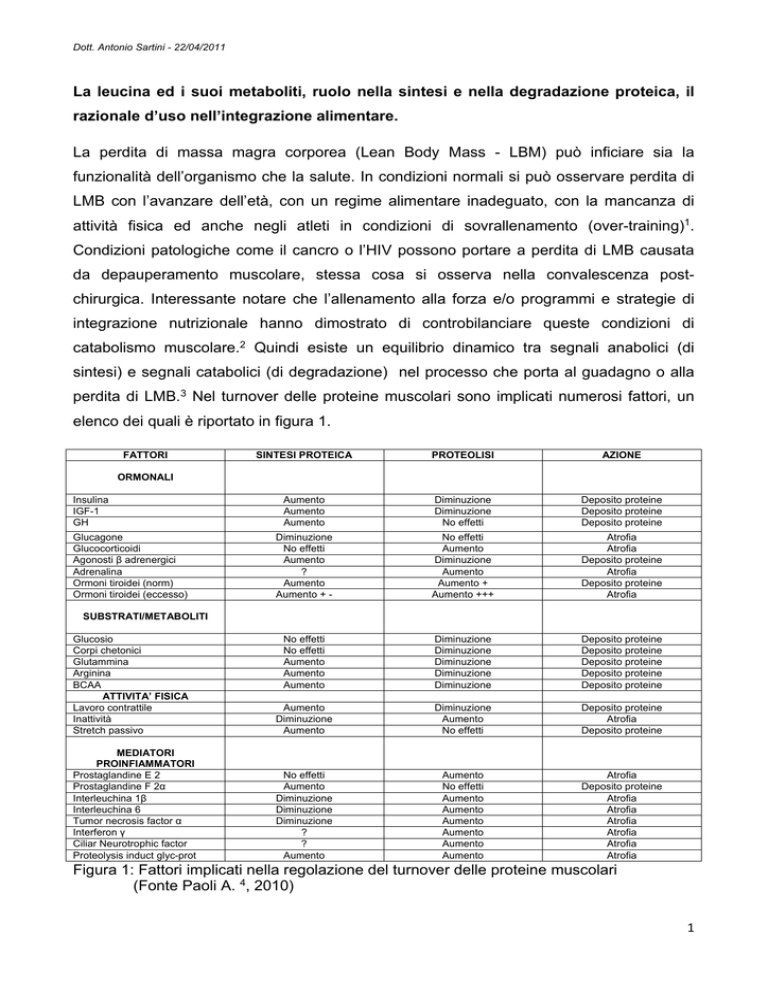
perdita di LMB.3 Nel turnover delle proteine muscolari sono implicati numerosi fattori, un
elenco dei quali è riportato in figura 1.
FATTORI
SINTESI PROTEICA
PROTEOLISI
AZIONE
Aumento
Aumento
Aumento
Diminuzione
Diminuzione
No effetti
Deposito proteine
Deposito proteine
Deposito proteine
Diminuzione
No effetti
Aumento
?
Aumento
Aumento + -
No effetti
Aumento
Diminuzione
Aumento
Aumento +
Aumento +++
Atrofia
Atrofia
Deposito proteine
Atrofia
Deposito proteine
Atrofia
ORMONALI
Insulina
IGF-1
GH
Glucagone
Glucocorticoidi
Agonosti β adrenergici
Adrenalina
Ormoni tiroidei (norm)
Ormoni tiroidei (eccesso)
SUBSTRATI/METABOLITI
Glucosio
Corpi chetonici
Glutammina
Arginina
BCAA
ATTIVITA’ FISICA
Lavoro contrattile
Inattività
Stretch passivo
No effetti
No effetti
Aumento
Aumento
Aumento
Diminuzione
Diminuzione
Diminuzione
Diminuzione
Diminuzione
Deposito proteine
Deposito proteine
Deposito proteine
Deposito proteine
Deposito proteine
Aumento
Diminuzione
Aumento
Diminuzione
Aumento
No effetti
Deposito proteine
Atrofia
Deposito proteine
MEDIATORI
PROINFIAMMATORI
Prostaglandine E 2
Prostaglandine F 2α
Interleuchina 1β
Interleuchina 6
Tumor necrosis factor α
Interferon γ
Ciliar Neurotrophic factor
Proteolysis induct glyc-prot
No effetti
Aumento
Diminuzione
Diminuzione
Diminuzione
?
?
Aumento
Aumento
No effetti
Aumento
Aumento
Aumento
Aumento
Aumento
Aumento
Atrofia
Deposito proteine
Atrofia
Atrofia
Atrofia
Atrofia
Atrofia
Atrofia
Figura 1: Fattori implicati nella regolazione del turnover delle proteine muscolari
(Fonte Paoli A. 4, 2010)
1 Dott. Antonio Sartini - 22/04/2011
La risposta cronica adattativa all’esercizio fisico contro-resistenza promuove un
incremento delle dimensioni muscolari e della forza. L’attivazione delle fibre muscolari per
produrre forza portano ad una conseguente cascata di eventi che includono risposte
ormonali ed immunitarie, stimolazione di pathway di segnalazione della cellula muscolare
ed infine, incremento della sintesi proteica5 6. Globalmente, questa risposta promuove un
accrescimento delle proteine muscolari ed aumenta le capacità di produzione della forza.
Anche se l’esercizio fisico contro-resistenza in stato di digiuno aumenta la sintesi di
proteine, il bilancio proteico netto rimane negativo se non vengono forniti nutrienti7 . In
particolare, sembra che l’assunzione di amino acidi essenziali indirizza un incremento
netto della sintesi proteica muscolare dopo esercizio fisico contro-resistenza8. Gli
aminoacidi essenziali (Essential Amino Acids – EAA) svolgono due funzioni critiche per la
sintesi proteica:
1) Gli EAA sono substrati per la sintesi proteica perché la translazione dell’mRNA non
può progredire a meno che l’intera varietà di amino acidi non sia immediatamente
disponibile, il che sottolinea l’importanza dell’assunzione di EAA (quelli cioè che
non possono essere sintetizzati De Novo),
2) Gli EAA sono segnali per la sintesi proteica.
Diversi studi recenti hanno messo in evidenza il ruolo deli EAA come segnali indipendenti
per l’iniziazione della traduzione9
10 11
. Per esempio, l’assunzione di EAA prima e dopo
esercizio fisico contro –resistenza potenzia l’attivazione esercizio-indotta della proteina
ribosomiale S6 chinasi 70-kDa, un enzima importante per aumentare l’efficienza
traduzionale e la sintesi proteica dopo esercizio12
13
. Ulteriormente sembra che la leucina
abbia un ruolo primario e non-insulino dipendente nello stimolare questa via metabolica14.
Diversi studi hanno verificato che diete ad alto tenore proteico stimolano la sintesi proteica
ed alcuni dei benefici promossi da questo tipo di alimentazione sulla composizione
corporea possono essere attribuiti ad un elevato consumo di amino acidi a catena
ramificata ( Branched Chain Amino Acids – BCAA) che includono la Leucina (Leu), la
Valina (Val) e la Isoleucina (Ile) 15 16 17. In accordo con questa ipotesi, un elevato consumo
di proteine non incrementa solo la disponibilità dei substrati (aminoacidi) per la sintesi
proteica, ma stimola anche i processi anabolici osservati dopo elevato consumo di
proteine18.
2 Dott. Antonio Sartini - 22/04/2011
La leucina in particolare ha dimostrato di modulare il tasso di sintesi proteica, in particolar
modo stimolando l’attività delle proteine coinvolte nel processo di traduzione, che è
cruciale nelle cellule per il controllo della sintesi proteica19
20 21 22 23
. Questa modulazione
può coinvolgere l’attivazione di specifici pathway intracellulari coinvolti a loro volta nella
sintesi proteica e che includono l’attivazione della via metabolica del “bersaglio nei
mammiferi della rapamicina (mTOR)”. E’ stato osservato che l’mTOR risponde
immediatamente a qualsiasi modificazione nel consumo di aminoacidi e di energia24
25
.Quando la scorta aminoacidica, in particolar modo la leucina, è elevata, l’mTOR
interagisce con altre proteine per formare complessi proteici in modo da fosforilare i loro
componenti-chiave, che sono a turno responsabili per la traduzione dell’mRNA in proteine.
Questi complessi includono il legame con il fattore di iniziazione eucariotico 4E nella
regione cap; l’attivazione tramite fosforilazione della della proteina ribosomiale 70kDa- S6
kinasi-1(S6K1); e l’elungazione del fattore eIF226
27
. D’altronde, la deplezione del pool di
aminoacidi intracellulare, o la loro rimozione dal mezzo di comunicazione extracellulare
provoca la soppressione della sintesi proteica28. Comunque, come la leucina moduli questi
eventi molecolari non è stato ancora del tutto chiarito29.
Tuttavia, nell’ultimo decennio si sono consolidati gli studi sul ruolo metabolico della leucina
e dei suoi metaboliti nello stimolare la crescita proteica e di conseguenza l’incremento
della massa muscolare. La L-leucina (Leu), rientra molto spesso nelle formulazioni di
integratori alimentari utilizzati nello sport, ma anche in alimenti speciali per l’infanzia, per il
sostentamento negli stati di malnutrizione e, non ultimo, nelle formulazioni per la nutrizione
artificiale, enterale e parenterale. Una delle principali conseguenze del riconoscimento del
ruolo di protagonista della leucina come segnale non-insulino dipendente nella sintesi
proteica è l’aumento della proporzione con cui viene inserita nelle formulazioni alimentari.
Mentre per decenni il suo rapporto rispetto agli altri BCAA, valina (Val) e isoleucina(Ile), è
sempre stato “2:1:1” , in cui la leucina veniva introdotta con proporzioni doppie rispetto agli
altri ramificati, ora è possibile vedere integratori nutrizionali con rapporti “8:1:1” o alimenti
speciali specificatamente “fortificati” con aggiunta di leucina. Non ultimo, si vedono ormai
in commercio, quali principi nutritivi a se stanti o in formulazioni varie, i metaboliti della
leucina quali il α-Keto-iso-caproato (KIC) o il β-idrossi-β-metilbutirrato(HMB).
Scopo di questo lavoro è quindi approfondire i meccanismi biologici molecolari che stanno
alla base del razionale d’uso di integratori nutrizionali contenti Leucina e/o i suoi metaboliti,
Kic e HMB.
3 Dott. Antonio Sartini - 22/04/2011
Il METABOLISMO DELLA LEUCINA
Differentemente dagli altri aminoacidi che vengono degradati nel fegato, i BCAA (leucina,
isoleucina e valina), allo stesso modo dell’alanina, del glutammato e dell’aspartato,
vengono ossidati nel muscolo scheletrico30. Tra i tre BCAA la leucina ha un marcato e più
elevato tasso di ossidazione se paragonata a valina e isoleucina. Gli enzimi coinvolti nel
catabolismo della leucina includono le aminotransferasi per aminoacidi a catena ramificata
(BCAT) mitocondriali e citosoliche ed il complesso enzimatico chetoacido a catena
ramificata deidrogenasi (BCKDH)31. Dalla reazione catalizzata dal BCAT, che è reversibile,
l’aminoacido è transaminato e convertito nel rispettivo chetoacido, il α-keto-iso-caproato
(KIC). Simultaneamente a questa reazione, si verifica la conversione dell’α – ketoglutarato
(un accettore di azoto derivato dai BCAA) in glutammato. Questa reazione è in grado di
promuovere la sintesi di altri aminoacidi, come l’alanina e la glutammina. Dopo la reazione
catalizzata dal BCAT, il chetoacido subisce la decarbossilazione ossidativa, una reazione
irreversibile mediata dal BCKDH. Questo complesso enzimatico si trova sulla faccia
interna della membrana mitocondriale. Da questa reazione il KIC è convertito
nell’isovaleril-CoA che è ossidato da due differenti deidrogenasi, generando acetil-CoA e
aceto acetato come prodotto finale32. Si veda la figura n. 2
Fig. 2 Il metabolismo della Leucina. Adattato da Smith, Marks e Liberman (2005) 33
4 Dott. Antonio Sartini - 22/04/2011
Nei mammiferi il complesso BCKDH presenta tre enzimi: l’α-chetoacido decarbossilasi
(E1), costituito dalle subunità 2α e 2β, la diidrolipoil-transacetilasi (E2) e la diidrolipoildeidrogenasi (E3)34. Nei tessuti, l’attività di questo complesso è regolata da un ciclo di
fosforilazione e de fosforilazione catalizzate da specifiche chinasi e fosfatasi. Lo stato di
fosforilazione è controllato dall’attività della BCKDH chinasi(BCKDHK). Questo enzima
pruomuove l’inattivazione del complesso BCKDH attraverso la fosforilazione dell’unità
E1α. L’attivazione del complesso BCKDH può essere ottenuta rapidamente tramite
l’inibizione del BCKDHK promossa dal chetoacido della leucina o dai suoi analoghi
strutturali tra i quali l’octanoato, l’α-cloroisocaproato e l’acido clorofibrico. Il processo di de
fosforilazione è invece mediato dalla BCKDH fosfatasi (BCKDHP) che è resposabile
dell’attivazione del complesso BCKDH che avviene de fosforilando la sub-unità E1α
35 36
.
L’attività di questo complesso è influenzata dagli ormoni, dal diabete, dall’esercizio, dal
digiuno, da acidosi o diete ipoproteiche, ed è dipendente dal tessuto37.
La conversione reversibile della leucina in KIC avviene nelle cellule muscolari ad opera
delle citate BCAT mitocondriali e citosoliche. Una volta prodotto il KIC viene escreto dal
muscolo e trasportato nel fegato dove è trasformato per il 95 % dalla BCKDH
mitocondriale in Isovaleril-coA. Approssimativamente il 5% di tutta la L-leucina del KIC
prodotto viene metabolizzato a HMB da un enzima citosolico chiamato KIC-diossigenasi .
Circa il 10-40 % dell’HMB prodotto viene escreto con le urine il restante viene trasformato
in HMG-coA38 39.
Fig. 3 Fonte : Nissen SL, Abumrad NN ( 1997)
5 Dott. Antonio Sartini - 22/04/2011
La leucina ha dimostrato di stimolare la sintesi delle proteine muscolari e di attenuare la
proteolisi muscolare. L’effetto della leucina è mimato dal KIC, tuttavia sembra che questo
effetto dipenda dalla riconversione del KIC in Leucina
40
. Viceversa alcuni dei benefici
attribuiti alla leucina sono stati attribuiti alla conversione della stessa in HMB41 42 43 44.
Infatti l’HMB avrebbe di essere anticatabolico ed efficace nel l’attenuare l’atrofia muscolare
durante lo stress da esercizio fisico45
46 47
, in certi tipi di cancro
48 49
, nello scompenso
cardiaco congestizio, nella sepsi e nell’HIV50 51.
EFFETTO PARADOSSO DELLA LEUCINA
Una dieta sbilanciata contenete elevate concentrazioni di leucina di solio provoca una
riduzione dei livelli corporei di valina ed isoleucina. Questo fenomeno è conosciuto come
“Leucine
paradox”.
Questa
situazione
potrebbe
essere
parzialmente
spiegata
dall’attivazione dell’ossidazione dei BCAA, tramite il complesso BCKDH, che porta ad una
regolazione negativa del BCKDHK, cocomitante ad un effetto inibitorio del KIC su questo
enzima. Questi due fattori promuovono l’attivazione della BCKDH, provocando deplezione
di chetoacidi a partire da altri aminoacidi, soprattutto valina ed isoleucina. D’altra parte si
dovrebbe tener conto che la somministrazione di leucina stimola la sintesi proteica ed
inibisce
la
degradazione
di
proteine.
Entrambe
queste
situazioni
influenzano
indirettamente i livelli di valina ed isoleucina52
CONTROLLO DELLA SINTESI PROTEICA
La sintesi proteica nelle cellule eucariotiche è principalmente regolata nel passaggio di
inizio e di elongazione della traduzione, tramite i fattori di inizio eIF2 ed eIF453 54 55 56 57 .
Ci sono due punti di controllo nell’inizio della traduzione :
1. Il legame del metionil-tRNA alla sub unità ribosomiale 40S. Questo processo è
regolato dal fattore eucaristico di iniziazione 2 (eIF2) ed inibito quando eIF2 è
fosforilato sulla sub unità α.
2. Legame del mRNA alla sub unità 43S. Questo processo è stimolato dall’attivazione
del bersaglio della rapamicina nei mammiferi (mTOR) che fosforilando la proteina di
legame per eIF4E (4EPB1), permette la dissociazione di eIF4E, che è libero di
unirsi con il eIF4G per formare la forma attiva del complesso eIF4F, permettendo il
legame con il cappuccio 5’ di metilguanosina (5’-cap) .
6 Dott. Antonio Sartini - 22/04/2011
Uno schema riassuntivo della sintesi delle proteine o traduzione è riportato in fig. 4
Fig. 4 Schema riassuntivo della traduzione dell’informazione genica
(fonte : http://www.biocarta.com/pathfiles/h_eifpathway.asp)
Le cellule eucariotiche richiedono almeno 12 fattori di inizio, per un totale di oltre 25
catene polipeptidiche. Parecchi di questi eIF (ad es. eIF1, eIF2 ed eIF3) legano la sub
unità 40S, preparando la stessa sub unità a legarsi all’mRNA. Anche il tRNAi iniziatore,
legato alla metionina,si attacca alla sub unità 40S prima di interagire con l’mRNA. Il tRNA
si inserisce nel sito P della sub unità minore (40S) in associazione con eIF2-GTP. Una
volta realizzati queti eventi, la sub unità minore del ribosoma con i suoi fattori di inizio
7 Dott. Antonio Sartini - 22/04/2011
associati ed il tRNA caricato (che insieme costituiscono il complesso di pre-inizio 43S) , è
pronta per cercare l’estremità 5’ dell’mRNA, che porta il cappuccio di metilguanosina. Il
complesso 43S viene inizialmente indirizzato verso l’mRNA con l’aiuto di un gruppo di
fattori di inizio che sono già legati all’mRNA. Tra questi fattori: eIF4E si lega al cappuccio
all’estremità 5’ dell’mRNA eucariotico; eIF4A si muove lungo l’estremità de5’ del
messaggio rimuovendo tutte le regioni a doppio filamento che interferirebbero con lo
scorrimento del del complesso 43S lungo l’mRNA; eIF4G serve da collegamento tra
l’estremità 5’ incappucciata e quella 3’ poliadenilata dell’mRNA, convertendo un mRNA
lineare in un messaggio circolare. Una volta che il complesso 43S si è legato all’estremità
5’ dell’mRNA, il complesso scorre lungo il messaggio finchè raggiunge una sequenza
riconoscibile di nucleotidi (tipicamente 5’-CCACCAUGC-3’) che contiene il codone di inizio
AUG. Una volta che il complesso 43S ha raggiunto il codone AUG appropriato, eIF2-GTP
viene idrolizzato, eIF2-GDP ( e altri eIF associati) viene rilasciato e la sub unità maggiore
(60S) si unisce al complesso per completare la fase di inizio58.
INFLUENZA DELLA LEUCINA SUL MECCANISMO DI SINTESI PROTEICA
L’effetto anabolico della leucina sulla sintesi proteica è di grande interesse nutrizionale per
attenuare la perdita di massa magra che si verifica in stati catabolici ed in condizioni
fisiologiche specifiche come l’invecchiamento59. Sono stati condotti molti studi per chiarire
questo effetto, come precedentemente osservato, la leucina, oltre a servire da diretto
substrato per la sintesi proteica, può contribuire ulteriormente stimolando questo processo,
esercitando un effetto positivo sulla fosforilazione delle proteine coinvolte nella fase
iniziale della traduzione dell’mRNA60.
La leucina esercita un effetto sulla regolazione a breve termine delle a livello della
traduzione nella sintesi proteica. Questo effetto è sinergico con l’insulina, che è un ormone
anabolico con un ruolo chiave nel mantenimento della sintesi proteica muscolare.
Ciononostante, l’insulina da sola non è in grado di ripristinare completamente il tasso di
sintesi proteica nel muscolo nella fase di post-assorbimento, richiedendo l’assunzione di
amino acidi o proteine per ristabilire completamente l’effetto anabolico61 62.
La leucina ha anche un ruolo insulino-secretagogo. L’assunzione orale di Leucina provoca
un lieve e transitorio incremento dei livelli sierici di insulina, che stmola anche la sintesi
proteica indotta da questo amino acido63
64
. Sebbene i livelli basali di insulina sono
importanti a potenziare l’effetto mediato dalla leucina65, questo aminoacido agisce come
8 Dott. Antonio Sartini - 22/04/2011
un segnale nutritivo che regola la sintesi proteica negli adipociti, allo stesso modo che in
altre cellule, attraverso un meccanismo non-insulino-dipendente66. Questo risultato può
essere la conseguenza di una azione diretta (effetto della leucina) o indiretta (ad opera di
metaboliti della leucina come il KIC) sul tessuto adiposo, come è stato già osservato in
cellule isolate. In aggiunta, questi effetti sono stati osservati in studi in vitro e in vivo con
l’infusione di norleucina un composto analogo alla leucina che non stimola la secrezione di
insulina, ne è incorporato nella struttura proteica67 68 69.
Ci sono specifiche vie metaboliche che favoriscono le sintesi proteiche, come la crescita e
la proliferazione cellulare, come il pathway mediato dalla proteina mTOR. Questa via
metabolica è stimolata da mitogeni, inclusi insulina e BCAA ma in particolar modo dalla
leucina70. Comunque il meccanismo che è alla base di tale effetto non è del tutto chiaro.
La via di amplificazione del segnale inizia con il legame dell’insulina al suo recettore. Una
volta attivato il recettore dell’insulina fosforila la tirosina di molti substrati proteici. Alcuni di
questi substrati appartengono alla famiglia di substrati dei recettori dell’insulina, le proteine
di attracco IRS71
72
. La fosforilazione delle proteine IRS per la tirosina crea siti di
riconoscimento per per un certo numero di molecole come l’enzima fosfatidil-inositol-3chinasi (PI 3-K). Si verifica una stretta associazione tra questo enzima ed il substrato
recettore dell’insulina-1 (IRS-1)73. La proteina bersaglio per questo enzima è la Akt/PKB,
che è responsabile per la regolazione del trasporto del glucosio. Nei tessuti periferici la
fosforilazione dell’Akt/PKB promuove un certo numero di effetti come la traslocazione del
trasportatore di glucosio tipo 4(GLUT-4) verso la membrana plasmatica, la regolazione
della sintesi proteica, il metabolismo cellulare e la progressione del ciclo cellulare.74
La leucina stimola la sintesi proteica anche modulando elementi coinvolti nella via di
segnalazione metabolica dell’insulina tramite PI3-K (vedi fig. 5). Pertanto questo amino
acido inibisce il segnale dell’insulina75, possibilmente attivando l’mTOR, che stimola la
fosforilazione dell’IRS-1 e di conseguenza riduce l’attività del PI3-K76.
Inoltre, questa proteina riduce anche l’azione inibitoria del complesso di sclerosi tuberosa
(TSC1/TSC2). L’inibizione di questo complesso permetta l’attivazione della proteina
chinasi Rheb (Ras homologue-enriched in brain), che conduce alla fosforilazione
dell’mTOR ed alla attivazione della della proteina ribosomiale S6, favorendo la sintesi del
RNA, di proteine ribosomi ali e di fattori di elongazione77.
La leucina esplica effetti anche sulla regolazione della proteina S6K1, sulla proteina 1
legante il fattore di iniziazione eucariotico 4E (4E-BP1), che sono coinvolti nella fase di
inizio della traduzione proteica.78
9 Dott. Antonio Sartini - 22/04/2011
La leucina stimola l’attività dell’mTOR che fosforila e quindi inibisce la 4E-BP1. La
dissociazione del complesso 4E-BP1 promuove la formazione del fattore di iniziazione
eucaristico 4E (eIF4E) che è di importanza cruciale per l’iniziazione del processo di
traduzione. Inoltre la leucina stimola un secondo fattore di iniziazione il 4G (eIF4G)
attraverso il una via metabolica indipendente dall’mTOR. L’associazione di questi due
fattori, il eIF4E e il eIF4G, forma il complesso ribosomiale eIF4F, che regola l’iniziazione
della traduzione79
80
. Quindi la leucina è in grado di stimolare la sintesi di complessi che
sono responsabili per il processo di traduzione delle proteine e di conseguenza aumenta
la capacità cellulare di sintesi proteica81 82.
Fig. 5 Regolazione della via metabolica dell’mTOR tramite insulina
e leucina (fonte: Hinault et all. 2006)
Per quanto finora riportato l’mTOR gioca un ruolo cruciale nella regolazione della sintesi
delle proteine muscolari. La proteina TOR è una serina/treonina chinasi ed è un membro
della famiglia delle chinasi fosfatidilinositol chinasi correlate (PIKK)83. L’mTOR esiste in
10 Dott. Antonio Sartini - 22/04/2011
due distinti complessi proteici, il complesso mTOR -1 (mTORC1) ed il complesso mTOR2(mTORC2)84 85 86 87. L’mTORC1 regola il tasso di sintesi proteica e di crescita cellulare in
modo rapamicina dipendente88
89
(vedi fig. 6). Il complesso mTORC 1 è formato
dall’mTOR, dal Raptor,mLST8(GβL),PRAS40 e dal FKBP38
90 91
. La proteina di struttura
Raptor recluta i substrati per l’mTOR ed è essenziale affinché possa esercitare la sua
attività fisiologica92
93
. La proteina mLST8 lega il dominio della mTOR chinasi attivandone
l’attività indipendentemente da Raptor94, anche se non sembra sia indispensabile per
l’attività del mTORC1. La proteina PRAS40 agisce come regolatore negativo di mTORC1
sia legando direttamente il domino chinasico quindi inibendo l’attività chinasica95, sia
associandosi con Raptor inibendone la capacità di attivazione96
97 98
. L’insulina esplica il
suo effetto inibitorio sul mTORC1 fosforilando il PRAS40 che attivato esercita il suo effetto
inibitorio. Anche la proteina FKBP38 esplica attività inibitrica sul mTORC 1. Una sovra
espressione di FKBP38 inibisce la fosforilazione della S6K1, dellaS6 e della 4EPBP199.
Il substrato proteico di mTORC1 più caratterizzato sono le S6K1 e la 4EBP1 che è in
grado di fosforilare
100 101
. Fosforilando la S6K1 si ha un incremento della biogenesi
ribosomiale. Il 4EBP1 è un repressore traduzionale che, se fosforilato, non esrcita la sua
azione inibitrice sulla traduzione. Infatti se defosforilato il 4EBP1 è in grado di legare e
quindi di inibire il eIF4E. Quando l’mTOR fosforila il 4EBP1, libera il eIF4E che è in grado
di dare inizio alla traduzione.
La S6K1 è in grado di stimolare anche la fase di elongazione della traduzione, fosforilando
e quindi inattivando la eEF2chinasi che a sua volta quando è attiva fosforila inibendola, il
fattore eucariotico di elongazione 2(eEF2)102.
La Rheb (Res-homolog enriched in brain) è una piccola GTPasi che agisce come
regolatore positivo dell’mTORC1 ed è indispensabile per l’attività dell’mTOR. La S6K1 e la
4EBP1 non possono essere fosforilate se manca la Rheb mentre una sovra espressione
della stessa stimola la fosforilazione e quindi l’attività di S6K1 e 4EBP1103 104 105.
Infine la TSC1/TSC2 integrano una varietà di segnali quali quelli dell’insulina, dei fattori di
crescita e dello stato di energia ed agiscono come regolatori negativi dell’mTORC1106
11 Dott. Antonio Sartini - 22/04/2011
Figura 6
Regolazione dell’mTORC1. Fonte: Kim (2009)107
Sono stai studiate le ipotesi che la leucina influisse sull’mTORC1 tramite un sistema
recettoriale di membrana o tramite un meccanismo specifico di assorbimento tuttavia allo
stato attuale non ci sono evidenze che possano supportare tale possibilità. L’inibizione del
sistema
di
trasporto
degli
L
aminoacidi
non
inibisce
l’attività
leucina
indotta
dell’mTORC1108. E’ stato anche ipotizzato un meccanismo di rigonfiamento della cellula
aminoacido-indotto. Infatti alcuni aminoacidi entrano nella cellula tramite trasportatori Na+ dipendente, l’accumulo di Na+ e di aminoacidi intracellularmente aumenterebbe
l’osmolarità109 . Il rigonfiamento cellulare ha dimostrato essere già da solo uno stimolo per
la sintesi proteica ed esercita una azione inibitoria sulla degradazione proteica110, tuttavia
il più efficace trasportatore di leucina, il sistema trasportatore di L aminoacidi è Na+-
12 Dott. Antonio Sartini - 22/04/2011
indipendente. Tutto ciò suggerisce che sia la concentrazione aminoacidica intracellulare o
un recettore intracellulare a regolare la via metabolica di segnalazione del mTORC 1111.
E’ stato inoltre proposto un meccanismo di attivazione dell’mTORC1 tramite i vari
metaboliti della leucina incluso l’ATP. Infatti la leucina può essere metabolizzata a KIC,
come già riferito, e finire a produrre ATP nel ciclo di Krebs, allo stesso tempo stimola
anche la glutammato-deidrogenasi (GDH)112con un risultato simile. La deplezione di ATP
inibisce la fosforilazione del S6K1 e del 4EBP1113
114
. C’è da dire però che la
concentrazione di ATP non viene influenzata da una stimolazione o da privazione di amino
acidi a breve termine115.
Anche trattamento con un inibitore delle amino trasferasi reversibili(BCAT) l’acido amino
acetico (AOAA) inibisce la fosforilazione del della S6K1 nel trattamento con leucina delle
cellule β del pancreas116.
Come già riportato il metabolita KIC è in grado di fosforilare il 4EBp1 in assenza di leucina
anche se in maniera minore rispetto alla leucina ed la sua attività viene inibita dalla
presenza di AOAA. Inoltre la norleucina ,che è un analogo della leucina ma non viene
incorporata nella sintesi proteica, è efficace tanto quanto la leucina nel fosforilare la
4EBP1, questo suggerisce che potrebbe esserci uno specifico recettore intracellulare117.
Fig. 6 Meccanismi proposti per l’azione della L-Leu su mTORC1. Fonte: Kim(2009)
13 Dott. Antonio Sartini - 22/04/2011
Sebbene non sia ancora conosciuto l’esatto meccanismo appare ovvio che gli amino acidi
regolano l’attività dell’mTORC1 e quindi la ricerca si è concentrata su questo tipo di
regolazione. La presenza di aminoacidi non cambiano l’interazione tra Rheb-Raptor o tra
Rheb-mTOR118, né l’interazione tra mLST8-mTOR è influenzata dagli aminoacidi.
Anche la presenza o meno di TSC1/TSC2 non è rilevante ai fini della regolazione
dell’mTORC1119
120
. Tra i meccanismi proposti quello che sembra essere più interessante
è quello secondo cui gli aminoacidi agirebbero modulando l’interazione tra mTOR e
Raptor. Infatti la privazione di aminoacidi causa un legame più stretto tra mTOR e Raptor,
ciò inibisce l’attività chinasica dell’mTOR, mentre la presenza di aminoacidi stimola tale
attività allentando il legame tra mTOR e Raptor121.
Rimangono comunque da investigare ipotesi che coinvolgono diversi altri tipi di proteine.
Ultimamente l’interesse della ricerca farmaceutica è rivolto ad un altro metabolita della
leucina il già citato HMB.
L’HMB interrompe due vie metaboliche di degradazione proteica come riportato in fig. 7
Fig. 7 Azione anticatabolica dell’HMB (Fonte: www.abbotnutrition.com)
La perdita di massa magra (LBM) associata a malattie croniche, a malattie acute, a
patologie oncologiche o a emorragie croniche è il risultato dell’aumento nella circolazione
di citochine infiammatorie o di segnali metabolici che aumentano la degradazione
proteica122
123 124
. Per esempio il TNF –α (Tumor necrosis factor), l’INF –γ (interferone γ),
l’AngII (Angiotensina II), il LPS (Lipopolisaccaride) o il PIF (Proteolysis-inducing factor). La
cascata infiammatoria attivata dalle citochine attiva il fattore nucleare kB(NFkB) il quale a
14 Dott. Antonio Sartini - 22/04/2011
sua volta, accumulandosi nel nucleo della cellula muscolare, è in grado di aumentare la
produzione di proteosomi, enzimi che, a loro volta, incrementano la degradazione proteica.
Inoltre il TNF-α, l’INF-γ, l’Ang-II e la LPS attivano la CASPASI-8 che a sua volta attiva la
CASPASI-3 riducendo la sintesi proteica nel nucleo. L’HMB è in grado di inibire la
CASPASI 8 125 126.
L’HMB esercita anche un’azione diretta sulla sintesi proteica attivando l’mTOR. Viene
sostenuta l’ipotesi che l’HMB sia il metabolita attivo della Leucina che attiva la via
metabolica dell’mTOR 127. Tuttavia questo meccanismo deve ancora essere chiarito.
Da quanto si è potuto ricavare da una analisi della letteratura reperibile, il razionale d’uso
di integratore nutrizionali a base di L-leucina trova giustificazione nelle evidenze
scientifiche che riguardano l’azione di questo amino acido sulla sintesi proteica. Tuttavia è
ancora prematuro considerare una certezza l’esatto meccanismo di azione sulla via
metabolica del mTORC1. Comunque che sia la leucina la più importante tra gli aminoacidi
essenziali non sembra esserci dubbio, tuttavia la sua capacità di stimolare la sintesi
proteica beneficia di un effetto sinergico degli altri aminoacidi anche se sembrerebbe in
quantità e proporzioni minori di quanto si è sempre presupposto.
Il KIC a sua volta sembra agire in virtù del fatto che può essere reversibilmente
transaminato a Leucina e quindi avendo una azione equivalente, anche se leggermente
minore, per una applicazione alimentare andrebbe indagato soprattutto in funzione della
biodisponibilità e del costo di produzione.
L’HMB infine è quello che sta subendo la maggiore spinta industriale farmaceutica sembra
avere una azione maggiore sulla sintesi proteica rispetto alla leucina ma sicuramente è più
vicino al concetto di farmaco che non di nutriente. Essendo un metabolita prodotto in
bassa percentuale dal metabolismo della leucina, sarebbe giusto interrogarsi su quanto
sia logico e sicuro integrare qualcosa di cui l’organismo stesso ne limita la produzione.
Antonio Sartini
15 Dott. Antonio Sartini - 22/04/2011
BIBLIOGRAFIA
Fleck SJ, Kraemer WJ: Designing Resistance Training Programs. Champaign, IL: Human Kinetics
Publishers, 2004.
2
Kraemer WJ, Ratamess NA: Fundamentals of resistance training: Progression and exercise prescription.
Med Sci Sports Exerc 2004; 36:674-688.
3
Spiering BA, Kraemer WJ, Anderson JM, et al: Resistance exercise biology: Manipulation of resistance
exercise programme variables determines the responses of cellular and molecular signalling pathways.
Sports Med 2008; 38:527-540.
4
Paoli A. Neri M.: Principi di metodologia del fitness. Editrice Elika, Cesena 2010.
5
Guttridge DC. Signaling pathways weigh in on decisions to make or break skeletal muscle. Curr Opin Clin
Nutr Metab Care.
2004;7(4):443–50.
6
Hayashi AA, Proud CG. The rapid activation of protein synthesis by growth hormone requires signaling
through mTOR. Am JPhysiol Endocrinol Metab. 2007;292(6): E1647–55.
7
Rasmussen BB, Phillips SM. Contractile and nutritional regulation of human muscle growth. Exerc Sport Sci
Rev. 2003; 31(3):127–31.
8
Wolfe RR. Effects of amino acid intake on anabolic processes.Can J Appl Physiol. 2001;26(Suppl):S220–7.
9
Blomstrand E, Eliasson J, Karlsson HK, Kohnke R. Branched-chain amino acids activate key enzymes in
protein synthesis after physical exercise. J Nutr. 2006;136(1 Suppl):269S–73S.
10
Bohe J, Low A, Wolfe RR, Rennie MJ. Human muscle protein synthesis is modulated by extracellular, not
intramuscular amino acid availability: a dose–response study. J Physiol. 2003;552 (Pt 1):315–24.
11
Bohe J, Low JF, Wolfe RR, Rennie MJ. Latency and duration of stimulation of human muscle protein
synthesis during continuous infusion of amino acids. J Physiol. 2001;532(Pt 2):575–9.
12
Blomstrand E, Op. cit. n. 9 13
Baar K, Esser K. Phosphorylation of p70(S6k) correlates with increased skeletal muscle mass following
resistance exercise. Am J Physiol. 1999;276(1 Pt 1):C120–7.
14
KRAEMER, W. J., D. L. HATFIELD, J. S. VOLEK, M. S. FRAGALA, J. L. VINGREN, J. M. ANDERSON, B.
A. SPIERING, G. A.THOMAS, J. Y. HO, E. E. QUANN, M. IZQUIERDO, K. HAKKINEN, and C. M. MARESH.
Effects of Amino Acids Supplement on Physiological Adaptations to Resistance Training. Med. Sci. Sports
Exerc., Vol. 41, No. 5, pp. 1111–1121, 2009
15
DONATO, J. J.; PEDROSA, R. G.; TIRAPEGUI, J. Conseqüências da avaliação do peso corporal e da
suplementação de L-leucina e L-fenilalanina na composição corporal e em parâmetros metabólicos em
ratos. Rev. Bras. Cienc. Farm., v.40, p.124-127, 2004. 16
LAYMAN, K. L.; WALKER, D. A. Potential importance of leucine in treatment of obesity and the metabolic
syndrome. J. Nutr., v. 136, p.319s-323s, 2006.
17
KATSANOS, C. S.; KOBAYASHI, H.; SHEFFIELD-MOORE, M.; AARSLAND, A.; WOLFE, R. R. A high
proportion of leucine is required for optimal stimulation of the rate of muscle protein synthesis by essential
amino acids in the elderly. Am. J. Physiol. Endocrinol. Met., v.291, p.381- 387, 2006
18
CAMPOS, P. L.; LUZ, S. S.; RIBEIRO, S. M. L.; TIRAPEGUI, J.; LANCHA Jr., A. H. Importância dos
aminoácidos de cadeia ramificada: considerações sobre o metabolismo de proteínas e energia. Rev. Bras.
Nutr. Clin., v.14, p.18-30, 1999. 19
ANTHONY, J. C.; ANTHONY, T. G.; KIMBALL, S. R.; JEFFERSON, L. S. Oral administration of leucine
stimulates ribosomal protein mRNA translation but not global rates of protein synthesis in the liver of rats. J.
Nutr., v.131, p.1171-1176, 2001.
20
LYNCH, C. J.; HUTSON, S. M.; PATSON, B. J. Tissue-specific effects of chronic dietary leucine and
norleucine supplementation on protein synthesis in rats. Am. J. Physiol. Endocrinol. Metab., v.283, p.824835, 2002a.
21
CROZIER, S. J.; KIMBALL, S. R.; EMMERT, S. W.; ANTHONY, J. C.; JEFFERSON, L. S. Oral leucine
administration stimulates protein synthesis in rat skeletal muscle. J. Nutr., v.13593, p.376-382, 2005.
22
GARLICK, P. J. The role of leucine in the regulation of protein metabolism. J. Nutr., v.135, p.1553S1556S, 2005.
1
16 Dott. Antonio Sartini - 22/04/2011
23
KIMBALL, S. R.; JEFFERSON, L. S. Signaling pathways and molecular mechanisms through which
branched-chain amino acids mediate translational control of protein synthesis. J. Nutr., v.136, p.227-231,
2006.
24
BRUHAT, A.; AVEROUS, J.; CARRARO, V.; ZHONG, C.; REIMOLD, A. M.; KILBERG, M. S.,
AFOURNOUX, P. Differences in the molecular mechanisms involved in the transcriptional activation of the
CHOP and asparagines synthetase genes in response to amino acid deprivation or activation of the unfolded
protein response. J. Biol. Chem., v.277, p.48107-18414, 2002.
25
ASNAGHI, L.; BRUNO, P.; PRIULLA, M.; NICOLIN, A. mTOR: a protein kinase switching between life and
death. Pharmacol. Res., v.50, p.545-549, 2004. 26
FINGAR, D. C.; BLENIS, J. Target of rapamycin TOR: an integrator of nutrient and growth factor signals
and coordinator of cell growth and cell cycle progression. Ontogeny, v.23, p.3151-3171, 2004.
27
HAY, N.; SONENBERG, N. Upstream and downstream of mTOR. Genes. Dev., v.18, p.1926-1945, 2004.
28
MALONEY, C. A.; REES, W. D. Gene-nutrient interactions during fetal development. Reproduction, v.130,
p.401-410, 2005. 29
D. Vianna, G. Fullin Resende Teodoro, F. L. Torres-Leal, J. Tirapegui B J P S vol. 46, n. 1, jan./mar.,
2010;
30
ROSSI, L.; TIRAPEGUI, J. Aminoácidos de cadeia ramificada e atividade física. In: TIRAPEGUI, J.
Nutrição, metabolismo e suplementação na atividade física. São Paulo: Editora Atheneu, 2005. p.153-161. 31
SHIMOMURA, Y.; MURAKAMI, T.; NAGASAKI, M.; HONDA, T.; GOTO, H.; KOTAKE, K.;KUROKAWA, T.;
NONAMI, T. Regulation of branched-chain amino acid metabolism and pharmacological effects of branchedchain amino acids. Hepatol. Res., v.30, p.3-8, 2004.
32
Anthony et al., 2001 Op. cit. n.19;
33
SMITH, C.; MARKS, A.; LIBERMAN, M. Mark´s Basic Medical Biochemistry – A Clinical approach. (2.ed.)
Baltimore: Lippincott Williams & Wilkins, 2005. p.712- 730. 34
HARRIS, R. A.; PAXTON, R.; POWELL, S. M.; GOODWIN, G. W.; KUNTZ, M. J.; HAN, A. C. Regulation of
branched-chain alpha-ketoacid dehydrogenase complex by covalent modification. Adv. Enzyme. Regul.,
v.25, p.219-237, 1986.
35
Anthony et al., 2001 Op. cit. n.19; 36
SHIMOMURA, Y.; HARRIS, R. A. Metabolism and physiological function of branched-chain amino acids:
discussion of session 1. J. Nutr., v.136, p.232S-233S, 2006. 37
ARAUJO, Jr. J.; FALAVIGNA, G.; ROGERO, M. M.; PIRES, I. S. O.; PEDROSA, R. G.; CASTRO, I. A.;
TIRAPEGUI, J. Effect of chronic supplementation with branched–chain amino acids on the performance and
hepatic and muscle glycogen content in trained rats. Life Sci., v.79, p.1343- 1348, 2006. 38
Sabourin PJ, Bieber LL: Formation of b-hydroxyisovalerate by an α ketoisocaproate
oxygenase in human liver. Metabolism 1983, 32:160-164. 39
Nissen SL, Abumrad NN: Nutritional role of the leucine metabolite β-hydroxy-β-methylbutyrate (HMB).
J.Nutr Bioch 1997, 8:300-311.
40
Fox, Heather L., Phuong T. Pham, Scot R. Kimball, Leonard S. Jefferson, and Christopher J. Lynch.
Amino acid effects on translational repressor 4E-BP1 are mediated primarily by L-leucine in isolated
adipocytes. Am. J. Physiol. 275 (Cell Physiol. 44): C1232–C1238, 1998.
41
Hider RC, Fern EB, London DR: Relationship between intracellular amino acids and protein synthesis in
the extensor digitorum longus muscle of rats. Biochem J 1969, 114:171-8.
42
Nissen S, Sharp R, Ray M, et al: Effect of leucine metabolite β-hydroxy-β-methylbutyrate on muscle
metabolism during resistance-exercise training. J Appl Physiol 1996, 81:2095-2104
43
Nissen SL, Abumrad NN: Nutritional role of the leucine metabolite β-hydroxy-β-methylbutyrate (HMB). J
Nutr Biochem 1997, 8:300-311.
44
Caperuto EC, Tomatieli RV, Colquhoun A, et al: β-Hydoxy-b-methylbutyrate supplementation affects
Walker 256 tumor-bearing rats in a timedependent manner. Clin Nutr 2007, 26:117-122.
45
Nissen S, Sharp R, Ray M, et al. Op.cit. n.42 46
Panton LB, Rathmacher JA, Baier S, et al: Nutritional supplementation of the leucine metabolite βHydroxy-β-Methylbutyrate (HMB) during resistance training. Nutrition 2000, 16:734-739
47
Jówko E, Ostaszewski P, Jank M, et al: Creatine and beta-hydroxy-betamethylbutyrate (HMB) additively
increase lean body mass and muscle strength during a weight-training program. Nutrition 2001, 17:558-566.
48
Caperuto EC, Tomatieli RV, Colquhoun A, et al. Op. cit. n. 44; 49
Nunes EA, Kuczera D, Brito GA, et al: Beta-hydroxy-beta-methylbutyrate supplementation reduces tumor
growth and tumor cell proliferation ex vivo and prevents cachexia in Walker 256 tumor-bearing rats by
modifying nuclear factor-kappaB expression. Nutr Res 2008, 28:487-493. 17 Dott. Antonio Sartini - 22/04/2011
Clark RH, Feleke G, Din M, et al: Nutritional treatment for acquired immunodeficiency virus-associated
50
wasting using beta-hydroxy betamethylbutyrate,glutamine, and arginine: a randomized, double-blind,
placebo-controlled study. J Parenter Enteral Nutr 2000, 24:133-139.
51
Eley HL, Russel ST, Tisdale MJ: Attenuation of depression of muscle protein synthesis induced by
lipopolysaccharide, tumor necrosis factor,and angiotensin II by β-hydroxy-β-methylbutyrate. Am J Physiol
Endocrinol Metab 2008, 295:1409-1416.
52
Shimomura, Y Harris, RA (2006) OP. cit. n. 36; 53
Dever T.E. (1999) Translation initiation: adept at adapting. Trends Biochem. Sci. 24, 398-403.
54
Gingras AC et al. (1999) eIF4 initiation factors: effectors of mRNA recruitment to ribosomes and regulators
of translation. Annu. Rev. Biochem. 68, 913-963.
55
Gray, N.K. and Wickens, M. (1998) Control of translation initiation in animals. Annu. Rev. Cell Dev. Biol.
14, 399-458.
56
Palfrey, H.C. and Nairn, A.C. (1995) Calcium-dependent regulation of protein synthesis. Adv. Second
Messenger Phosphoprotein Res. 30, 191-223.
57
Rhoads, R.E. (1999) Signal transduction pathways that regulate eukaryotic protein synthesis. J. Biol.
Chem. 274, 30337-30340
58
Karp G Biologia cellulare e molecolare- 3° edizione EdiSES-Napoli,2009
59
STIPANUK, M. H. Leucine and protein synthesis: mTOR and Beyond. Nut. Rev., v.65, p.122-129, 2007.
60
ROGERO, M. M.; TIRAPEGUI, J. Aspectos atuais sobre aminoácidos de cadeia ramificada e exercício
físico. Rev. Bras. Cien. Farm., v.44, p.536-575, 2008
61
ROGERO, M. M.; TIRAPEGUI, J.(2008) Op. cit.n.60 62
BOLSTER, D. R.; KIMBALL, S. R.; JEFFERSON, L. S. Translation control mechanisms modulate skeletal
muscle gene expression during hypertrophy. Exerc. Sport. Sci. Rev., v.31, p.111-116, 2003.
63
ROGERO, M. M.; TIRAPEGUI, J.(2008) Op. cit.n.60 64
BOLSTER, D. R.; KIMBALL, S. R.; JEFFERSON, L. S.(2003) Op. cit. n. 63 65
STIPANUK, M. H. (2007) Op.cit. n. 59 66
LYNCH, C. J.; PATSON, B. J.; ANTHONY, J.; VAVAL, A.; JEFFERSON, L. S.; VARY, T. C. Leucine is a
direct-acting nutrient signal that regulates protein synthesis in adipose tissue. Am. J. Physiol. Endocrinol.
Metab., v.283, p.503- 153, 2002b.
67
Anthony et al., 2001 Op. cit. n.19; 68
LYNCH, (2002a), Op.cit.n. 20 69
LYNCH, (2002b), Op.cit.n. 66 70
COTA, D.; PROULX, K.; SMITH, K. A. B.; KOZMA, S. C.; THOMAS, G.; WOODS, S. C.;SEELEY, R. L.
Hypothalamic mTOR signaling regulates food intake. Science, v.312, p.927-930, 2006.
71
ZICK, Y. Uncoupling insulin signalling by serine/threonine phosphorylation: a molecular basis for insulin
resistance. Biochem. Soc. Trans., v.32, p.812-816, 2004.
72
GUAL, P.; LE MARCHND-BRUSTEL, Y.; TANTI, J. F. Positive and negative regulation of insulin signaling
through IRS-1 phosphorylation. Biochimie, v.87, p.99-109, 2005
73
KIM, Y. B.; NIKOULINA, S. E.; CIARALDI, T. P.; HENRY, R. R.; KAHN, B. B. Normal insulin-dependent
activation of Akt/protein kinase B, with diminished activation of phosphoinositide 3-kinase, in muscle in type
2 diabetes. J. Clin. Invest., v.104, p.733-741, 1999.
74
GEORGAKIS, G. V.; YOUNES, A. From rapa nu rapamicina: targeting PI3K/Akt/mTOR for cancer therapy.
Expert. Rev. Anticancer Ther., v.6, p.131-140, 2006.
75
KREBS, M.; KRSSAK, M.; BERNROIDER, E.; ANDERWALD, C.; BREHM, A.; MEYERSPEER, M.;
NOWOTNY, P.; ROTH, E.; WALDHÄUSL, W.; RODEN,M. Mechanism of amino acid-induced skeletal
muscle insulin resistance in humans. Diabetes, v.51, p.599-605, 2002.
76
KIMBALL, S. R.; SHANTZ, L. M.; HORETSKY, R. L.; JEFFERSON, L. S. Leucine regulates translation of
specific mRNAs in L6 myoblasts through mTOR-mediated changes in availability of eIF4E and
phosphorylation of ribosomal protein S6. J. Biol. Chem., v.274, p.11647-11652, 1999.
77
YANG, O.; GUAN, K. L. Expanding mTOR signaling. Cell Res., v.17, p.666-681, 2007. 78
KIMBALL, S. R et all.(1999) Op. cit. n.76 79
DONATO, J. J.; PEDROSA, R. G.; ARAÚJO, J. A. J.; PIRES, I. S. O.; TIRAPEGUI, J. Effects of leucine
and phenylalanine supplementation during intermittent periods of food restriction and refeeding in adults rats.
Life Sci., v.81, p.31-39, 2007.
80
STIPANUK, M. H. (2007) Op.cit. n. 59 81
KIMBALL, S. R et all.(1999) Op. cit. n.76 82
DONATO, J. J et all. Op. cit. n. 79 83
Schmelzle T & Hall MN (2000). TOR, a central controller of cell growth. Cell 103:253-262.
18 Dott. Antonio Sartini - 22/04/2011
Hara K, Maruki Y, Long X, Yoshino K, Oshiro N, Hidayat S, Tokunaga C, Avruch J & Yonezawa K (2002).
84
Raptor, a binding
partner of target of rapamycin (TOR), mediates TOR action. Cell 110:177-189.
85
Kim DH, Sarbassov DD, Ali SM, King JE, Latek RR, Erdjument- Bromage H, Tempst P & Sabatini DM
(2002). mTOR interacts with raptor to form a nutrient-sensitive complex that signals to the cell growth
machinery. Cell 110:163-175.
86
Jacinto E, Loewith R, Schmidt A, Lin S, Ruegg MA, Hall A & Hall MN (2004). Mammalian TOR complex 2
controls the actin cytoskeleton and is rapamycin insensitive. Nat Cell Biol 6:1122-1128.
87
Sarbassov DD, Ali SM, Kim DH, Guertin DA, Latek RR, Erdjument-Bromage H, Tempst P & Sabatini DM
(2004). Rictor, a novel binding partner of mTOR, defines a rapamycin-insensitive and raptor-independent
pathway that regulates the cytoskeleton. Curr Biol 14:1296-1302.
88
Fingar DC, Salama S, Tsou C, Harlow E & Blenis J (2002). Mammalian cell size is controlled by mTOR
and its downstream
targets S6K1 and 4EBP1/eIF4E. Genes Dev 16:1472-1487.
89
Hay N & Sonenberg N (2004). Upstream and downstream of mTOR. Genes Dev 18:1926-1945.
90
Bai X, Ma D, Liu A, Shen X, Wang QJ, Liu Y & Jiang Y (2007). Rheb activates mTOR by antagonizing its
endogenous inhibitor,
FKBP38. Science 318:977-980.
91
Yang Q & Guan KL (2007). Expanding mTOR signaling. Cell Res17:666-681
92
Hara K, Maruki Y, Long X, Yoshino K, Oshiro N, Hidayat S, Tokunaga C, Avruch J & Yonezawa K (2002).
Raptor, a binding
partner of target of rapamycin (TOR), mediates TOR action. Cell 110:177-189.
93
Kim et all. (2002) Op.cit. n. 85 94
Kim DH, Sarbassov DD, Ali SM, Latek RR, Guntur KV, Erdjument-Bromage H, Tempst P & Sabatini DM
(2003). GbetaL,
a positive regulator of the rapamycin-sensitive pathway required for the nutrient-sensitive interaction
between raptor and mTOR. Mol Cell 11:895-904.
95
Vander Haar E, Lee SI, Bandhakavi S, Griffin TJ & Kim DH (2007). Insulin signalling to mTOR mediated by
the Akt/PKB substrate PRAS40. Nat Cell Biol 9:316-323.
96
Oshiro N, Takahashi R, Yoshino K, Tanimura K, Nakashima A, Eguchi S, Miyamoto T, Hara K, Takehana
K, Avruch J, Kikkawa U & Yonezawa K (2007). The proline-rich Akt substrate of 40 kDa (PRAS40) is a
physiological substrate of mammalian target of rapamycin complex 1. J Biol Chem 282:20329-20339.
97
Sancak Y, Peterson TR, Shaul YD, Lindquist RA, Thoreen CC, Bar-Peled L & Sabatini DM (2008). The
Rag GTPases bind raptor and mediate amino acid signaling to mTORC1. Science
320:1496-1501.
98
Wang L, Harris TE, Roth RA & Lawrence JC Jr (2007). PRAS40 regulates mTORC1 kinase activity by
functioning as a direct inhibitor of substrate binding. J Biol Chem 282:20036-20044.
99
Bai X, Ma D, Liu A, Shen X, Wang QJ, Liu Y & Jiang Y (2007). Rheb activates mTOR by antagonizing its
endogenous inhibitor, FKBP38. Science 318:977-980.
100
Fingar et all. (2002); Op. cit. n. 88 101
Hay N & Sonenberg N (2004); Op.cit. n. 89 102
Redpath NT, Foulstone EJ & Proud CG (1996). Regulation of translation elongation factor-2 by insulin via
a rapamycin-sensitive signalling pathway. EMBO J 15:2291-2297.
103
Zhang Y, Gao X, Saucedo LJ, Ru B, Edgar BA & Pan D (2003). Rheb is a direct target of the tuberous
sclerosis tumour suppressor proteins. Nat Cell Biol 5:578-581.
104
Inoki K, Li Y, Xu T & Guan KL (2003a). Rheb GTPase is a direct target of TSC2 GAP activity and
regulates mTOR signaling. Genes Dev 17:1829-1834.
105
Tee AR, Manning BD, Roux PP, Cantley LC & Blenis J (2003). Tuberous sclerosis complex gene
products, Tuberin and Hamartin, control mTOR signaling by acting as a GTPase-activating protein complex
toward Rheb. Curr Biol 13:1259-1268.
106
Inoki K, Zhu T & Guan KL (2003b). TSC2 mediates cellular Energy response to control cell growth and
survival. Cell 115:577-590.
107
Kim E (2009) Mechanisms of amino acid sensing in mTOR signaling pathway Nutrition Research and
Practice, 3(1), 64-71
108
Lynch CJ, Fox HL, Vary TC, Jefferson LS & Kimball SR (2000). Regulation of amino acid-sensitive TOR
signaling by leucine analogues in adipocytes. J Cell Biochem 77:234-251.
109
Hyde R, Taylor PM & Hundal HS (2003). Amino acid transporters: roles in amino acid sensing and
signalling in animal cells. Biochem J 373:1-18.
19 Dott. Antonio Sartini - 22/04/2011
Haussinger D (1996). The role of cellular hydration in the regulation of cell function. Biochem J 313:697-
110
710.
111
Kim E (2009) Op, cit. n. 107 112
Fahien LA, MacDonald MJ, Kmiotek EH, Mertz RJ & Fahien CM (1988). Regulation of insulin release by
factors that also modify glutamate dehydrogenase. J Biol Chem 263:13610-13614.
113
Inoki K, Zhu T & Guan KL (2003b) Op.cit.n. 106 114
Xu G, Kwon G, Cruz WS, Marshall CA & McDaniel ML (2001). Metabolic regulation by leucine of
translation initiation through the mTOR-signaling pathway by pancreatic beta-cells. Diabetes 50:353-360.
115
Dennis PB, Jaeschke A, Saitoh M, Fowler B, Kozma SC & Thomas G (2001). Mammalian TOR: a
homeostatic ATP sensor. Science 294:1102-1105
116
XU et all(2001) Op.cit.n. 114
117
Lynch CJ, Fox HL, Vary TC, Jefferson LS & Kimball SR (2000). Regulation of amino acid-sensitive TOR
signaling by leucine analogues in adipocytes. J Cell Biochem 77:234-251.
118
Long X, Ortiz-Vega S, Lin Y & Avruch J (2005b). Rheb binding to mammalian target of rapamycin (mTOR)
is regulated by amino acid sufficiency. J Biol Chem 280:23433-23436
119
Roccio M, Bos JL & Zwartkruis FJ (2006). Regulation of the small GTPase Rheb by amino acids.
Oncogene 25:657-664.
120
Smith EM, Finn SG, Tee AR, Browne GJ & Proud CG (2005). Thetuberous sclerosis protein TSC2 is not
required for the regulatio of the mammalian target of rapamycin by amino acids and certaincellular stresses.
J Biol Chem 280:18717-18727.
121
Kim et all. (2002) Op.cit. n. 85 122
Hsieh LC, Chien SL, Huang MS, Tseng HF, Chang CK. Anti-inflammatory and anticatabolic effects of
short-term -hydroxy--methylbutyrate supplementation on chronic obstructive pulmonary disease
patients in intensive care unit. Asia Pac J Clin Nutr. 2006; 15:544-550. 123
Eley HL, Russell ST, Tisdale MJ. Attenuation of depression of muscle protein synthesis induced by
lipopolysaccharide, tumor necrosis factor, and angiotensin II by -hydroxy--methylbutyrate. Am J
Physiol Endocrinol Metab. 2008;295: E1409-E1416. 124
Yeh SS, Blackwood K, Schuster MW. The cytokine basis of cachexia and its treatment: are they ready
for prime time? J Am Med Dir Assoc. 2008; 9:219-236. 125
Eley HL, Russell ST, Tisdale MJ. Mechanism of attenuation of muscle protein degradation induced by
tumor necrosis factor-and angiotensin II by -hydroxy--methylbutyrate. Am J Physiol Endocrinol
Metab. 2008;295: E1417-E1426. 126
Eley HL, Russell ST, Baxter JH, Mukerji P, Tisdale MJ. Signaling pathways initiated by -hydroxy-methylbutyrate to attenuate the depression of protein synthesis in skeletal muscle in response to
cachectic stimuli. Am J Physiol Endocrinol Metab. 2007;293: E923-E931. 127
Manzano M, Giron MD, Salto R, Sevillano N, Rueda R, Lopez-Pedrosa JM. Is -hydroxy-methylbutyrate (HMB) the bioactive metabolite of L-leucine (LEU) in muscle? Molecular evidence and
potential implications. Abstract presented at: European Society for Clinical Nutrition and Metabolism 31st
Congress; Vienna, Austria; August 29-September 1, 2009. Abstract P267. 20