“ Esploriamo i cromosomi”
Un percorso didattico per le Scuole
Secondarie di Secondo Grado
1
INDICE
1. La doppia elica
p. 1
2. Replicazione del DNA
p. 2
3. Com’è impacchettato il DNA?
p. 4
4. Caratteristiche strutturali dei cromosomi degli eucarioti
p. 5
5. Analisi citogenetica
5.1. Cromosomi umani
5.2. Le mappe cromosomiche e i bandeggi
5.3. Applicazioni dell’analisi citogenetica
p. 6
p. 6
p. 7
p. 10
6. Osservazione e ricostruzione del cariotipo umano in laboratorio
6.1. Allestimento di preparati cromosomici
6.2. Ricostruzione del cariotipo umano
p. 10
p. 10
p. 11
7. Ibridazione molecolare e mappatura geni
7.1. Ibridazione molecolare
7.2. Ibridazione in situ fluorescente
7.3. Classificazione sonde
p. 12
p. 12
p. 13
p. 14
8. Modello didattico della ibridazione in situ
p. 17
9. Domande di autovalutazione
p. 18
10. Glossario
p. 22
2
1. LA DOPPIA ELICA
Essendo stato dimostrato negli anni ’40 che il DNA
(sigla dell'acido desossiribonucleico) era il materiale
ereditario, la domanda cruciale che ci si poneva agli
inizi degli anni ’50 era come il DNA svolgesse la sua
funzione. Per trovare la risposta a questo quesito era
d’altra parte indispensabile determinarne la struttura
chimica.
Nel 1953, James Watson e Francis Crick riuscirono a
decifrarla, determinando l’inizio di una nuova era
della biologia, quella della biologia molecolare.
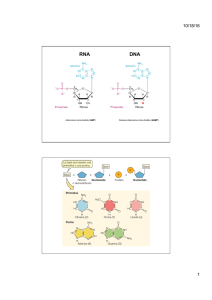
Oggi sappiamo che il DNA è un polimero, ossia un
insieme di tanti monomeri:i nucleotidi (Fig.1). Ogni
Fig 1.
Un nucleotide è formato da un
nucleotide è co-stituito da
gruppo fosfato, da uno zucchero a cinque
tre componenti:
atomi di carbonio e da una base azotata. In
questo
caso
è
mostrato
un
uno
zucchero
(il
desossiribonucleotide monofosfato, il
desossiribosio) a cui sono
monomero del DNA.
legati un gruppo fosfato e
una base azotata (adenina,
A; guanina, G; timina, T; citosina, C). Adenina e guanina sono basi
puriniche, timina e citosina sono basi pirimidiniche. Gli atomi di
carbonio del desossiribosio vengono numerati da 1’ a 5’ (per distinguerli
da quelli della base azotata, numerati 1, 2, 3, ecc.). Il carbonio in
posizione 5’ si lega al gruppo fosfato, mentre l’ossidrile (OH) nella
posizione 3’ è libero per formare il legame con il nucleotide successivo.
La molecola di DNA è
formata da due catene
polinucleotidiche
avvolte
l’una intorno all’altra con
andamento destrorso. (Fig.2).
Le
due
catene
sono
antiparallele, ossia i due
singoli
filamenti
sono
orientati
in direzioni
opposte.
Gli
scheletri
zucchero–fosfato si trovano
all’esterno, mentre le basi
azotate sono rivolte verso
l’interno. Le basi delle due
catene sono unite tra loro
mediante legami idrogeno e
Fig 3 .
Schema di una molecola
si appaiano secondo una
di DNA. I due filamenti hanno
orientamento 5’→3’ opposto. La
regola di complementarietà
sequenza delle basi azotate lungo un
precisa: adenina con timina
filamento determina la sequenza
delle basi lungo il filamento
A=T (con formazione di due Fig 2. Struttura a doppio filamento di un breve
complementare. Tra un’Adenina di
di DNA. I montanti della scala sono costituiti da
legami a idrogeno) citosina segmento
un filamento e una Timina del
molecole di zucchero alternate a gruppi fosfato. La distanza
filamento opposto sono presenti due
con guanina G≡C (con tra i due montanti misura 2 nm. La distanza tra due basi
legami a idrogeno; analogamente tra
successive lungo il filamento è di 0,34 nm. Le basi si
formazione di tre legami a azotate
una Guanina e una Citosina sono
appaiano sull’asse centrale dell’elica. Il passo dell’elica è di
presenti tre legami a idrogeno.
idrogeno)
(Fig.3).
Ne 3,4 nm.
consegue che il legame AT è
3
più debole di quello GC. L’appaiamento di una base purinica (più grande) con una pirimidinica (più
piccola) consente di mantenere costante il diametro dell’elica del DNA. In base alle regole di
complementarietà delle basi, conoscendo la sequenza di uno dei due filamenti di una molecola di
DNA, è quindi possibile prevedere esattamente quella del filamento complementare. Inoltre, in una
molecola di DNA a doppio filamento, la frequenza con cui compare l’Adenina sarà sempre uguale a
quella con cui compare la Timina e analogamente per Citosina e Guanina.
2. REPLICAZIONE DEL DNA
Caratteristica peculiare del DNA è la sua capacità di autoreplicarsi, che rappresenta il fondamento
della trasmissione dell’informazione genetica. La modalità di duplicazione del DNA era già stata
intuita da Watson e Crick nel 1953, come conseguenza della struttura della molecola stessa.
Il processo di duplicazione del DNA avviene nella fase S (sintesi) del ciclo cellulare (Fig.4) e deve
essere completo perché la cellula possa andare incontro alla divisione.
Ad ogni ciclo di divisione cellulare, ciascun cromosoma deve
essere fedelmente duplicato in modo che ciascuna delle due
cellule figlie ne possa ricevere una copia identica.
La replicazione del DNA in tutte le cellule viventi, dai batteri
all’uomo, è un processo complesso, che richiede l’intervento di
più di una dozzina di enzimi diversi.
Fig. 4. Il ciclo cellulare: le quattro fasi
principali in cui è suddiviso, G1, S, G2
e mitosi.
La replicazione inizia in corrispondenza di siti detti origini di
replicazione presenti ad intervalli lungo i cromosomi. In questi
siti, alcune proteine enzimatiche (ad esempio la DNA elicasi)
srotolano la doppia elica di DNA, rompendo i legami a
idrogeno tra le basi dei due filamenti complementari e generando così i filamenti „stampo“.
La polimerizzazione di una nuova catena di DNA si realizza grazie all’appaiamento successivo di
nucleotidi complementari alla catena „stampo“, uniti tra loro tramite legami fosfodiesterici ad opera
dell’enzima DNA polimerasi. La DNA polimerasi, per iniziare il processo, ha anche bisogno di un
innesco, detto primer, a cui attaccare il primo nucleotide per procedere con la polimerizzazione. Il
primer è costituito da una corta sequenza di RNA (sintetizzata ad opera di una RNA polimerasi,
chiamata primasi). Poichè la polimerizzazione avviene solo in direzione 5‘→ 3‘, la sintesi dei nuovi
filamenti può avvenire senza interruzione solo nella direzione 5‘→ 3‘ (filamento guida), mentre
nella direzione opposta 3‘→ 5‘(filamento in ritardo) avviene attraverso la sintesi di corti frammenti
di 100-2000 nucleotidi, detti frammenti di Okazaki, assemblati da parte della DNA ligasi.
L’avanzamento bidirezionale della replicazione genera due forcelle di replicazione, visualizzabili
come bolle di replicazione (Fig.5).
I cromosomi eucariotici si duplicano rapidamente poichè la sintesi
inizia a partire da molte origini di replicazione presenti nel genoma (è
stato dimostrato che se in un cromosoma eucariotico fosse presente una
sola origine di replicazione, il genoma impiegherebbe 35 giorni per
completare la sua replicazione!).
Negli eucarioti è detto replicone o unità di replicazione la porzione
Fig.5.
Fotografia al microscopio
di DNA che va dall‘origine della replicazione ai due punti di
elettronico della formazione di una
bolla di replicazione. Le frecce
terminazione della stessa, ovvero dove due forcelle di replicazione
indicano le forcelle replicative.
adiacenti si incontrano.
4
La replicazione è semiconservativa: ogni emielica (singolo filamento) della molecola madre serve
da stampo per la sintesi di un nuovo filamento, per cui ogni doppia elica figlia sarà costituita da un
filamento vecchio e da un filamento nuovo (Fig.6). Le molecole risultanti sono copie esatte
dell’originale, per cui da una doppia elica madre derivano due doppie eliche figlie uguali tra loro e
uguali alla molecola madre.
Fig 6.
Rappresentazione grafica
del DNA in replicazione, con la
formazione dei due nuovi filamenti
complementari ai filamenti “stampo”
della molecola originaria.
5
3. COM’È IMPACCHETTATO IL DNA?
L’intero DNA genomico di una cellula eucariotica è contenuto nel nucleo. Dal momento che la
lunghezza totale del genoma umano, se potessimo distendere e allineare tutti i cromosomi, sarebbe
di circa 2 metri e il diametro medio del nucleo è di soli 5-10 µm, il DNA deve accorciarsi e
compattarsi di almeno 10 000 volte, attraverso diversi livelli di avvolgimento (Fig.7).
Il livello più semplice di impacchettamento è la fibra a “nucleosomi”, formata da una successione
regolare di subunità costituite dalla doppia elica del DNA avvolta attorno a un nucleo di 8 proteine
basiche, gli istoni, che si assemblano a formare un ottamero (il DNA è acido per la presenza dei
gruppi fosfato). Questa struttura è chiamata anche “collana di perle” dove le perle sono i
nucleosomi e il filo della collana sono i tratti di DNA fra un nucleosoma e l’altro (chiamati anche
DNA linker).
Questa struttura viene resa ancora più compatta da un ulteriore avvolgimento della collana di perle
su se stessa a formare una struttura a solenoide, chiamata fibra di 30 nm. Questa fibra, per
aumentare ulteriormente l’impacchettamento, è ancorata su una struttura proteica, chiamata matrice,
con formazione di vere e proprie anse. Il maggior
a)
grado di condensazione del DNA si ottiene durante la
mitosi, quando infatti i singoli cromosomi risultano
visibili anche al microscopio ottico come formazioni
allungate.
b)
c)
d)
Fig. 7.
Stadi del ripiegamento della cromatina
che daranno origine ad un cromosoma eucariotico in
metafase completamente condensato.
a) Doppia elica di DNA
b)
Cromatina a” perle”
c)
Fibra di 30 nm
d)
Domini ad anse
e)
Spirali condensate
f)
Cromosoma metafasico
e)
f)
6
4. CARATTERISTICHE STRUTTURALI DEI CROMOSOMI DEGLI EUCARIOTI
La struttura del cromosoma eucariotico
Il cromosoma degli eucarioti è costituito da cromatina, un complesso di DNA, proteine
cromosomiche e RNA, che può presentarsi sotto forma di:
• Eucromatina che si colora debolmente perché despiralizzata ed è geneticamente attiva
• Eterocromatina che si colora intensamente perché condensata ed è geneticamente inattiva;
replica nella fase S dopo l’eucromatina e può, a sua volta, essere suddivisa in costitutiva,
sempre spiralizzata, e facoltativa, potenzialmente despiralizzabile.
Le proteine cromosomiche possono essere suddivise in:
• Proteine istoniche: hanno una carica positiva che facilita il loro legame col DNA. Ci sono 5
tipi di istoni: H1 che stabilizza l’avvolgimento a solenoide, e H2A, H2B, H3, H4 costituenti
degli ottameri. Le sequenze aminoacidiche indicano un elevato grado di conservazione tra
specie filogeneticamente distanti.
• Proteine non istoniche: alcune hanno un ruolo strutturale, alcune sono enzimi e altre sono
proteine implicate nella replicazione e trascrizione.
Nei procarioti tutto il genoma è presente come DNA a sequenze uniche. Il genoma umano può
essere descritto come costituito da :
-64% di DNA a sequenze uniche
-25% di DNA a sequenze moderatamente ripetute
-10% di DNA a sequenze altamente ripetute
Le sequenze altamente ripetute tendono ad essere localizzate nelle regioni eterocromatiche, ad
esempio attorno ai centromeri e alle estremità dei cromosomi.
Le sequenze uniche e moderatamente ripetute tendono ad essere sparse.
La morfologia dei cromosomi
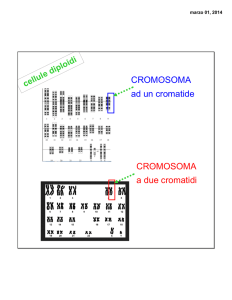
I cromatidi
Nel cromosoma mitotico corrispondono ai due filamenti di DNA duplicati, in attesa di essere
separati durante la divisione cellulare.
Fig. 8. Morfologia di un cromosoma metafasico. 1. centromero o costrizione primaria; 2. telomeri. Nello schema a destra è evidenziato in nero (3)
uno dei due cromatidi
I centromeri
Nei cromosomi mitotici il centromero corrisponde al punto di collegamento dei due cromatidi. Alla
presenza del centromero è associato il comportamento dei cromosomi alla mitosi ed alla meiosi. Se
i centromeri non funzionano correttamente, può avvenire una non-disgiunzione dei cromosomi
omologhi alla meiosi e dei cromatidi alla mitosi. La regione del centromero si associa al cinetocoro
al quale si attaccano le fibre del fuso durante la divisione.
7
Fig. 9. Diversi tipi morfologici di cromosoma
A Metacentrico B Submetacentrico
C Acrocentrico.
p= braccio corto;
q= braccio lungo c.s.= costrizione secondaria,
s.= satellite, c.=centromero
In base alla posizione del centromero i cromosomi vengono classificati in (Fig. 9):
- metacentrici, quando il centromero divide il cromosoma in due bracci circa di
dimensioni uguale;
- submetacentrici, quando il centromero divide il cromosoma in due bracci di
dimensioni diverse;
- acrocentrici, quando il centromero è subterminale.
I telomeri
Alle estremità dei cromosomi si trovano delle strutture speciali, i telomeri, costituiti da una
sequenza di nucleotidi ripetuta migliaia di volte; in tutti i mammiferi questa sequenza è TTAGGG. I
telomeri hanno una funzione di protezione dell’estremità e svolgono un ruolo fondamentale
nell’assicurare una corretta replicazione delle doppie eliche lineari dei cromosomi eucariotici
5. ANALISI CITOGENETICA
5.1 Cromosomi umani
Una differenza fondamentale tra procarioti ed eucarioti risiede nel fatto che i procarioti hanno un
singolo cromosoma, mentre la maggior parte degli eucarioti ha un numero diploide di
cromosomi in quasi tutte le cellule somatiche.
Un insieme completo di tutti i cromosomi metafasici di una cellula è definito cariotipo; il cariotipo
è specie-specifico. In particolare, il cariotipo umano normale diploide è costituito da 46 cromosomi
(22 paia di autosomi e un paio di cromosomi del sesso): le femmine sono 46, XX e i maschi 46,
XY (Fig. 10).
Pertanto, per effettuare un’analisi citogenetica è quindi necessario allestire un preparato
cromosomico per identificare e classificare i diversi cromosomi.
Un preparato si può allestire a partire da qualsiasi cellula che si divida, sia spontaneamente, sia in
seguito a stimolazione in coltura con sostanze che inducono la divisione.
Le analisi cromosomiche possono essere effettuate a partire da cellule sia di un feto sia di un
individuo dopo la nascita. Generalmente prima della nascita, il cariotipo si effettua su amniociti
ottenuti mediante amniocentesi o su villi coriali ottenuti mediante villocentesi. Dopo la nascita, i
preparati cromosomici per le indagini citogenetiche si allestiscono da linfociti di sangue periferico.
8
I cromosomi umani
Centromero
b)
Telomero
Cromatidi
Fig.10.
a) Struttura di un cromosoma metafasico.
b) Esempio di cariotipo umano maschile
5.2 Le mappe cromosomiche e i bandeggi
L’analisi del cariotipo o mappa cromosomica viene effettuata attraverso la ricostruzione del
cariogramma, ottenuto disponendo i cromosomi metafasici appaiati secondo un sistema di
classificazione internazionale. I cromosomi sono suddivisi in gruppi omogenei e disposti in ordine
decrescente secondo la lunghezza e l’indice centromerico (rapporto fra la lunghezza del braccio
corto e la lunghezza totale del cromosoma) (Fig. 10 b).
Un’identificazione inequivocabile di ogni cromosoma del cariotipo deriva da tecniche di
colorazione dei cromosomi denominate bandeggi.
Intorno al 1970 sono state introdotte infattimetodiche di colorazione dei cromosomi che utilizzano
sostanze in grado di colorare specifiche regioni cromosomiche (bande) in modo più evidente di
altre. Il bandeggio che ne deriva è specifico e riproducibile per ogni coppia di cromosomi omologhi
e permette di ricostruire il cariotipo in modo inequivocabile, evidenziando eventuali anomalie
cromosomiche sia di numero sia di struttura.
Esistono diversi tipi di bandeggi di cui i principali sono:
• Bande Q, ottenute mediante l’impiego di un colorante fluorescente, la Quinacrina: le bande
più luminose intensamente fluorescenti corrispondono alle zone ricche in Adenina e Timina
(Fig. 11 a)
9
• Bande G, ottenute col colorante Giemsa: le bande più scure corrispondono alle zone ricche
in Adenina e Timina, relativamente povere di geni, e risultano quindi corrispondenti e
sovrapponibili alle bande Q (Fig. 11 b e Fig. 11 c).
• Bande R, ottenute mediante denaturazione al calore e opportuna colorazione: sono l’inverso
delle bande Q e G e corrispondono a zone ricche in Citosina e Guanina.
Fig. 11. Differenti esempi di tecniche di bandeggio di cromosomi umani
a) Le bande Q si ottengono con la tecnica di bandeggio con
quinacrina
b)
Le bande G si ottengono con la tecnica di bandeggio Giemsa
c)
Schema riassuntivo delle bande G
a) bande Q
b) bande G
10
c) Bande G
11
5.3 Applicazioni dell’analisi citogenetica
•
•
•
•
Diagnosi prenatale di sindromi associate ad anomalie cromosomiche
Diagnosi postnatale di anomalie cromosomiche in individui portatori di sindromi o di
malattie genetiche
Diagnosi postnatale di anomalie cromosomiche bilanciate in individui sani che mostrano
poliabortività
Citogenetica delle cellule tumorali: identificazione dei cromosomi “marcatori” e mappaggio
di regioni subcromosomiche che contengono geni associati allo sviluppo o alla progressione
tumorale.
6.OSSERVAZIONE E RICOSTRUZIONE DEL CARIOTIPO UMANO IN LABORATORIO
6.1 Allestimento di preparati cromosomici a partire da una linea di cellule linfoblastoidi
(cellule a moltiplicazione continua ottenuta a partire da linfociti)
Preparazione della sospensione cromosomica
NB:questa procedura non verrà effettuata direttamente dagli studenti
•
Allestimento della coltura da una linea di cellule linfoblastoidi
Le cellule vengono messe in coltura in una fiasca a 37°C e quando raggiungono una fase di
crescita esponenziale viene aggiunta per 1 ora la colchicina, una sostanza che inibisce la
formazione del fuso mitotico e quindi determina il blocco delle mitosi in metafase.
•
Recupero metafasi
Le cellule vengono raccolte mediante centrifugazione (e scarto del surnatante) e trattate con
soluzione ipotonica per determinare il rigonfiamento delle cellule e la rottura della membrana
cellulare.
•
Fissazione metafasi
Segue un trattamento con fissativo che stabilizza la struttura del cromosoma, altrimenti fragile.
Il fissativo permette infatti di conservare il materiale organico, ritardando l’azione degli agenti
ossidanti.
Il sedimento cromosomico è quindi mantenuto in una soluzione di fissativo (costituita da
metanolo: acido acetico=3:1). Il citoplasma, in cui sono immersi i cromosomi, viene disidratato
dall’alcool e ridotto dall’acido acetico; i cromosomi mantengono la medesima posizione che
presentavano prima del fissaggio nel citoplasma. Il passaggio in fissativo viene ripetuto almeno
un’altra volta, al termine di questi passaggi la sospensione cromosomica può essere conservata
in provetta a – 20°C anche per qualche anno, oppure può venire strisciata su vetrino.
Attività effettuate direttamente dagli studenti
•
Striscio su vetrino
La sospensione cromosomica viene strisciata su un vetrino sgrassato e lavato, effettuando i
seguenti passaggi:
-
inserire un vetrino in una vaschetta di acqua distillata
12
-
•
riempire il capillare della pipetta Pasteur con la sospensione cromosomica
togliere il vetrino dalla vaschetta e lasciare cadere gocce di sospensione su metà della
superficie del vetrino
asciugare con carta assorbente il retro del vetrino
lasciare asciugare all’aria per 5’-10’ e quindi procedere alla colorazione
Colorazione con colorante Giemsa
E’ una metodica che determina la colorazione omogenea di tutti i cromosomi, evidenziando la
loro dimensione e morfologia rispetto alla posizione del centromero.
La colorazione si effettua in due passaggi:
- immergere i vetrini per 25’ in una soluzione di Giemsa al 10% in tampone
Soerensen
[Tampone Soerensen (pH = 6.88) 100 ml = 50ml sol.A+ 50ml sol.B
Sol.A 1/15M KH2PO4 (9.08g/l)
Sol.B 1/15M Na2HPO4 H2O (11.88 g/l)]
-
sciacquare i vetrini con acqua distillata e lasciare asciugare all’aria.
osservare i vetrini direttamente al microscopio ottico. I vetrini possono essere montati con
vetrino copriogetto in modo permanente utilizzando apposite resine.
NB: Tutti i procedimenti per la colorazione vanno effettuati coi guanti.
6.2. Ricostruzione del cariotipo umano
La ricostruzione del cariotipo a partire dai preparati allestiti dagli studenti in laboratorio risulta
decisamente laboriosa e complessa. In alternativa, è possibile effettuare delle attività di simulazione
al computer.
Viene proposta la “Karyotyping activity” allestita dalla University of Arizona.
Il riferimento è:
www.biology.arizona.edu/human_bio/activities/karyotyping/karyotyping.html
Si tratta di una simulazione della ricostruzione di cariotipi umani a partire da cromosomi con
bandeggio G, fatta utilizzando immagini digitali. Vengono presentati i cariotipi parzialmente
ricostruiti di tre pazienti portatori di anomalie cromosomiche. Lo studente è invitato dapprima a
completare il cariotipo appaiando ai rispettivi omologhi una serie di cromosomi non appaiati
presentati in successione. E' chiamato poi ad osservare e interpretare i risultati così ottenuti,
formulando alla fine una diagnosi, come se stesse effettivamente lavorando in un centro di analisi.
Questo tipo di attività consente essenzialmente di identificare in individui affetti da particolari
patologie di anomalie del numero dei cromosomi.
13
7. IBRIDAZIONE MOLECOLARE E MAPPATURA DEI GENI
La possibilità di visualizzare la posizione dei geni o di altre regioni non codificanti del genoma su
un cromosoma (mappatura) consente anche l’individuazione di anomalie submicroscopiche di
struttura dei cromosomi, in quanto è possibile visualizzare lo spostamento (traslocazioni), la
perdita (delezioni), o l’aggiunta (duplicazioni) di tratti cromosomici anche molto piccoli, non
rilevabili con la citogenetica convenzionale.
7.1. Ibridazione molecolare
La procedura su cui si basa la mappatura di un gene è l’ibridazione molecolare.
Quando una soluzione acquosa di DNA è riscaldata a 100 °C o esposta ad un pH molto alto, i
legami idrogeno tra le coppie di basi complementari che normalmente tengono insieme i due
filamenti della doppia elica vengono rotti e la doppia elica si dissocia in due filamenti singoli.
Questo processo viene chiamato denaturazione del DNA.
I singoli filamenti di DNA in particolari condizioni (abbassamento della temperatura a 65 °C,
variazione di pH ) possono riassociarsi a riformare doppie eliche secondo un processo chiamato
rinaturazione del DNA. Reazioni simili possono avvenire fra due catene di DNA a singolo
filamento di diversa origine, purché abbiano sequenze nucleotidiche complementari, e il risultato è
definito ibrido molecolare.
Una sonda molecolare o probe è un frammento noto di DNA a singolo filamento usato in un
esperimento di ibridazione molecolare per identificare molecole specifiche di DNA nelle quali è
presente una sequenza complementare alla sonda stessa.
Una sonda molecolare viene marcata con isotopi radioattivi o sostanze fluorescenti per consentirne
la rivelazione. Le reazioni di ibridazione con sonde di DNA possono essere effettuate in provetta (in
vitro), o con campioni di DNA posizionati su filtri di nylon o di nitrocellulosa (Southern Blotting)
per rivelare l’eventuale presenza in un particolare campione di DNA di sequenze complementari
alla sonda.
Questa tecnica, come la maggior parte delle tecniche di biologia molecolare prevede l’utilizzo di
acidi nucleici purificati, il che implica la distruzione delle strutture cellulari e cromosomiche del
campione e l’impossibilità di localizzare direttamente sui cromosomi geni o qualsiasi altra sequenza
di DNA rappresentata dalla sonda molecolare.
Per superare questo problema sono state sviluppate tecniche di ibridazione molecolare in cui sonde
marcate di acidi nucleici sono usate per localizzare sequenze specifiche di acidi nucleici in una
determinata struttura biologica, cioè in situ, attraverso un procedimento chiamato ibridazione in
situ. L’ibridazione viene fatta avvenire non in provetta, ma mettendo a contatto la sonda con un
14
substrato solido su cui, nel caso in cui si voglia localizzare un gene su un cromosoma, è stato
allestito un preparato cromosomico. L'ibridazione in situ permette quindi di visualizzare l'acido
nucleico d'interesse nella sua sede naturale, preservando perfettamente la struttura tessutale,
cellulare o cromosomica del preparato.
7.2 Ibridazione in situ fluorescente
L'ibridazione in situ fluorescente (FISH : Fluorescence In Situ Hybridization) è una tecnica di
ibridazione in situ che permette di visualizzare a livello cromosomico la localizzazione di un gene o
di una qualsiasi sequenza genomica d'interesse, usando sonde di DNA marcate con composti
fluorescenti. Il vantaggio, rispetto alla marcatura con isotopi radioattivi, è rappresentato
dall’eliminazione del radioattivo, dall’ottenimento di un “segnale” più nitido e dalla possibilità di
usare fluorocromi a diversa emissione cromatica, consentendo di usare visualizzare
contemporaneamente e più sonde.
Attraverso questa tecnica è possibile pertanto localizzare su cromosomi metafasici sequenze
genomiche di limitata estensione e di identificare una loro eventuale localizzazione alterata
(traslocazione e inversione) o una loro assenza (delezione), o una loro aggiunta (duplicazione).
Tale metodica, applicata ai cromosomi metafasici, ha incrementato il livello di risoluzione della
citogenetica convenzionale che permette di identificare le anomalie cromosomiche superiori a 4 Mb
(1 Mb = 106 paia di basi). La FISH invece consente di visualizzare anomalie che coinvolgono poche
migliaia di paia di basi.
Inoltre, la possibilità di utilizzare per questo tipo di studi anche nuclei interfasici permette di
analizzare anche tipi cellulari che difficilmente vanno incontro a divisione cellulare, oppure inclusi
in paraffina e come tali non più coltivabili in vitro.
La procedura è abbastanza semplice e prevede:
1. La scelta di una sonda, ossia di una sequenza conosciuta di DNA
2. La marcatura della sonda con composti fluorescenti che vengono legati ad essa
3. L’allestimento dei preparati da studiare
4. La denaturazione del DNA, sia dei preparati sia della sonda, per consentire successivamente la
formazione di legami idrogeno tra il filamento del DNA cromosomico e i filamento del DNA
della sonda
5. L’ibridazione: appaiamento del DNA dei preparati con il DNA sonda, introdotto in eccesso
6. Il lavaggio dalla sonda in eccesso non appaiata
7. L’osservazione dei preparati con microscopio a fluorescenza
15
7.3. Classificazione delle sonde
Fig. 12
Sonde cromosoma-specifiche
La sonda cromosoma-specifica è costituita da un insieme di sonde di DNA ognuna delle quali è omologa all’intero cromosoma considerato.
L’insieme delle sonde copre l’intero cromosoma. L’ibridazione con una sonda cromosoma-specifica marcata con un fluorocromo permetterà di
visualizzare segnali fluorescenti su una determinata coppia di cromosomi omologhi lungo tutto il cromosoma.
Sonde centromero-specifiche
La sonda centromero-specifica è costituita da un tipo di sequenze ripetute, dette alfoidi, presenti nei centromeri. Sono state isolate sequenze alfoidi
specifiche per il centromero di ogni cromosoma. L’ibridazione con una sonda centromero-specifica marcata con un fluorocromo permetterà di
visualizzare segnali fluorescenti a livello dei centromeri di una coppia di cromosomi omologhi.
Sonde telomero-specifiche
La sonda telomero-specifica è costituita da sequenze localizzate nella parte terminale dei cromosomi. Sono state isolate vicino alla parte terminale
della molecola di DNA, che ha una sequenza comune a tutti i cromosomi, sequenze specifiche per il telomero di ogni cromosoma. E’ più corretto
definire le sequenze telomeriche specifiche per uno dei due bracci di un determinato cromosoma (braccio corto “p”, braccio lungo “q”) “sequenze sub
telomeriche”. L’ibridazione con una sonda telomerica specifica per uno dei bracci di un determinato cromosoma, marcata con un fluorocromo,
permetterà di visualizzare segnali fluorescenti a livello dei telomeri di uno dei due bracci di una coppia di cromosomi omologhi.
Se la sonda telomero-specifica è costituita dalle sequenze che costituiscono la parte terminale della molecola di DNA, comuni a tutti i cromosomi, la
sonda viene detta specifica per i “telomeri comuni”. In questo caso in seguito all’ibridazione si visualizzeranno segnali fluorescenti sui telomeri di
tutti i cromosomi.
Sonde locus-specifiche
La sonda locus-specifica è costituita dalla sequenza di una regione delimitata lungo uno specifico cromosoma. L’ibridazione con una sonda locusspecifica marcata con un fluorocromo permetterà di visualizzare segnali fluorescenti a livello di una regione sul braccio corto o sul braccio lungo di
una coppia di cromosomi omologhi.
16
Fig. 13. Ibridazione in situ con diverse tipologie di sonde
a) Ibridazione in situ con una sonda specifica per il cromosoma 8: i segnali fluorescenti sono presenti lungo tutta l’estensione dei due cromosomi
omologhi. b) Ibridazione in situ con una sonda specifica per le sequenze centromeriche del cromosoma X: i segnali fluorescenti identificano due
cromosomi X. c) Ibridazione in situ con una sonda specifica per i telomeri del braccio lungo del cromosoma 4: i segnali fluorescenti sono presenti sui
telomeri del braccio lungo dei due cromosomi 4 omologhi. d) Ibridazione in situ con una sonda specifica per una regione del braccio lungo del
cromosoma 17: i segnali fluorescenti sono presenti in una regione intracromosomica del braccio lungo di entrambi gli omologhi 17.
17
Fig. 14. Livello di risoluzione della FISH
FISH su cromosomi metafasici
Il livello di risoluzione della FISH viene definito in base alla distanza minima alla
quale si vedono due segnali distinti prodotti dall’ibridazione di due sonde, questo
dipende dal grado di allungamento e/o di despiralizzazione della cromatina. Il
cromosoma metafasico rappresenta lo stadio in cui la cromatina raggiunge il
massimo grado di compattamento.
Il livello di risoluzione della FISH su cromosomi metafasici (vedi allestimento
preparati cromosomici convenzionalli.) va da 2 a 3 megabasi (Mb).
FISH su cromosomi metafasici allungati meccanicamente
I cromosomi metafasici possono essere allungati partendo da preparazioni di
cromosomi metafasici convenzionali sottoposti a stiramento meccanico.
Il livello di risoluzione della FISH su cromosomi metafasici stirati è al massimo di
200 kilobasi (Kb).
Fig. 15. FISH con due sonde marcate con due fluorocromi
diversi su cromosomi a diverso grado di allungamento
a) FISH a doppio colore su cromosomi metafasici con due
sonde localizzate a 2,5 Mb di distanza: si può notare la quasi
totale sovrapposizione dei segnali fluorescenti. b) FISH a
doppio colore su cromosomi metafasici stirati
meccanicamente con le stesse sonde utilizzate in a:
l’allungamento del cromosoma permette di visualizzare i due
segnali fluorescenti rosso e verde separati da un segmento
cromatinico. c) FISH a doppio colore su fibre di DNA con le
stesse sonde utilizzate in a e in b: l’ulteriore despiralizzazione
della cromatina è evidenziato dall’aumento della distanza tra i
segnali fluorescenti rosso e verde.
FISH su fibre di DNA
Le fibre di DNA possono essere ottenute incubando nuclei interfasici strisciati su un
vetrino portaoggetto in una soluzione di lisi che rompe la membrana nucleare. Il
vetrino viene quindi estratto verticalmente dalla soluzione di lisi. Questo movimento
permette alle fibre di DNA, fuori uscite dal nucleo, di srotolarsi e di disporsi
longitudinalmente lungo il vetrino.
Il livello di risoluzione della FISH su fibre di DNA può arrivare a 1 Kb.
18
8. MODELLO DIDATTICO DELLA IBRIDAZIONE IN SITU
Per rendere maggiormente comprensibile questa tecnica si propone l’utilizzo di un modello che
permetterà agli studenti di acquisirne i principi di base, apprezzandone le diverse applicazioni.
Simulazione ibridazione in situ (Brevetto n°: MI2005A000710, Milano 20/05/05)
Il modello è costituito da una tavola di legno, che simula il vetrino porta oggetto su cui vengono
strisciati i cromosomi. Sopra il piano di legno vengono collocate 3 coppie di cromosomi. Ogni
cromosoma è suddiviso in regioni contraddistinte da numeri sulla superficie superiore, mentre sulla
superficie inferiore, appoggiata al piano di legno, esiste una guida entro cui far scorrere una o più
calamite. Un ulteriore componente dello strumento è costituito da sfere di ferro che rappresentano le
molecole della sonda da utilizzare per l’ibridazione.
La specificità di appaiamento mediante legame idrogeno tra il DNA della sonda e quello del gene
localizzato sul cromosoma viene simulato dal legame tra il metallo (sonda) e la calamita (DNA
complementare cromosomico) dovuto alla forza
magnetica.
Localizzazione di un gene su un cromosoma
Uno studente posiziona 4 calamite sotto i cromatidi
fratelli di cromosomi omologhi
Un secondo studente mette le biglie magnetiche che
rappresentano diverse molecole della sonda A, a
localizzazione ignota, in contatto con il piano su cui sono
stati posizionati i cromosomi per stabilire la
localizzazione della sonda stessa e della sequenza di DNA che essa rappresenta.
Si simula l’incubazione con una quantità di sonda in eccesso, inclinando dolcemente più volte il
piano in modo tale che tutti i cromosomi possano venire coperti dalla sonda
Si elimina la sonda che è stata messa in eccesso per favorire l’ibridazione aprendo lo sportello ad un
lato del piano, inclinando il piano stesso dolcemente: le sfere non trattenute dalla forza magnetica
usciranno similmente le molecole presenti sui cromosomi e non legate mediante legame idrogeno
potranno essere selettivamente allontanate. Se non viene effettuato questo passaggio, che simula il
lavaggio nell’ibridazione in situ, la sonda depositata casualmente sui cromosomi non permette di
individuare il segnale specifico.
Si visualizza la localizzazione della sonda A sui cromatidi dei cromosomi omologhi (ibridazione
specifica) localizzandola poi più precisamente nell’ambito di regioni cromosomiche specifiche
Identificazione di anomalie cromosomiche sub-microscopiche
Traslocazione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma e
due sotto i cromatidi fratelli di un cromosoma non omologo. Un secondo studente mediante
l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda (sfere di metallo) a
localizzazione nota, da dichiarare, osserverà che la sequenza specifica è traslocata su un altro
cromosoma, diverso dall’omologo atteso
Delezione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma senza
posizionare la calamita nella stessa regione dei cromatidi del cromosoma omologo. Un secondo
studente mediante l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda a
localizzazione nota (sfere di metallo), da dichiarare, osserverà che la sequenza specifica è deleta su
uno dei due cromosomi omologhi
19
Duplicazione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma e 4
calamite sotto i cromatidi fratelli del cromosoma omologo. Un secondo studente mediante
l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda (sfere di metallo) a
localizzazione nota, da dichiarare, osserverà che la sequenza specifica è duplicata su uno dei due
cromosomi omologhi.
Studio della cinetica di ibridazione
Lo studente posiziona 4 calamite sotto i cromatidi fratelli di 2 cromosomi omologhi.
Lo studente mette a contatto con il piano numerose sfere di metallo e, inclinando più volte
dolcemente il piano, verifica quanto tempo occorre dall’inizio dell’oscillazione perchè le sfere si
leghino ai due cromatidi fratelli di entrambi i cromosomi omologhi
Lo studente toglie le sfere lasciandone solo quattro, cioè in rapporto molare 1:1 sufficiente per
saturare le possibilità di legame e, inclinando più volte dolcemente il piano, verifica quanto tempo
occorre dall’inizio dell’oscillazione perchè le sfere si leghino ai due cromatidi fratelli di entrambi i
cromosomi omologhi.
Potrà facilmente essere dedotto che la cinetica di ibridazione dipende dalla quantità di sonda posta a
contatto con i cromosomi poichè, in caso di eccesso di sonda, l’incontro tra molecole a sequenza
specifica, è statisticamente favorito.
9. DOMANDE DI AUTOVALUTAZIONE
1) Nella molecola di DNA:
a) lo zucchero è il ribosio
b) i nucleotidi si differenziano tra loro per il gruppo fosfato e il pentoso
c) vi sono legami ionici tra le basi azotate complementari
d) ciò che varia è la sequenza delle basi azotate
e) vi possono essere differenti tipi di gruppi fosfato
2) Al termine del processo di duplicazione del DNA:
a) ogni molecola di DNA è composta da due nuovi filamenti
b) ogni molecola di DNA è composta da un filamento originario e da uno nuovo
c) una molecola di DNA è nuova , l’altra è originaria
d) nei filamenti si alternano parti originarie a parti nuove
e) sono vere tutte le precedenti, in quanto il processo è casuale
3) Nel processo di duplicazione del DNA, quale enzima catalizza il legame tra i nucleotidi per
formare un nuovo filamento?
a) DNA polimerasi
b) DNA elicasi
c) RNA polimerasi
d) DNA ligasi
e) DNA girasi
4) La citogenetica viene applicata per:
la selezione delle piante
la difesa dell’ambiente
le diagnosi pre e postnatale
lo studio dei tessuti
la trasmissione ereditaria
20
5) Il cariotipo e’:
il numero dei cromosomi di una cellula
il corredo aploide
l’insieme delle forme dei cromosomi
l’allineamento dei cromosomi alla metafase
l’insieme dei cromosomi di una cellula in metafase
6) La sindrome di Down e’:
la presenza di un cromosoma Y in più
una monosomia del cromosoma X
una trisomia del cromosoma 21
una malattia a trasmissione sessuale
una malattia infettiva
7) Il corredo genetico dell’uomo è:
2n=16
2n=64
2n=46
2n=48
2n=23
8) Le fasi della mitosi sono:
a) metafase,anafase,telofase,profase
b) telofase,profase,anafase,metafase
c) profase,metafase,anafase,telofase
d) interfase,metafase,anafase, telofase
e) anafase.profase,telofase,metafaase
9) I cromosomi umani contengono:
a) DNA e proteine
b) DNA e aminoacidi
c) DNA e protammine
d) DNA e vitamine
e) DNA
10) I geni coincidono con:
a) un cromosoma
b) un cromosoma di una coppia
c) un locus su una coppia di omologhi
d) un locus su un cromosoma di una coppia di omologhi
e) un allele
11) Il numero di autosomi presenti nelle cellule somatiche della specie umana è:
a) 44
b) 22
c) 23
d) 46
e) 21
21
12) I cromosomi metafasici vengono osservati con:
microscopio elettronico a scansione
microscopio ottico
microscopio elettronico a trasmissione
lente d’ingrandimento
occhio nudo
13) Per allestire i preparati cromosomici al fine di osservare i cromosomi di una cellula, è
necessario che le cellule:
a) provengano da tessuti indifferenziati
b) siano capaci di moltiplicarsi
c) siano in fase di quiescenza
d) provengano solo da individui giovani
e) derivino solo da individui sani
14) L’analisi del cariotipo viene effettuata osservando i cromosomi in:
a) interfase
b) fase G1 del ciclo cellulare
c) metafase
d) telofase
e) fase S del ciclo cellulare
15) Cariogramma significa:
a) visione del cariotipo
b) taglio del cariotipo su carta
c) esame del cariotipo
d)ricostruzione del cariotipo
e) colorazione del cariotipo
16) Le due parti in cui è diviso longitudinalmente ogni cromosoma metafasico sono dette:
a) centromeri
b) centrioli
c) telomeri
d) bracci
e) cromatidi
17) Completa la tabella mettendo in corrispondenza i termini (lettere) con le rispettive
definizioni (numeri).
A) cromosomi metacentrici
1) si hanno quando il centromero divide il
cromosoma in due bracci di dimensioni diverse
A
B) cromosomi submetacentrici
2) si hanno quando il centromero è subterminale
C) cromosomi acrocentrici
3) si hanno quando il centromero divide il cromosoma
in due bracci di dimensioni circa uguali
B
C
22
18) L’analisi del cariotipo consente di evidenziare:
a) le anomalie di numero o di struttura dei cromosomi
b) la perdita di un gene
c) le mutazioni geniche
d) l’acquisto di un gene
e) il crossig-over
19) Cosa si intende per bandeggio G a livello dei cromosomi ?
a) una colorazione specifica con verde janus
b) una colorazione a macchie con eosina
c) una colorazione a bande Gram-positivo
d)un trattamento con tripsina e colorazione con Giemsa
e) un trattamento specifico che rompe i cromosomi in bande chiare e scure
20) La disidratazione del citoplasma per il recupero dei cromosomi viene effettuata in:
a) alcool denaturato
b) solvente idrofobo
c) resina
d) colorante ematossilinico
e) enzima Rna polimerasi
21) La rottura della membrane cellulari avviene con trattamento di:
a) soluzioni ipertonica
b) soluzione ipotonica
c) soluzione isotonica
d) alcool
e) soluzione di NaCl
22) La soluzione di fissativo ha la funzione di: ( piu’ di una risposta esatta)
a) bloccare i cromosomi in metafase
b) rendere stabile la struttura dei cromosomi
c) abbassare la temperatura della cellula
d) causare la rottura della membrana cellulare
e) ritardare l’azione degli agenti ossidanti
23) Completa il brano, scegliendo tra i seguenti termini:
gruppi fosforici, 100°C, 65°C, legami idrogeno, rinaturato, basi azotate, sonde,
ibrida, legami ionici, doppia elica, denaturato, marcata, appaiano, separano, spaiate,
complementari.
La tecnica dell’ibridazione molecolare si basa sulla proprietà che hanno le .......................
degli acidi nucleici di appaiarsi in ben precise coppie. Infatti, se si scaldano a ...................
delle molecole di DNA in soluzione, i ........................ che tengono uniti i due filamenti si
spezzano e i filamenti si separano. In questo modo, il DNA risulta ...................... . Se poi
la soluzione viene raffreddata, i ........................ si riformano ricostituendo la ................... .
Quando vengono mescolate delle molecole di DNA provenienti da fonti diverse e poi
vengono scaldate, i filamenti si ...................... e subiscono delle collisioni casuali.
Se, mentre la soluzione si raffredda, si incontrano due filamenti con sequenze ..................,
essi formeranno una doppia elica ................. .
23
24) La durata del tempo di ibridazione molecolare dipende da:
a) temperatura
b) pH
c) luce
d) quantita’ della sonda
e) quantita’ di Dna da ibridare
25) Scrivi il significato dell’acronimo FISH.
.....................................................................................................................................
26) Scegli il completamento che ritieni esatto:
Una sonda molecolare
a) è in grado di individuare un determinato gene dotato di una sequenza nucleotidica
complementare
b) può copiare certi geni in possesso di basi azotate identiche a quelle della sonda stessa
c) viene inserita nei cromosomi batterici per portare al loro interno determinate sequenze
geniche
d) si lega al trascritto di RNA e mette in evidenza gli esoni
e) induce le cellule batteriche a moltiplicarsi
27) Completa la tabella mettendo in corrispondenza i termini (lettere) con le rispettive
definizioni (numeri).
A) delezione
1) il frammento di un cromosoma si attacca al cromosoma
omologo
B) duplicazione
2) il frammento di un cromosoma si attacca a un cromosoma
non omologo
C) traslocazione
3) il frammento di un cromosoma si riattacca al cromosoma
originario, ma in posizione rovesciata
D) inversione
4) il frammento di un cromosoma viene perso
A
B
C
D
10. GLOSSARIO
Bandeggio
Basi azotate
Cariotipo
Procedura di trattamento e colorazione dei cromosomi che induce una
differenziazione trasversale (alternanza di segmenti intensamente colorati
e segmenti più chiari). Permette il riconoscimento senza ambiguità di
ogni paio cromosomico.
Molecole contenenti azoto, costituenti i nucleotidi del DNA insieme allo
zucchero deossiribosio e al gruppo fosfato. Sono quattro e si classificano
in basi puriniche (A e G) e pirimidiniche (T e C).
Assetto cromosomico di una specie.
24
Centrifugazione
Centromero
Ciclo cellulare
Citogenetica
Colchicina
Crescita esponenziale
Cromatidio
Cromatina
Cromosoma
Cromosomi omologhi
Delezione
Denaturazione DNA
DNA
DNA elicasi
DNA polimerasi
Eucarioti
Eucromatina
Eterocromatina
FISH
Fissativo
Fuso mitotico
Giemsa
Metodo di separazione di sostanze o di componenti cellulari a diversa
densità
Regione di congiunzione tra i due cromatidi che compongono uno stesso
cromosoma
Sequenza ciclica e regolare degli eventi di crescita e divisione di una
cellula eucariotica; è costituito dalle fasi G1, S, G2 , mitosi, citodieresi.
Branca della genetica che studia i meccanismi dell'eredità a livello
cromosomico
Sostanza che determina il blocco della mitosi in metafase, inibendo la
formazione del fuso mitotico
Modalità di crescita delle cellule per cui ad ogni generazione raddoppia il
numero delle cellule della generazione precedente
Subunità longitudinale di un cromosoma metafasico, costituito da due
cromatidi fratelli uniti a livello del centromero.
Materiale di cui sono costituiti i cromosomi degli eucarioti. Risulta
dall’associazione tra DNA e proteine basiche.
Struttura generalmente allungata, costituita da cromatina, visibile al
microscopio ottico durante la divisione cellulare e contenente i geni in
successione lineare.
Coppie di cromosomi di forma generalmente simile, che contengono
informazioni per gli stessi caratteri. Portano gli stessi geni, non
necessariamente gli stessi alleli. Si appaiano alla meiosi.
Mutazione del DNA che comporta la perdita di uno o più nucleotidi
Separazione dei due filamenti della doppia elica, per rottura dei legami a
idrogeno tra le basi azotate, con conseguente perdita della struttura
tridimensionale
Abbreviazione di acido deossiribonucleico, macromolecola che
costituisce il materiale genetico di tutti gli organismi (ad eccezione di
alcuni virus). E’ costituito da due catene polinucleotidiche avvolte a
doppia elica.
Proteina che facilita l'apertura della doppia elica del DNA durante la sua
replicazione.
enzima che sintetizza DNA unendo insieme nucleotidi e usando un
filamento di DNA come stampo
Organismi costituiti da una o più cellule, in cui il materiale genetico è
localizzato entro un nucleo circondato da una membrana.
Parte della cromatina che va incontro a condensazione e decondensazione
durante il ciclo cellulare, attiva dal punto di vista trascrizionale.
Cromatina condensata, inattiva dal punto di vista trascrizionale
Fluorescence In Situ Hybridization. Procedura di ibridazione molecolare,
in base alla quale sequenze note di DNA (sonde) marcate con fluorocromi
vengono ibridate su DNA cromosomico denaturato fissato su preparati
cromosomici (in situ). Un segnale luminoso fluorescente rivela la regione
dell’avvenuta ibridazione.
Reagente chimico utilizzato per conservare le cellule per l'osservazione al
microscopio
Struttura presente nelle cellule in divisione, coinvolta nei movimenti dei
cromosomi
Colorazione specifica per il DNA cromosomico
25
Ibridazione in situ
Tecnica basata sull’ibridazione molecolare, mediante la quale una sonda
di RNA o DNA a singolo filamento viene utilizzata per localizzare una
sequenza nucleotidica
Ibridazione molecolare Processo per cui due filamenti complementari di acido nucleico si
riassociano a formare una doppia elica
Istoni
proteine basiche ricche in arginina e lisina che partecipano alla
formazione dei nucleosomi e alla struttura della cromatina nei cromosomi
eucariotici
Linea cellulare
Popolazione di cellule di origine animale e vegetale, in grado di
riprodursi indefinitamente in coltura
Linfoblastoide
Cellula immatura che nel processo di maturazione darà origine ai globuli
bianchi
Mappa genetica
Rappresentazione grafica della posizione dei geni lungo i cromosomi,
ottenuta in base ai dati di ricombinazione genetica.
Metafase
Stadio della mitosi o della meiosi in cui i cromosomi si dispongono sul
piano equatoriale della cellula e hanno una morfologia ben definita
Mitosi
Divisione di un nucleo in due nuclei figli geneticamente identici.
Corrisponde alla fase M del ciclo cellulare.
Nucleo interfasico
Nucleo durante le fasi del ciclo cellulare in cui non c'è divisione. Non
sono visibili i cromosomi
Nucleosoma
Unità fondamentale di condensazione della cromatina, costituita da DNA
avvolto attorno a otto molecole di istoni
Nucleotide
unità base del DNA e dell'RNA, formato da gruppo fosfato, zucchero e
base azotata
Polimero
Molecola di grandi dimensioni costituita dall’aggregazione di molecole
più semplici chiamate monomeri
Primer (innesco)
oligonucleotide di DNA o RNA al quale vengono aggiunti nuovi
nucleotidi nel corso della sintesi di un filamento di DNA
Procarioti
Microrganismi unicellulari le cui cellule sono prive di un nucleo avvolto
da membrana
Rinaturazione DNA
Processo in cui si forma la doppia elica del DNA dopo la denaturazione
Soluzione ipotonica
Soluzione che ha concentrazione del soluto minore di quella di una
soluzione di riferimento
Sonda
Frammento di DNA o RNA, marcato chimicamente o radioattivamente, a
sequenza conosciuta, usato per localizzare sequenze specifiche di acidi
nucleici mediante ibridazione
Surnatante
Sostanza che affiora dopo la centrifugazione e che è possibile separare
A cura di:
• Prof.ssa Silvana Dolfini Faccio, Dipartimento di Biologia e Genetica per le Scienze Mediche,
Università degli Studi di Milano;
• Prof.ssa Paola Riva, Dipartimento di Biologia e Genetica per le Scienze Mediche, Università degli
Studi di Milano;
• Prof.ssa Patrizia Barbaccia, Liceo Scientifico Cremona, Milano
• Prof.ssa Raffaella Giordano, Liceo Scientifico Cremona, Milano
• Prof.ssa Marina Porta, Liceo Scientifico Banfi, Vimercate
• Prof.ssa Paola Pozzi, Liceo Scientifico Cremona, Milano
26