25/03/2015
Proteomica: Gel
bidimensionale e Sistemi
di Spettrometria di Massa
DNA
RNA
GENOMICA
Mantenimento e
trasmissione
Trasferimento
informazione
informazione
L’informazione di base
di un organismo è uguale
per tutte le sue cellule
L’utilizzo dell’informazione
è differenziale a seconda
del tipo di cellula e del particolare
contesto biologico preso
in considerazione
PROTEINE
PROTEOMICA
Effettori
molecolari
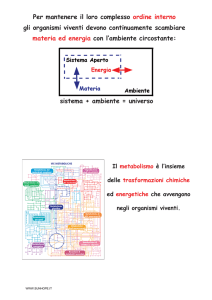
Il complemento proteico è differente
a seconda del tipo di cellula e
del particolare
contesto biologico preso
in considerazione
WWW.SUNHOPE.IT
1
25/03/2015
Gestione dell’immagine
(scanner,etc)
dalla tecnologia dei
DNA array
Colorazioni proteiche in gel
(Coomassie, argentiche,
Protein array
Derivatizzazioni Chimiche
Fluorofori differenziali (2D)
(nanotecnologie)
(scanner con lapossibilità di
illuminare i gel
a determinate lunghezze d’onda)
Metodologie di studio per
le interazioni proteina-proteina
2D gel
Proteomica
Spettrometria di massa
MS Protein sequencing
Derivatizzazioni Chimiche
ICAT (MS) -shotgun proteomics
Cromatografia
RP-HPLC / Multi dimensional chromatography
(Miniaturalizzazione colonne)
Preparazione del campione
sviluppo di detergenti, agenti riducenti, etc
Robotica
(Dalla preparazione del campione
all’interpretazione dei dati)
Bioinformatica
Banche dati
Swiss-prot. etc
Peptide fingerprinting
Sequenziamento genomi
DNA RNA
Proteine
La cellula
GENOMA
Funzionamento del macchinario
cellulare (enzimi, proteine strutturali)
PROTEOMA
WWW.SUNHOPE.IT
2
25/03/2015
A che cosa serve la
proteomica?
Sicuramente a complicarci
la vita!!!!
Quali sono le domande alle quali la Proteomica può dare una risposta
Quali proteine sono espresse in un determinato tipo cellulare/tessuto/organo/organismo
Quali proteine sono differenzialmente espresse oppure modificate (sia in
termini qualitativi che quantitativi) confrontando due (o più) fenotipi
cellulari
Un esempio di diversi fenotipi cellulari potrebbe essere:
• Cellule di origine diversa;
• Cellule dello stesso tipo sottoposte a stimoli diversi;
• Cellule dello stesso tipo soggette o meno a condizioni patologiche.
IDENTIFICARE PROTEINE CARATTERISTICHE di UNA
DETERMINATA CONDIZIONE CELLULARE
MARKERS
?
FUNZIONE
WWW.SUNHOPE.IT
3
25/03/2015
Quali sono le domande alle quali la Proteomica può dare una risposta
Quali proteine sono differenzialmete soggette ad una particolare PTM
confrontando fentotipi cellulari diversi: studio delle modifiche posttraduzionali
Fosfoproteoma
Metilproteoma
Acetilproteoma
Ubiquitinproteoma
…
Stabilire l’insieme di tutte le proteine soggette alla medesima
modificazione post-traduzionale e seguirne i cambiamenti in risposta a
determinate condizione
Identificare pathway specificamente attivate in seguito ad uno stimolo
tramite l’identificazione dei substrati
Risponde a domande cui non può rispondere lo studio del materiale genetico: perché ci
sono modifiche che avvengono dopo la trascrizione e la traduzione e che sono
importanti per la determinazione delle funzioni cellulari.
Post-Translational Maturation of Ras
GDP
S
GDP
C
S
C-(Me)
a1a2X
GDP
S
Ca1a2X
GDP
SH
CaaX
WWW.SUNHOPE.IT
4
25/03/2015
HMG Co-A
Statine
Mevalonato
Colesterolo
FPP Sintasi
Farnesil-PP
Geranil-Geranil-PP
Membrana Plasmatica
Farnesil-transferasi
Geranil-geranil
transferasi
RAS
RAS
2D gel electrophoresis and Western Blotting for ras
M. Caraglia et al., unpublished results
WWW.SUNHOPE.IT
5
25/03/2015
Quali sono le domande alle quali la Proteomica può dare
una risposta
Quali proteine costituiscono/sono presenti in un
determinato complesso macromolecolare/organello
IDENTIFICAZIONE
RUOLO NEL COMPLESSO/ORGANELLO
MODULAZIONE ATTIVITA’ COMPLESSO/ORGANELLO
ATTRAVERSO PROTEINA
Quali sono le domande alle quali la Proteomica può dare
una risposta
Quali proteine interagiscono con una determinata proteina (X)
Determinazione del network molecolare di una proteina (X)
IDENTIFICAZIONE
FUNZIONE DELL’INTERAZIONE
MODULAZIONE ATTIVITA’ DELLA PROTEINA (X)
WWW.SUNHOPE.IT
6
25/03/2015
WWW.SUNHOPE.IT
7
25/03/2015
Approccio proteomico
Prefrazionemento
Preparazione del Campione
Separazione delle proteine
Rilevazione delle proteine
Identificazione
ELETTROFORESI
BIDIMENSIONALE
Cerca una variazione di A) espressione o
di
B)
modifica
strutturale
posttrascrizionale di una proteina, ma non
necessariamente da sola è in grado di
identificare “quale proteina è” e/o quale
“modifica post-trascrizionale è”.
WWW.SUNHOPE.IT
8
25/03/2015
Preparazione del Campione
Requisiti:
=> Riflettere la composizione proteica della cellula/tessuto in indagine
=> Evitare contaminazioni
=> Evitare modificazioni
Agenti caotropici:
UREA-TIOUREA permettono alle proteine di destrutturarsi mostrando il loro core idrofobico,
quindi servono a mantenere in soluzione le proteine idrofobiche
Agenti surfattanti:
CHAPS per solubilizzare i residui idrofobici..
Agenti riducenti non carichi (no DTT)
Tributilfosfina (TBP) Necessari per rompere i ponti disolfuro S-S e mantenere in forma ridotta le proteina.
Non carico: la solubilità proteinca aumenta di molto e si migliora il trasferimento.
Iodoacetamide Ridurre ed alchilare la proteina prima della IEF.
Rimozione degli acidi nucleici:
ANFOLITI CARRIER: piccole molecole organiche polimeriche, altamente solubili, formano complessi con gli
acidi nucleici che vengono rimossi dopo ultracentrifugazione.
Sono molecole che vengono utilizzate per creare gradienti di pH. A seconda dell’intervallo di pH scelto per
l’analisi di isolettrofocusing vengono selezionate miscele di anfoliti su misura (IPG buffer - immobilzed pH
gradient buffer).
Rimozione degli acidi nucleici:
Sali precipitazione
Lipidi etanolo/acetone
Proteasi pH
ISOELETTROFOCALIZZAZIONE
PRIMA DIMENSIONE
SESSSKSSQP LASKQEKDGT EKRGRGRPRK QPPVSPGTAL VGSQKEPSEV PTPKRPRGRP
Number of amino acids: 106
Molecular weight: 11544.8
KGSKNKGAAK TRKTTTTPGR KPRGRPKKLE KEEEEGISQE SSEEEQ
1 Nterm + 11 R + 1 D + 14 E + 16 K + 1 Cterm
Theoretical pI: 10.31
Amino acid composition:
Ala (A) 4
Arg (R) 11
Asn (N) 1
Asp (D) 1
Cys (C) 0
Gln (Q) 6
Glu (E) 14
Gly (G) 11
His (H) 0
Ile (I) 1
Leu (L) 3
Lys (K) 16
Met (M) 0
Phe (F) 0
Pro (P) 13
Ser (S) 14
Thr (T) 8
Trp (W) 0
Tyr (Y) 0
Val (V) 3
3.8%
10.4%
0.9%
0.9%
0.0%
5.7%
13.2%
10.4%
0.0%
0.9%
2.8%
15.1%
0.0%
0.0%
12.3%
13.2%
7.5%
0.0%
0.0%
2.8%
algoritmo
pI: pH al quale la carica netta della proteina = 0
Se sottoposta all’azione di un campo elettrico
non si muove
Focalizzazione - CONCENTRAZIONE
WWW.SUNHOPE.IT
9
25/03/2015
ddp
pI = 7.4 0
pI = 8.4 0
-
+
0
+
-
-
0
+
pH10
pH3
pH = 7.4
pH = 8.4
Una proteina dispersa in un gradiente di pH, si troverà ad avere carica netta positiva, negativa oppure nulla (se si
trova gia ad un pH pari al suo pI). Sottoposta all’azione di un campo elettrico opportunamente orientato essa si
muoverà, a seconda della carica che reca verso l’elettrodo di segno opposto, fino a raggiungere il pH pari al suo pI.
In questo punto essa assume carica netta nulla e non è più sottoposta all’azione del campo elettrico. Se per una
qualsiasi ragione essa si muove a dx o a sx, allontanandosi dalla regione dove pH=pI, essa assume una carica e
viene nuovamente focalizzata. => Questo conferisce l’alta risoluzione che si ha nelle analisi di
isoelettrofocalizzazione. E’ doveroso ricordare che più distante una proteina si trova dal suo pI (in termini di pH)
maggiore sarà la sua carica e dunque anche la sua mobilità elettroforetica, mano a mano che essa si avvicina al pH
pari al suo pI, la sua carica netta diminuisce e di conseguenza diminuisce anche la sua mobilità elettroforetica => il
processo di focalizzazione è un processo che solitamente richiede tempi lunghi, proprio per questo fatto!
Gradienti di pH immobilizzati: reidratazione
I gel recanti i gradienti di pH immobilizzati sono di solito deidratati e devono essere reidratati in opportune
condizioni:
Si reidratano in modo da poter condurre in seguito la corsa elettroforetica:
Il metodo più comunemente usato prevede che i gel vengano messi a contatto con un volume specifico (a seconda
delle loro dimensioni) di una soluzione di reidratazione:
8 M Urea (o in alternativa la composizione usata per il lysis buffer), 4-0,5 % CHAPS, 0,2 % DTT (o altri agenti
riducenti), 0,5% (fino a 2% nel caso di strip da 24 cm) IPG buffer, 10% glicerolo e 0,002% BBF.
Fenomeno dell’ elettroendosmosi:
Nel caso ci siano cariche fisse (in particolare cariche negative (-) sulla matrice che forma il gel queste non possono
migrare in un campo elettrico ma hanno solitamente dei controioni (possono essere dei cationi con la loro sfera
d’idratazione o stessi H3O+) che migrano verso il catodo creando un vero e proprio flusso di acqua. (fenomeno che
portava a dei problemi notevoli utilizzando gradienti di pH non immobilizzati => distorsione/spostamento del
gradiente di pH). Il glicerolo aumentando la densità del mezzo diminuisce/minimizza questo effetto - importante
anche durante il trasferimento delle proteine dalla prima alla seconda dimensione. Tale fenomeno si sfrutta in modo
positivo nell’elettroforesi capillare (elettroforesi condotta in assenza di una matrice solida reticolante, ovvero in
soluzione libera), dove crea un vero e proprio flusso in grado di trasportare anche molecole con carica opposta verso
il catodo).
Flusso di acqua
anodo
+ + + +
- - - -
+
pH3
+ +
- -
+ ++ + +
- - - - -
+
-
+
-
catodo
pH10
WWW.SUNHOPE.IT
10
25/03/2015
Seconda dimensione: SDS PAGE
Separazione elettroforetica che discrimina le varie proteine in base al loro PESO MOLECOLARE
Sodio dodecilsolfato (SDS)
L’SDS si lega alle proteine mediante la sua
porzione idrofobica e rompe sia le strutture
secondaria che terziaria (non i legami
disolfuro). Ogni molecola di SDS possiede
una carica negativa e su ogni mg di proteina
si legano circa 1,4 mg di SDS [equivale a
dire che c’è una molecola di SDS circa ogni
2 aa]. L’elevata carica negativa (la forza di
repulsione che le cariche negative esercitano
fra di loro) conferita alla catena polipetidica
la rende praticamente lineare. L’elevata
carica negativa conferita dall’SDS rende
trascurabile la carica propria della proteina.
Dato che l’SDS si lega con la stessa
stechiometria a tutte le proteine il rapporto
massa/carica è uguale per tutte le proteine.
Questo fa si che sia esclusivamente
l’ingombro sterico ad influenzare la
migrazione (mobilità elettroforetica) in una
matrice a porosità controllata (gel di
poliacrilammide)
con
applicata
una
differenza di potenziale.
Orientamento canonico della seconda dimensione
pH minore
pH maggiore
pH minore
pH maggiore
PM minore
PM minore
PM minore
PM minore
Supporto plastica
1a dimensione
Supporto plastica
2a dimensione
Supporto plastica
2a dimensione
Supporto plastica
1a dimensione
WWW.SUNHOPE.IT
11
25/03/2015
Metodi per la rilevazione di spot proteici
Requisiti ideali per un metodo di rilevazione di spot proteici da 2D
Alta sensibilità
Rapido
Permettere analisi quantitative
Economico
Compatibile con MS
Non tossico
WWW.SUNHOPE.IT
12
25/03/2015
2D-DiGE
(2-D fluorescence difference gel electrophoresis)
La colorazione (labelling)
avviene prima della corsa
elettroforetica
Campioni proteici
“marcati” in modo
differenziale possono
essere separati sul
medesimo gel
bidimensionale
- minimizzare veariazioni
gel-gelImpostando le opportune
lunghezze d’onda d’eccitazione
e di emissione è possibile
visualizzare le proteine
derivanti dal campione 1, quelle
dal campione 2 oppure ottenere
un’immagine relativa alla
sovrapposizione dei due
campioni
Derivatizzazione delle proteine
con molecole fluorescenti
(aventi differenti ecc. & emiss.)
Separazione proteine
tramite 2D gels
Visualizzazione mediante
speciali scanner
WWW.SUNHOPE.IT
13
25/03/2015
IDENTIFICAZIONE
DEGLI SPOTS
Segue necessariamente alla elettroforesi
bidimensionale ed ha lo scopo di
identificare le proteine e le loro modifiche
post-trascrizionali!!!!
Si esegue un vero e
proprio
identikit
molecolare
SPOT PICKER
Queste strumentazioni possono operare anche
modo semi-automatico - controllo visivo
dell’operatore sugli spots prelevati -
Sistemi di lavaggio evitano
cross-contaminazioni
WWW.SUNHOPE.IT
14
25/03/2015
Digestione enzimatica
SPOT
PEPTIDI
MS
Peso Molecolare dei singoli peptidi
PEP1
PRO
PEP3
PEP4
PEP5
PEP2
Insieme di pesi molecolari
623.6
Insieme
816.9
caratteristico
548.5
di una
1465.7
determinata
427.5
proteina
....
PROTEINA: caratterizzata da una sua particolare e unica sequenza aa
PEPTIDE: caratterizzato da una sua particolare e unica sequenza aa
PEPTIDE: sequenza aa specifica => peso molecolare specifico
Assorbimento su Reversed Phase (C18) di peptidi generati da in gel digestion
ZipTip: puntali per pipette eppendorf alla cui punta è
impaccata una resina che supporta una fase inversa (tipo
C18) sulla quale, dopo che i peptidi eluiti dall’in-gel
digestion (sciolti in ambiente acquoso), gli stessi peptidi
si legano. E’ possibile, con i peptidi legati alla resina,
effettuare una serie di lavaggi in modo da eliminare
eventuali contaminanti ed eluire successivamente i
peptidi. (Si adottano le stesse strategie di eluizioni che si
adottano in cromatografia HPLC a fase inversa. Questa
procedura viene utilizzata soprattutto quando si hanno
piccoli volumi e si vuole eliminare la presenza di sali.
WWW.SUNHOPE.IT
15
25/03/2015
WWW.SUNHOPE.IT
16
25/03/2015
ESI Electrospray ionization
Una soluzione con l’analita viene nebulizzato tramite un capillare a pressione atmosferica in un forte
campo elettrico. Il solvente residuo viene evaporato e l’analita ionizzato viene introdotto
nell’analizzatore di massa ad alto vuoto.
ANALIZZATORI:
QUADRUPOLO,
IONTRAP
TOF
MALDI matrix assisted laser desorption ionization
Il raggio laser viene sparato su una miscela di matrice/analita cristallizzata su una superficie di
metallo. La matrice ha la funzione di assorbire l’energia del laser e trasmetterla in modo attenuato
alle molecole da analizzare. La formazione degli ioni di analita avviene con l’adesione dei cationi
come H+ o Na+.
(UV o IR)
ANALIZZATORI:
TOF
WWW.SUNHOPE.IT
17
25/03/2015
TOF
Time Of Flight
FT-ICR
Fourier Transfor
Ion ciclotron
resonance
Quadrupolo
ANALIZZATORE
Di MASSA
Trappole ioniche
Orbitrap
Magnetici
Discriminare molecole in base al loro
Rapporto massa/carica
m/z
(NB: informazioni circa il peso molecolare (m) si possono avere solo ed
esclusivamente se si hanno informazioni circa lo stato di carica
di quella determinata molecola (z))
Analizzatori a tempo di volo - TOF
Tubo di Volo (d)
Regione di accelerazione
V
Detector
Nella regione di accelerazione, gli
ioni vengono sottoposti ad una forte
differenza di potenziale che
impartisce ad ognuno degli ioni una
propria velocità che dipende dal
valore del rapporto massa/carica e
quindi il tempo per percorrere la
distanza tra sorgente e rilevatore è
differente. Misurando il tempo di volo
(Tempo che lo ione impiega per
percorrere il tubo di volo) e
calibrando con ioni di noto valore
massa/carica, è possibile risalire al
peso molecolare dell’analita.
WWW.SUNHOPE.IT
18
25/03/2015
Generazione degli ioni – Ad esempio tramite un impulso laser (MALDI)
Tubo di Volo (d)
Accelerazione degli ioni mediante V
V
Tubo di Volo (d)
WWW.SUNHOPE.IT
19
25/03/2015
Ioni hanno acquisito una propria velocità
in dipendenza del loro m/z e si muovono
verso il detector
Le loro diverse velocità separano progressivamente gli ioni con diverso m/z
durante il loro cammino verso il detector
WWW.SUNHOPE.IT
20
25/03/2015
Le loro diverse velocità separano progressivamente gli ioni con diverso m/z
durante il loro cammino verso il detector
(m/z)3
(m/z)2
(m/z)1
Gli ioni con m/z diversi raggiungono il detector in tempi diversi
(m/z)3
(m/z)2
(m/z)1
Ione (m/z)1
rilevato
WWW.SUNHOPE.IT
21
25/03/2015
V
d3
d2
d1
Gli ioni prodotti in sorgente non partono tutti dalla medesima posizione
=>
Sono soggetti ad accelerazione differenti
=>
Si muovono con velocità differenti
=>
Pur avendo m/z uguale, raggiungono il detector in tempi diversi
=>
Risoluzione strumentale bassa
Gli ioni con m/z uguali raggiungono il detector in tempi leggermente diversi
a causa della loro differente accelerazione iniziale
dispersione
(m/z)3
(m/z)2
(m/z)1
Ione (m/z)1
rilevato
WWW.SUNHOPE.IT
22
25/03/2015
Analizzatore TOF con reflectron che funge da specchio
ionico
Il reflector migliora la risoluzione estendendo il percorso di volo dello ione.
Ioni aventi m/z uguali risultano avere velocità differenti. Avendo energie cinetiche
differenti penetrano in modo differenziale all’interno di un campo elettrico riflettente.
All’uscita del campo elettrico riflettente tutti gli ioni aventi un determinato valore m/z
risultano raggiungere il detector al medesimo istante: focalizzati sul detector.
Riflettore elettrostatico
dispersione
Gli ioni accelerati si muovono con velocità diverse verso il reflectron
Riflettore elettrostatico
WWW.SUNHOPE.IT
23
25/03/2015
Gli ioni accelerati si muovono con velocità diverse verso il reflectron
Riflettore elettrostatico
Gli ioni accelerati si muovono con velocità diverse verso il reflectron
Riflettore elettrostatico
WWW.SUNHOPE.IT
24
25/03/2015
Gli ioni aventi lo stesso m/z ma diversa energia cinetica penetrano
differenzialemte nel riflettore. Quelli con energia cinetica maggiore in
maggior misura rispetto a quelli con energia cinetica minore.
Riflettore elettrostatico
All’uscita dal reflectron, le velocità sono le stesse rispetto all’entrata,
ma lo ione con velocità maggiore si trova dietro a quello con velocità
minore.
Riflettore elettrostatico
WWW.SUNHOPE.IT
25
25/03/2015
Riflettore elettrostatico
Riflettore elettrostatico
WWW.SUNHOPE.IT
26
25/03/2015
Riflettore elettrostatico
Qualora il ritardo dato dal reflectron è opportunamente tarato, i due
ioni, aventi comunque velocità diverse, raggiungono il detector in
contemporanea.
Riflettore elettrostatico
WWW.SUNHOPE.IT
27
25/03/2015
Spettrometri di massa: MALDI-TOF e ESI-TOF
MALDI-TOF
ESI-TOF
PEPTIDE MASS FINGERPRINTING
Una proteina, digerita da un enzima proteolitico noto - è nota quindi la specificità di taglio - dà origine ad una
serie di peptidi e ciascuno di essi è caratterizzato da uno specifico peso molecolare.
Anche se peptidi che presentano la stessa composizione aa ma sequenza diversa presentano pesi molecolari uguali
è statisticamente poco probabile che da proteine diverse si generino più peptidi che presentano la stessa
composizione aa ma diversa sequenza.
Questo significa che l’insieme dei pesi molecolari dei peptidi generatisi dalla digestione endoproteolitica di una
proteina risulta essere in pratica una sola e specifica per quella proteina
IMPRONTA DIGITALE
IDENTITIFICAZIONE (DATABASE)
L’identificazione risulterà più certa quanto maggiore è l’accuratezza con la quale viene determinato il peso
molecolare dei singoli peptidi e quanto maggiore è la copertura della intera sequenza della proteina di partenza
(sequence coverage)
WWW.SUNHOPE.IT
28
25/03/2015
WWW.SUNHOPE.IT
29
25/03/2015
Spettrometria di Massa Tandem (MS/MS)
La spettrometria di Massa tandem permette, senza l’ausilio di separazioni
cromatografiche, l’analisi di miscele di prodotti o di molecole complesse che
presentano frammentazioni assai complicate.
Il primo analizzatore separa ioni ad alta massa (generati da una sorgente soft) tipici
di ogni composto della miscela; detti ioni vengono sequenzialmente introdotti in
una cella a collisione con atomi di He (seconda sorgente di ionizzazione), gli ioni
ulteriormente frammentati vengono avviati al secondo analizzatore e
successivamente al rivelatore.
Un’applicazione abbastanza frequente della spettrometria di massa tandem
è quella che vede l’impiego di tre quadrupoli, il secondo dei quali funge da
cella di collisione con He.
L’accoppiamento MS/MS può esser utilizzato come:
1. Selezione del primo analizzatore su massa singola (es: ione molecolare
protonato) e scansione del secondo analizzatore su un intervallo m/z prefissato
(MS/MS di ioni figli). Serve per discriminare composti chimicamente dissimili
ma dotati di pari
peso molecolare.
2. Scansione del primo analizzatore su un intervallo m/z prefissato e selezione del
secondo analizzatore su un rapporto m/z prefissato di uno ione figlio (MS/MS di ioni
progenitori). Serve per misurare la concentrazione di membri di una classe
simile.
WWW.SUNHOPE.IT
30
25/03/2015
Gli ioni sono sottoposti ad una ddp, subiscono delle
oscillazioni stabili permettendo allo ione di fuoriuscire
dal quadrupolo, o instabili portando lo ione in collisione
con le barre metalliche. Solo gli ioni aventi un preciso
rapporto massa/carica usciranno dal quadrupolo stesso.
WWW.SUNHOPE.IT
31
25/03/2015
Trappola ionica
Gli ioni sono intrappolati tra due elettrodi
e un elettrodo anello. Variando il
potenziale elettrico e la frequenza del
campo elettrico si espellono in sequenza
gli ioni secondo un valore mass/carica
crescente
WWW.SUNHOPE.IT
32
25/03/2015
PROTEOMICA ALTERNATIVA
ICAT (ISOTOPE-CODED AFFINITY TAG)
Consente l’analisi differenziale quantitativa di due campioni. Le proteine del campione
vengono marcate ai residui di cisteina con etichette di iodoacetamide-biotina con diversi
isotopi (idrogeno/deuterio, C12/C13). Le proteine vengono miscelate e digerite e i peptidi
marcati vengono separati da quelli non marcati tramite cromatografia di affinità seguita da
MS con ionizzazione ESI.
MudPIT
Tecnologia di identificazione multidimensionale delle proteine: utilizza una separazione
cromatografica in due stadi di tutti i peptidi derivanti dalle miscele proteiche digerite con
successiva analisi MS.
SELDI-TOF
Consente di rilevare le differenze nel pattern di espressione delle proteine ottenuti mediante
MALDI-TOF-MS delle proteine legate a del materiale cromatografico spottato su un target
della MALDI.
WWW.SUNHOPE.IT
33