GENETICA
GENETICA = è la scienza che studia:
STRUTTURA
FUNZIONE
EREDITARIETA’
del materiale genetico e della informazione genetica che esso porta.
STRUTTURA = Qual è il supporto fisico (la base) dell’informazione genetica;
equivale a dire com’è fatto il materiale che porta l’informazione
genetica (MATERIALE GENETICO)
FUNZIONE = Quale tipo di informazione porta il materiale genetico e con quali modalità
questa informazione si esprime;
INFORMAZIONE GENETICA
CARATTERE
EREDITARIETA’ = Come si trasmette l’informazione genetica:
- da cellula a cellula ( trasversalmente);
- da generazione a generazione ( verticalmente ).
Osservazione di partenza: non tutti gli organismi sono uguali. Tra i diversi organismi è
osservabile:
variabilità interspecifica ( procarioti – eucarioti; vegetali – animali; funghi – piante
superiori; piante arboree – piante erbacee; graminacee – leguminose; frumento – mais;
frumento duro – frumento tenero );
variabilità intraspecifica ( anche tra individui strettamente imparentati come tra
genitori e figli );
nell’insieme: esiste una diversità (variabilità) biologica di cui è responsabile la
diversità (variabilità) dell’informazione genetica portata dai diversi organismi.
Origine della variabilità genetica:
modificazioni trasmissibili (ereditarie) e più o meno rilevanti del materiale genetico
(informazione genetica) = mutazione;
riassetto del materiale genetico (informazione genetica) da una generazione alla
successiva (assortimento casuale e crossing over) = ricombinazione.
Sulla variabilità che si origina agisce la selezione naturale innescando il processo
della evoluzione.
1
Il materiale genetico deve quindi godere di alcune proprietà che devono essere compatibili
con:
continuità delle caratteristiche specifiche (continuità della specie);
modificazioni evolutive.
Il materiale genetico deve perciò:
portare informazione biologicamente utile mantenuta in forma stabile (non deve
essere soggetto a modificazioni frequenti);
consentire il mantenimento e la trasmissione fedele dell’informazione (da cellula a
cellula e da generazione a generazione);
consentire che l’informazione genetica si esprima (possa essere tradotta) in
carattere;
poter variare per consentire l’evoluzione (mutazioni, anche se non frequenti, e
ricombinazione).
Fino agli anni ’40 si riteneva che le proteine potessero soddisfare a queste esigenze
perché molecole complesse; questa loro caratteristica – la complessità strutturale – non
era però sufficiente.
CENNI SULLA STRUTTURA DELLE MOLECOLE PROTEICHE.
AMINOACIDI = numero e tipo
STRUTTURA delle PROTEINE
primaria = sequenze aminoacidiche
secondaria =
elica; configurazione
terziaria = legami H; forze di Van der Waals; legami disolfuro:
- S – S-
quaternaria = dimeri e strutture più complesse; gruppi prostetici
Dalla configurazione assunta dalle molecole proteiche, determinata dalla loro struttura,
dipende spesso la loro capacità di svolgere la funzione che è loro propria. Emblematico il
caso delle proteine enzimatiche.
Dopo un serie di esperimenti condotti tra il 1928 e il 1953 fu raggiunta la conclusione che
IL MATERIALE GENETICO E’ COSTITUITO DAGLI ACIDI NUCLEICI E NON DALLE
PROTEINE.
2
DNA: elementi essenziali
1 - molecola costituita da due strutture (eliche, nastri, filamenti) avvolte una sull’altra
in una configurazione più complessa che e’ la doppia elica.
2 - ogni elica e’ un polinucleotide (polimero): e’ formata da più nucleotidi tenuti insieme dai
legami fosforo – zucchero (legame covalente, stabile).
3 – un nucleotide è costituito da una molecola di zucchero a 5 atomi di carbonio (pentoso)
che, nel DNA, è il deossiribosio, un gruppo fosfato legato in posizione 5’ e una
base azotata portata in posizione 1’.
4 - la successione fosforo – zucchero costituisce l’ossatura del polinucleotide e si sviluppa,
per ciascuna delle due eliche, con polarità 5’ - 3’ : le due eliche sono quindi
antiparallele. Questo vuol dire che percorrendo una delle due eliche secondo una
direzione (5’
3’) l’altra viene percorsa nella direzione opposta (3’
5’).
5 - su questa struttura, situata esternamente alla molecola, si inseriscono le basi azotate
adenina, guanina (basi puriniche o purine), timina e citosina (basi pirimidiniche o
pirimidine) le quali si affacciano verso l’interno della doppia elica.
6 - tra purine ( A o G ) e pirimidine ( T o C ) di un’elica e le pirimidine e le purine,
rispettivamente, dell’elica antiparallela si stabiliscono dei legami a idrogeno che
tengono unite le due eliche ( giustificazione della costanza del diametro della
doppia elica).
7 – l’appaiamento tra purine e pirimidine non è casuale ma avviene secondo una
corrispondenza ben precisa e costante : Adenina – Timina ( A = T ) e Guanina –
Citosina ( G ≡ C ); il numero di legami è pari a due nel primo caso e a tre nel
secondo; l’unione tra Adenina e Timina è quindi meno stabile di quella tra Guanina
e Citosina e, per essere rotta, richiede un minore dispendio di energia.
8\ - per quanto detto, le due eliche sono complementari e la successione delle basi
diun’elica determina la successione delle basi dell’altra. Questa rigida e, nello
stesso tempo, variabile successione di basi (nucleotidi) lungo la molecola spiega sia
la diversita’ dell’informazione genetica (determinata dalla successione delle basi
lungo l’elica) sia la possibilita’ di mantenere inalterata tale informazione nel corso
della divisione cellulare.
_°_°_°_°_°_°_°_°_°_°_
3
Duplicazione del DNA
La duplicazione del DNA è definita semiconservativa in quanto ciascuna delle due
eliche da cui è costituita la doppia elica funge da stampo per la sintesi dell’elica
complementare, dando così luogo alla formazione di due molecole complete (due doppie
eliche) ciascuna delle quali è costituita da un’elica vecchia e da un’elica di nuova sintesi.
La duplicazione del DNA avviene secondo il modello sintetizzato qui di seguito:
1 – DENATURAZIONE E SVOLGIMENTO della doppia elica.
Proteina iniziatrice + DNA elicasi
Sequenza di origine
idrolisi ATP
energia per l’avanzamento
Forse 2 elicasi, una per l’elica LEADING (guida) e una per l’elica LAGGING (lenta)
2 – STABILIZZAZIONE del DNA a singola elica nella forca replicativa.
Proteine SSB (destabilizzatrici della doppia elica) (*)
(*) SSB proteins = Single Strand Binding Proteins: si uniscono alla singola
elica di DNA e ne stabilizzano la conformazione stesa in modo da
renderla disponibile alla copiatura
3 – INIZIO DELLA SINTESI di un nuovo filamento di DNA
DNA elicasi + Primasi
Primosoma
RNA Primer
POLIMERASI III
DNA
La necessità di mantenere inalterata la polarità 5’ – 3’ delle due eliche di nuova
formazione fa si che la direzione di sintesi sia diversa sui due filamenti stampo.
Elica leading = sintetizzata nella direzione in cui si muove la forca di replicazione;
Elica lagging = sintetizzata in direzione opposta ( sintesi discontinua).
4 – ALLUNGAMENTO delle eliche di DNA di nuova sintesi.
La sintesi dell’elica leading avviene in maniera continua.
La sintesi dell’elica lagging avviene fino a che è disponibile DNA stampo. A questo
punto il processo riprende sul tratto di DNA che nel frattempo si è svolto, previa sintesi di
un nuovo primer di RNA ad opera della primasi e successivo intervento della Polimerasi III
4
5 – GIUNZIONE dei frammenti di Okazaki dell’elica lagging per formare un filamento
continuo; questa operazione avviene in due fasi:
a) DNA Polimerasi I = continua l’azione della Polimerasi III che si è staccata e,
contemporaneamente, rimuove dalla estremità 5’ del primer del frammento
precedentemente formato i ribonucleotidi fino a lasciare un’interruzione di una
base tra due nucleotidi adiacenti (azione esonucleasica 5’ – 3’ )
b) DNA Ligasi che opera la saldatura dando luogo alla formazione di una unica
molecola di DNA.
La trascrizione
Trascrizione = consiste nel trasferimento dell’informazione genetica da una sequenza di
basi nel DNA ad una sequenza di basi nell’RNA.
E’ il primo dei due passi che costituiscono quello che Crick (1956) propose come Dogma
Centrale della Biologia Molecolare:
Trascrizione
DNA
Traduzione
RNA
PROTEINA
La trascrizione è anche il primo passo di quel processo che consente all’informazione
genetica contenuta in ben definite sequenze di basi di tradursi in caratteri. La trascrizione
è quindi indispensabile per consentire l’espressione genica.
La trascrizione interessa solamente una delle due eliche di DNA; per questo motivo, prima
che la trascrizione inizi, la doppia elica di DNA si deve svolgere in una regione vicina al
gene.
L’elica di DNA che viene trascritta prende il nome di elica stampo
Essa viene letta in direzione 3’ – 5’ in modo che il filamento di RNA possa essere
sintetizzato in direzione 5’ – 3’.
Elica stampo e filamento di RNA (trascritto) hanno quindi polarità inversa.
L’RNA che viene sintetizzato ha invece la stessa polarità della seconda elica di DNA la
quale prende il nome di elica codificante (5’ – 3’).
La trascrizione avviene ad opera di un enzima (RNA-polimerasi) il quale ha il compito di
scegliere tra i ribonucleosidi trifosfati (ribonucleotidi) disponibili (ATP, GDP, CTP, UTP)
quello che, di volta in volta, deve essere inserito in base al criterio di complementarietà
con le basi presenti sull’elica stampo.
5
Tale criterio è analogo a quello già visto per la duplicazione del DNA, con la variante che la
pirimidina Timina (T), complementare alla purina Adenina (A), viene sostituita dalla pirimidina
Uracile (U). Perciò:
elica DNA stampo :
3’ – A T A C G A A G C – 5’ (trascritta)
trascritto di RNA :
5’ – U A U G C U U C G – 3’
elica DNA codificante :
5’ – T A T G C T T C G – 3’ (non trascritta)
Classi di RNA
Con la trascrizione vengono prodotti quattro tipi di RNA:
RNA messaggero (mRNA)
RNA di trasferimento (tRNA)
RNA ribosomale (rRNA)
RNA piccolo nucleare (snRNA) (solamente negli eucarioti; coinvolto
nel processamento dell’mRNA).
Solamente l’RNA messaggero (mRNA) viene tradotto in proteina. Gli altri RNA sono da
vedere come “strumenti” della traduzione e funzionano come tali, svolgendo nella cellula
compiti diversi.
L’mRNA deriva perciò dalla trascrizione dei cosiddetti “geni strutturali” o geni che
codificano polipeptidi (PROTEINE).
Trascrizione dei geni strutturali
Un gene strutturale (che codifica per una proteina) può, in linea generale, essere suddiviso
in:
- sequenza promotore, in posizione adiacente al punto di inizio della trascrizione,
interagisce con RNA-polimerasi e segnala l’inizio della trascrizione;
- sequenza stampo, che viene trascritta dalla RNA-polimerasi in una sequenza di
RNA a singola elica;
- sequenza terminatore, adiacente alla fine della sequenza stampo e che segnala
la fine della trascrizione.
Il promotore è a monte e il terminatore è a valle del gene.
La trascrizione, almeno nei suoi tratti essenziali, è simile nei procarioti e negli eucarioti.
6
N.B. : Parlando di trascrizione, vengono spesso utilizzati i termini “a monte” e “a
valle” facendo riferimento alle regioni situate nelle vicinanze dell’estremità 5’ o 3’,
rispettivamente, del trascritto. L’uso di questi termini è giustificato dal fatto che la
sintesi di RNA avviene sempre in direzione 5’
3’. Le regioni a monte e a valle
dei geni sono sequenze di DNA che codificano per i corrispondenti segmenti 5’ e 3’
dei loro trascritti, rispetto ad uno specifico punto di riferimento.
PROCARIOTI.
Inizio della trascrizione.
Nei procarioti sono identificabili 2 regioni a monte del punto di inizio:
-35 = 5’ – TTGACA – 3’
-10 = 5’ – TATAAT – 3’ (Pribnow o TATA box)
Per quanto detto nel N.B., queste due sequenze, definite anche come sequenze
consenso, sono presenti in questa forma sull’elica codificante (non stampo) del DNA,
mentre sull’elica stampo, che viene letta in direzione 3’
5’, sono presenti come:
-35 = 3’ – AACTGT – 5’ e
-10 = 3’ – ATATTA – 5’ , rispettivamente.
La sequenza a –35 viene anche definita come sequenza di riconoscimento.
L’inizio della trascrizione si ha quando l’RNA-polimerasi si lega al promotore il quale
comprende entrambe le sequenze consenso a –35 e a –10; l’operazione avviene in fasi
successive:
a) la RNA-polimerasi oloenzima (*) si lega in modo lasco alla sequenza –35. Il DNA è a
doppia elica chiusa.
b) la RNA-polimerasi si lega più strettamente al DNA. Si ha uno svolgimento di 17 coppie di
basi della doppia elica (denaturazione) in corrispondenza della sequenza –10 che è tutta
costituita da coppie di basi A=T con due legami.
c) La RNA-polimerasi si orienta per iniziare la trascrizione del primo nucleotide situato a
valle della sequenza promotore.
(*) L’enzima RNA-polimerasi è formato da quattro catene polipeptidiche e da un quinto
polipeptide (il fattore ) il quale ha la funzione specifica di riconoscere il promotore.
N.B. = I promotori possono avere sequenze diverse. Come conseguenza, l’efficienza con la
quale l’RNA-polimerasi si lega varia a seconda del promotore e il tasso di inizio della
trascrizione non è costante. Ciò spiega perché geni diversi abbiano tassi di espressione
differenti a livello di RNA. (Una sequenza 5’ – GATACT –3’ in –10 ha un tasso di
trascrizione più basso rispetto a 5’ – TATAAT –3’)
In E. coli, oltre a promotori diversi, sono anche stati individuati fattori differenti che
intervengono nella regolazione dell’espressione genica.
7
Allungamento della catena di RNA.
Alla bolla di trascrizione (DNA denaturato), dopo la polimerizzazione di 8 o 9 ribonucleotidi, il
fattore si dissocia dal nucleo enzimatico e può essere riutilizzato in un’altra molecola di
RNA-polimerasi.
In corrispondenza del tratto di doppia elica denaturato si forma quello che viene chiamato un
ibrido DNA – RNA per indicare la breve regione in fase di trascrizione, compresa nella bolla
di trascrizione, nella quale l’elica stampo che viene trascritta e il filamento di RNA in fase di
sintesi rimangono temporaneamente appaiate
Il nucleo enzimatico (core) completa la trascrizione avanzando lungo la doppia elica di DNA.
La molecola di RNA-polimerasi contiene sia l’attività svolgente sia quella riavvolgente il
DNA. Essa svolge continuamente la doppia elica di DNA a valle del sito di polimerizzazione
e riavvolge i filamenti di DNA complementari a monte, non appena si sposta lungo la doppia
elica.
In E. coli, la lunghezza media di una bolla di trascrizione è di 18 paia di basi e, in una catena
di RNA in crescita, vengono incorporati circa 40 ribonucleotidi al secondo.
La distensione (srotolamento) della doppia elica di DNA ad una estremità è compensata,
all’altra estremità dal suo superavvolgimento negativo, provocato dalla topoisomerasi I.
Terminazione della trascrizione
La fine della trascrizione è segnalata da elementi di controllo detti terminatori dei quali, in E.
coli, possono essere:
dipendenti = vengono riconosciuti da un fattore (rho) (proteina con funzioni
enzimatiche) che si associa al core della RNA-polimerasi e destabilizza l’ibrido DNA-RNA;
indipendenti = vengono riconosciuti direttamente dal core dell’enzima.
In entrambi i casi, la sintesi di RNA si blocca, l’RNA sintetizzato si allontana, l’RNApolimerasi si stacca dal DNA.
Nei procarioti la sintesi di tutti i trascritti di RNA (mRNA, tRNA, rRNA) è opera di un’unica
RNA-polimerasi.
Le molecole di RNA appena trascritte (trascritti primari) non sono separate dal sito di
sintesi proteica da una membrana nucleare. Quindi, una volta sintetizzata l’estremità 5’ di
un mRNA, essa può essere subito utilizzata come stampo per la sintesi polipeptidica.
In maniera analoga, una volta che la molecola di mRNA sia stata utilizzata per la
traduzione, può essere demolita a partire dall’estremità 5’.
Pertanto, la trascrizione, la traduzione e la demolizione dell’mRNA, nei procarioti, possono
avvenire spesso contemporaneamente.
8
EUCARIOTI
Negli eucarioti la trascrizione avviene per opera di 3 diversi tipi di RNA-polimerasi,
localizzati diversamente e con funzione differenziata:
RNA-polimerasi
Localizzazione
Prodotti
I
nucleolo
RNA ribosomali (rRNA)
28S; 18S; 5,8S
II
nucleo
Pre - mRNA nucleari+ alcuni snRNA
III
nucleo
tRNA; rRNA 5S; i rimanenti snRNA
N.B. = I valori numerici con i quali si distinguono i diversi tipi di rRNA indicano il numero di Unità
Svedberg che servono ad indicare, sinteticamente, le dimensioni, il peso molecolare, la densità
delle diverse molecole di rRna separate per centrifugazione in gradiente di saccarosio. Di tali
frazioni si misura, cioè, il tasso di sedimentazione che viene opportunamente convertito in Unità
Svedberg (o valore S) per mezzo di una formula.
Le tre RNA polimerasi, per dare inizio alla trascrizione, richiedono l’intervento di altre
proteine , dette fattori di trascrizione, le quali hanno il compito di riconoscere il promotore
e, legandosi a questo, favorire l’aggancio dell’enzima.
Regolazione della trascrizione
La trascrizione , in particolare quella dei geni strutturali, è un processo regolato, che avviene
sotto il controllo di molecole diverse che interagiscono con i cosiddetti elementi di
regolazione: sequenze coinvolte nella regolazione della trascrizione facenti parte del gene
strutturale e che possono essere situate sia a monte che a valle del punto d’inizio della
trascrizione.
Questi elementi regolatori possono essere organizzati in maniera peculiare per numero, tipo
e disposizione sul DNA, in combinazioni di elementi regolatori positivi, che attivano la
trascrizione, o negativi, che la reprimono.
A questi elementi di regolazione possono legare fattori di trascrizione (TF) specifici, richiesti
per l’inizio della trascrizione, e fattori di regolazione, cioè proteine coinvolte nell’attivazione o
repressione della trascrizione.
Questi elementi di regolazione sono generalmente localizzati entro poche centinaia di basi
dal sito d’inizio della trascrizione, ma vi sono elementi regolatori posti anche a 1000 e fino a
30.000 paia di basi dal sito d’inizio.
La regione regolatrice posta nelle immediate vicinanze del punto d’inizio della trascrizione
costituisce il promotore, mentre quelle più lontane sono i cosiddetti enhancers
(intensificatori).
9
Gli elementi del promotore in un gene strutturale eucariotico sono rappresentati da:
TATA box (elemento TATA = Goldberg – Hogness box). Ha sequenza consenso 5’ –
TATAAA – 3’ ed è posto a –30;
CAAT . Ha sequenza consenso 5’ – CGCCAATCT – 3’ ed è generalmente situato a
–80, ma funziona anche se situato in altri punti più o meno distanti;
GC . Ha sequenza consenso 5’ – GGGCGG - 3’. Può essere presente in più copie
orientate in maniera diversa.
Se gli elementi promotori sono essenziali per l’inizio della trascrizione, gli enhancers sono
importanti per intensificare la trascrizione stessa. Essi possono avere orientamento diretto o
inverso rispetto al gene e possono essere collocati anche a notevole distanza dalla sequenza
che deve essere trascritta
Perché l’enhancer possa far sentire la sua azione, il DNA deve ripiegarsi, in modo che la
sequenza enhancer si trovi vicina al promotore.
Accanto agli elementi di intensificazione (enhancers) si riscontrano anche elementi
silenziatori (silencers).
Maturazione dell’mRNA
Negli Eucarioti, la trascrizione produce una molecola di RNA (pre-mRNA o trascritto primario
o m-RNA precursore) che è molto più lunga di quella che passerà dal nucleo al citoplasma
per essere tradotta (mRNA maturo). Il pre-mRNA è infatti costituito dalla trascrizione di
introni (sequenze non codificanti) e di esoni (sequenze codificanti).
Per poter essere trasferito dal nucleo al citoplasma ed essere tradotto, il pre-mRNA deve
andare incontro a modificazioni post-trascrizionali (maturazione o processamento) come di
seguito indicato:
- alla estremità 5’ viene aggiunto un cappuccio (CAP) mediante l’operazione di
“capping”, cioè l’aggiunta di una GUANINA metilata e di due gruppi metilici (CH3) ai primi due
nucleotidi;
- alla estremità 3’ viene aggiunta una sequenza di 50 – 250 adenine (Poly A) in
prossimità della sequenza AAUAAA posta 10 – 30 nucleootidi a monte del sito di attacco;
- gli introni vengono eliminati mediante l’operazione di “splicing”, gli esoni adiacenti
vengono legati insieme per formare la molecola continua di mRNA che sarà tradotta.
Esone 1
I
Esone 1
Esone 2
Esone 2
I
Esone 3
Esone 3
I
Esone 4
Esone 4
10
Nella molecola di mRNA si distinguono:
-
sequenza leader (sequenza guida) = è più o meno lunga a seconda della molecola
di mRNA e contiene l’informazione letta dai ribosomi per orientare correttamente la
lettura e la traduzione dell’mRNA;
-
sequenza codificante = contiene l’informazione che stabilisce la sequenza
aminoacidica del polipeptide ed è più o meno lunga a seconda della lunghezza del
polipeptide stesso;
-
sequenza trailer (coda) = non viene tradotta.
La sintesi proteica: la traduzione
Per la trattazione dettagliata di questo argomento alla cui comprensione può essere di molto
aiuto, se non indispensabile, la visualizzazione, anche se schematica, della serie di eventi
molecolari che la caratterizzano, si rimanda alla consultazione del testo:
Snustad e Simmons, “Principi di genetica” , EDISES 2000; capitolo 13 (La traduzione ed il
codice genetico)
In questa sede vengono solamente elencati i punti salienti che, comunque, sono illustrati a
lezione con l’aiuto di adeguate illustrazioni.
Sintesi proteica = è il risultato della traduzione ed è rappresentata dalla serie di eventi che,
partendo dalla molecola di mRna maturo, porta alla sintesi di una molecola costituita da una
serie di aminoacidi la cui successione è determinata dalla successione delle basi sulla
molecola di mRNA. In via normale, cioè, dalla traduzione non esce una molecola proteica
funzionante, ma un polipeptide il quale, da solo o in associazione con altri polipeptidi ed
eventualmente con molecole di altra natura, si struttura in vario modo così da assumere la
configurazione di una molecola proteica dotata delle proprie funzioni strutturali o enzimatiche.
La traduzione si verifica sui ribosomi, organelli presenti in abbondanza nel citoplasma e
costituiti dalla associazione tra molecole di rRNA di diverso peso molecolare (diverso valore
di S = sedimentazione) e molecole proteiche (proteine ribosomali). I ribosomi sono formati da
due sub unità, piccola e grande, anch’esse caratterizzate da diversi valori di S, le quali sono
normalmente separate e si riuniscono solamente nel momento in cui il ribosoma deve
assumere la sua funzionalità completa.
La sequenza nucleotidica di una molecola di mRna è tradotta nell’appropriata sequenza
aminoacidica secondo le regole del Codice Genetico (vedi paragrafo seguente)
Gli eventi che si succedono nel corso della traduzione e che portano alla sintesi proteica
sono sinteticamente descritti qui di seguito.
Quando la molecola di mRNA maturo è trasferita nel citoplasma per essere tradotta, la sua
estremità 5’si lega alla sub-unità piccola del ribosoma per l’intervento di un fattore d’inizio.
11
Alla tripletta di basi AUG, sempre presente in prossimità dell’estremità 5’, si lega la molecola
di tRNAfMet con l’anticodone UAC, che porta l’aminoacido formil-metionina (metionina
formilata). Si forma così il cosiddetto “complesso di inizio”.
Al complesso d’inizio si lega la sub-unità ribosomale grande la quale è provvista di tre siti: un
sito A = aminoacilico, un sito P = peptidilico ed un sito E = di uscita.
Nel momento in cui la sub-unità grande si unisce al complesso di inizio, il sito peptidilico
viene occupato dalla molecola di tRNAfMet della formilmetionina (fMet) mentre i due siti A e E,
posti ai lati, rimangono liberi.
A questo punto il ribosoma inizia a scorrere lungo la molecola di mRNA percorrendola in
direzione 5’
3’ e in corrispondenza del sito A viene a trovarsi una nuova tripletta
(codone) che codifica per un diverso aminoacido (es. CCU = Prolina)
Il sito A viene occupato dalla molecola di tRNAPro contraddistinta dall’anticodone
complementare (GGA) e che porta legato alla sua estremità 3’ l’aminoacido Prolina.
Per l’intervento di un enzima specifico (peptidil transferasi), l’aminoacido fMet si lega alla
Prolina portata dal tRnaPro che si trova al sito A lasciando libera, allo stesso tempo, la propria
molecola di tRNA (tRNAfMet).
Il ribosoma continua a scorrere lungo la molecola di mRNA in direzione 5’
3’; come
conseguenza, la molecola fRNAMet che era nel sito P si sposta nel sito E, la molecola tRNAPro
che era nel sito A si sposta nel sito P trascinando le due molecole di aminoacido (fMet-Pro)
che porta legate all’estremità 3’ e il sito A viene a trovarsi in corrispondenza di una terza
tripletta (es. UCC = Serina)
Nel sito A si inserisce la molecola tRNASer che porta legato l’aminoacido Serina al quale,
sempre per azione dell’enzima peptidil transferasi, si legano i due aminoacidi portati dalla
molecola di tRNAPro posta al sito P.
La storia si ripete per tutte le triplette di cui è formata la sequenza che deve essere tradotta
e, per ogni tripletta, la catena polipeptidica si allunga di un aminoacido fino a quando il
ribosoma, continuando a scorrere lungo la molecola di mRNA, non viene a trovarsi con una
delle tre triplette di STOP (UAA; UAG; UGA) in corrispondenza del sito A.
A questo punto, poiché le triplette di STOP non codificano per alcun aminoacido, nel sita A si
inserisce una molecola (fattore di rilascio) provocando l’interruzione della sintesi, l’uscita
delle ultime molecole di tRNA utilizzate dai rispettivi siti (E e P) del ribosoma e la liberazione
del polipeptide.
Le due sub unità ribosomali si staccano e tornano libere nel citoplasma, pronte ad essere
impiegate in un nuovo processo di sintesi.
Il codice genetico
Una volta accertato che la sequenza delle basi sulla molecola del DNA determinava la
sequenza degli aminoacidi nei polipeptidi, è stato affrontato il problema di chiarire il
12
meccanismo molecolare mediante il quale la successione di quattro diversi nucleotidi
(A;U;C;G) sulla molecola di mRNA fosse in grado di controllare la successione di un ventina
di aminoacidi nella molecola polipeptidica.
La prima considerazione fatta fu che con quattro basi prese singolarmente possono essere
identificati, come massimo, quattro aminoacidi. Se le basi vengono considerate due a due in
tutte le possibili combinazioni, come massimo possono essere identificati 16 aminoacidi,
quattro in meno del numero di aminoacidi fondamentali.
Per identificare tutti i 20 aminoacidi fondamentali è perciò necessario coinvolgere le 64
triplette di basi che si possono ottenere considerando le quattro basi prese a tre a tre in tutte
le possibili permutazioni e immaginare che ogni aminoacido sia codificato da almeno una
tripletta di basi.
L’intera serie di 64 triplette fu identificata come “codice genetico” e alle singole triplette fu
dato il nome di “codoni”.
Furono quindi identificati i gruppi di tre basi (triplette o codoni) corrispondenti a ciascun
aminoacido.
Furono necessari più di dieci anni, dopo la pubblicazione di Watson e Crick sulla struttura del
DNA, perché venisse definitivamente confermato che, in realtà, la corrispondenza tra la
sequenza di basi nella molecola di mRNA e la sequenza degli aminoacidi nella molecola
polipeptidica è determinata dalla successione dei codoni nella molecola di mRNA.
Successivamente, si cercò di comprendere meglio il funzionamento del processo di
traduzione e, alla fine, furono identificate le proprietà fondamentali del codice genetico così
come elencate qui di seguito:
-
il C.G. è composto da triplette nucleotidiche. Tre nucleotidi di un mRNA
specificano un aminoacido in un prodotto polipeptidico; ogni codone è quindi
costituito da tre ribonucleotidi.
-
il C.G. non è sovrapposto. Ogni nucleotide dell’mRNA appartiene soltanto ad un
codone (fanno eccezione rari casi di geni sovrapposti).
-
il C.G. è privo di interpunziioni. Non ci sono forme di punteggiatura nelle regioni
codificanti: durante la traduzione i codoni sono letti consecutivamente.
-
il C.G. è degenerato. Tutti gli aminoacidi, eccetto due, sono specificati da più di un
codone.
-
il C.G. è ordinato. I diversi codoni che specificano per lo stesso aminoacido o per
aminoacidi con proprietà chimiche simili differiscono , disolito, per un singolo
nucleotide.
-
il C.G. contiene codoni di inizio (AUG) e di termine (UAA; UAG; UGA).
-
il C.G. è quasi universale. Con pochissime eccezioni (mitocondri dei mammiferi,
dei lieviti e di altri organismi), i codoni hanno lo stesso significato – specificano per
lo stesso aminoacido – in tutti gli organismi viventi, dai virus agli esseri umani.
13
Regolazione dell’espressione genica
La regolazione dell’espressione genica è il meccanismo attraverso il quale gli organismi
viventi sono in grado di adeguare il proprio metabolismo alle modificazioni ambientali (in
senso lato).
La regolazione dell’espressione genica consiste infatti in una modificazione dell'attività dei
geni di un organismo in modo che vengano sintetizzati prodotti genici adeguati a consentire
all’organismo stesso di crescere e riprodursi in una diversità di situazioni ambientali.
Non tutti i geni, tuttavia, si comportano allo stesso modo.
Si suole infatti distinguere fra:
-
geni regolati = la cui attività è soggetta a controllo come risposta a specifiche
esigenze della cellula e dell’organismo;
-
geni costitutivi = la cui attività porta alla sintesi di prodotti indispensabili alla vita
della cellula e dell’organismo, indipendentemente dalle condizioni ambientali.
In linea generale si può dire che la regolazione genica è la conseguenza di specifiche
interazioni tra proteine (proteine regolatrici) e particolari sequenze di DNA.
Nello studio della regolazione genica si fa distinzione tra procarioti ed eucarioti.
Nei procarioti sono presenti sistemi semplici che controllano la sintesi coordinata di proteine
con funzioni correlate (operoni).
Negli eucarioti si fa distinzione fra:
-
regolazione a breve termine = riguarda geni che vengono accesi o spenti come
risposta a cambiamenti ambientali o fisiologici repentini e costituisce un elemento
di adattamento immediato;
-
regolazione a lungo termine = riguarda eventi regolativi su geni che devono essere
accesi o spenti (attivati o disattivati) in relazione alla successione di eventi legati al
differenziamento di organi particolari e allo sviluppo e dell’organismo.
I livelli di controllo dell’espressione genica sono diversi e riguardano la fase:
trascrizionale
post – trascrizionale
traduzionale
post - traduzionale
14
Struttura del cromosoma eucariotico.
Il cromosoma eucariotico è formato da : DNA, RNA e Proteine che, nell’insieme,
costituiscono la cromatina.
La suscettibilità della cromatina a colorarsi, se sottoposta ad opportuni trattamenti, ha
consentito di osservare i cromosomi, di contarli, descriverli, misurarli e studiarne le anomalie.
Le proteine associate al DNA sono di due tipi:
basiche, denominate istoni o proteine istoniche, aventi all’incirca la medesima
composizione in tutti gli eucarioti e presenti nella cellula in quantità uguale al DNA;
acide o neutre (proteine non istoniche) che costituiscono l’intelaiatura del
cromosoma.
Sugli istoni, opportunamente arrangiati in ottameri di molecole uguali due a due, si avvolge la
molecola di DNA dando luogo alla formazione dei nucleosomi che rappresentano il punto di
partenza per la strutturazione del DNA nei cromosomi.
I nucleosomi, infatti, uniti tra loro da un breve tratto di DNA, formano, nell’insieme, la
cosiddetta “fibra di cromatina” ( 10 nm) che si confeziona in forme avvolte in maniera
sempre più compatta fino a costituire il cromosoma.
E’ con questo meccanismo che le lunghissime molecole di DNA (una sola molecola per
ciascun cromosoma) riescono ad essere contenute in strutture molto piccole quali sono i
cromosomi.
A titolo di esempio si può osservare che la molecola di DNA che costituisce il cromosoma
umano più grande (diametro di 0,5 m e lunghezza di 10 m) è lunga circa 85 mm e che
l’intero DNA di una cellula somatica umana (2n = 46) copre una lunghezza di circa 2 metri.
Numero e forma dei cromosomi
Procarioti = unico cromosoma circolare.
Eucarioti = più cromosomi lineari di dimensione diversa riuniti nel nucleodove sono presenti
in forma estremamente rilassata, tanto da non essere visibili.
In ogni cromosoma, quando venga osservato in una certa fase della divisione cellulare nella
quale i cromosomi si rendono particolarmente evidenti e possono essere più facilmente
osservati, presenta una costrizione (centromero) che, separandolo in due parti di lunghezza
variabile, ne determina la forma.
Il centromero è una zona nella quale la cromatina appare più addensata e rappresenta una
regione molto importante del cromosoma in quanto ad essa si legano i microtubuli (fibre) del
fuso durante le divisioni cellulari (mitosi o meiosi) promuovendone la migrazione verso i due
poli.
I cromosomi osservabili in una cellula somatica di un individuo ne costituiscono il cariotipo.
15
Il numero di cromosomi che costituiscono il cariotipo viene indicato con 2n e rappresenta il
numero diploide.
Il cariotipo di un individuo è costituito da due serie di cromosomi, uguali due a due per forma
e dimensioni, una di derivazione materna e l’altra di derivazione paterna.
Il cariotipo di un individuo è quindi costituito da tante coppie di cromosomi omologhi.
Il numero di coppie di cromosomi omologhi è indicato con n e rappresenta il numero aploide
che è proprio delle cellule sessuali (gameti). Il set di cromosomi che costituisce il numero
aploide viene anche indicato come genoma aploide.
Il numero di coppie di cromosomi omologhi è generalmente pari alla metà del numero di
cromosomi che costituiscono il cariotipo.
Considerando la cosa da un altro punto di vista, si può anche dire che il numero di
cromosomi che costituiscono il cariotipo è generalmente pari al doppio del numero aploide.
Questa affermazione non è sempre e comunque vera in quanto in alcune specie animali (es.
le cavallette) le femmine hanno un cromosoma in più rispetto ai maschi. Questo cromosoma
extra è stato chiamato cromosoma X. Le femmine di queste specie hanno due cromosomi X
mentre i maschi ne hanno uno solo.
l numero di cromosomi che costituiscono il cariotipo è molto diverso da specie a specie
La disposizione ordinata per dimensione e forma dei cromosomi di un cariotipo costituisce il
cariogramma.
In un cariotipo il riconoscimento dei diversi cromosomi viene fatto in base a:
forma, determinata dalla posizione del centromero. Si distinguono in
Metacentrici
Submetacentrici
Acrocentrici
Telocentrici
dimensioni
presenza di satelliti
bandeggio
Il genoma di una specie, vegetale o animale, può anche essere descritto in base alla quantità
di DNA contenuto in una cellula aploide. Tale quantità viene indicata come “valore C” che è
un valore relativo al quale vengono riferiti i contenuti di DNA di cellule della stessa specie a
livello diverso di ploidia. Così, una cellula somatica avrà un contenuto pari a 2C, una cellula
dell’endosperma (triploide = 3n) pari a 3C, e così via.
Il valore C varia notevolmente da specie a specie, ma non è necessariamente correlato con
la quantità o la complessità dell’informazione genetica che il genoma di una certa specie
contiene.
16
LA RIPRODUZIONE NEI VEGETALI
Nei vegetali si può avere:
-
propagazione vegetativa,
-
propagazione apomittica,
-
propagazione sessuale (riproduzione propriamente detta).
Propagazione vegetativa = è il processo attraverso il quale, per la presenza di strutture
particolari (tuberi, rizomi, stoloni) da un unico individuo possono originarsi più individui
geneticamente identici tra loro e all’individuo di partenza.
La propagazione vegetativa, quindi, non prevede produzione di seme.
Può essere realizzata artificialmente anche in specie non provviste di organi specifici
impiegando tecniche particolari (talea radicata, margotta, innesto, propagazione “in vitro”).
L’insieme degli individui che si ottengono mediante propagazione vegetativa costituisce un
clone.
Si definisce allora come clone un insieme di individui geneticamente identici tra loro e
all’individuo dal quale si sono originati per propagazione vegetativa.
Propagazione apomittica = è quel tipo di riproduzione al quale prendono parte le strutture
fiorali e gli organi sessuali ad esse correlati ma nel quale si ha la formazione di seme senza
che avvenga l’unione tra gameti e cioè, senza fecondazione. La si può anche definire una
clonazione via seme.
Propagazione sessuale = prevede la fusione di due gameti aploidi (cellule sessuali), uno
maschile ( ♂ ) e uno femminile ( ♀ ) secondo quel processo indicato come fecondazione.
Da questa fusione si origina un’unica cellula diploide che prende il nome di zigote.
Lo zigote, per successive divisioni cellulari (mitosi) forma l’embrione, presente nel seme,
dal quale, dopo la germinazione, con lo stesso meccanismo, prende forma l’organismo
pluricellulare (sporofito).
Lo sporofito, a sua volta, partendo da cellule 2n specializzate (macro e microsporociti)
presenti negli organi riproduttivi (ovario e antera), forma, per meiosi, macrospore (♀) n e
microspore (♂) n dalle quali, per mitosi, si originano, rispettivamente, ovuli maturi
(gametofito ♀) e granuli pollinici maturi (gametofito ♂ ).
Granuli pollinici maturi e ovuli maturi fanno parte di quella che viene definita “generazione
gametofitica” che è rappresentata da cellule aploidi (n) e costituisce la “fase aploide” del
ciclo vitale di una pianta.
17
L’unione di un gamete maschile con un gamete femminile (fecondazione) e la conseguente
formazione dello zigote comporta il ripristino del numero diploide (2n) proprio della
“generazione sporofitica” la quale rappresenta la “fase diploide” del ciclo vitale (ciclo
ontogenetico).
La propagazione o riproduzione sessuale implica perciò un’alternanza tra fase aploide (n)
(generazione sporofitica) e fase diploide (2n) (generazione sporofitica) e il suo principale
significato biologico sta nel fatto che essa produce ricombinazione genetica e cioè nuova
variabilità ad ogni generazione.
In un vegetale superiore che si riproduce per via sessuale la trasmissione dell’informazione
genetica contenuta nel materiale ereditario avviene quindi secondo due modalità:
da cellula somatica 2n a cellula somatica 2n, per mitosi, con la formazione di
cellule diploidi, che a loro volta, dividendosi, danno luogo ad altre cellule diploidi per un
numero indefinito di volte.
Ogni cellula che si genera contiene la stessa informazione genetica, per quantità e qualità,
presente nella cellula madre. Cellula madre e cellule figlie sono geneticamente identiche.
da cellula madre della linea germinale 2n a cellule n, per meiosi, con la
formazione di cellule aploidi (gameti) il cui destino è quello di unirsi per formare uno zigote
diploide.
Ogni cellula che si genera contiene una metà dell’informazione genetica presente nella
cellula madre.
Tale informazione, inoltre, è di solito diversa, o diversamente assemblata, rispetto a quella
contenuta nelle due cellule n (gameti) dalla cui unione è derivato l’individuo al quale
appartiene la cellula madre.
La differenza tra gli esiti delle due modalità di trasmissione del materiale ereditario è
determinata dalla successione di eventi che si verificano nel corso della mitosi e della meiosi.
CICLO CELLULARE
Per affrontare appropriatamente la trattazione della divisione cellulare è opportuno
conoscere, almeno per sommi capi il ciclo cellulare.
Quando si parla di “ciclo cellulare” si fa riferimento alle cellule somatiche e si intende di
solito indicare la serie di eventi che portano una cellula a dividersi per dar luogo a due cellule
figlie in quel processo di accrescimento di cui sono responsabili, in un vegetale, i tessuti
meristematici.
Nelle cellule somatiche il ciclo cellulare consta di due fasi fondamentali:
Mitosi – riguarda gli eventi connessi con la divisione cellulare vera e propria;
Interfase – riguarda gli eventi che accadono nell’intervallo di tempo che
intercorre tra due divisioni cellulari successive.
18
L’interfase, a sua volta, è caratterizzata da tre tappe o sotto-fasi:
G1 (Gap o intervallo 1) = è la sotto-fase di pre-sintesi durante la quale la cellula si
accresce, si duplicano gli organelli cellulari e la cellula si predispone alla duplicazione del
proprio DNA e alla sua strutturazione in cromosomi. In questa sotto-fase è individuabile il
cosiddetto “punto di restrizione” che corrisponde al momento oltre il quale la cellula deve
inevitabilmente dividersi.
S (Sintesi) = è la sotto-fase di duplicazione del DNA e delle molecole proteiche
necessarie alla usa strutturazione (istoni);
G2 (Gap o intervallo 2) = è la sotto-fase di post-sintesi nella quale la cellula si
predispone alla vera e propria divisione che avviene nella fase M (Mitosi).
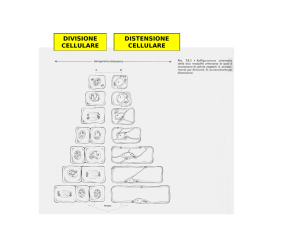
MITOSI: aspetti più significativi
1 – Nell’interfase ciascuna molecola di DNA si duplica per tutta la sua lunghezza, zone
centromeriche incluse, insieme con le proteine che ne costituiscono la struttura portante.
2 – Ogni cromosoma, all’inizio della mitosi (profase), si presenta pertanto sotto forma di due
filamenti separati (cromatidi fratelli) tenuti insieme dal centromero che è già duplicato, ma
non diviso.
3 – Nella metafase, ciascun cromosoma, indipendentemente da tutti gli altri, si dispone sulla
piastra equatoriale con i due cromatidi, uno da una parte e uno dall’altra della piastra,
fluttuanti verso i due poli.
4 – Nell’anafase, i due cromatidi di ciascun cromosoma si separano e vengono trascinati uno
verso un polo e l’altro verso il polo opposto, con la conseguenza che a ciascuno dei due poli
migrano 2n cromatidi che ora, in quanto divisi, sono da considerare veri e propri cromosomi.
5 – Nella telofase, i due gruppi di cromosomi vengono racchiusi nella membrana nucleare e
inizia a formarsi il fragmoplasto.
6 – Nella citocinesi si completa la formazione del fragmoplasto e le due cellule vengono
separate dalla membrana e, in un momento successivo, dalla lamella mediana.
La mitosi avviene con identiche modalità in cellule diploidi (2n) e in cellule aploidi (n)
Significato genetico della mitosi: garantisce il mantenimento dell’informazione genetica
inalterata dalla cellula madre alle due cellule figlie le quali, a loro volta, sono geneticamente
identiche tra loro.
19
Meiosi e Sporogenesi: generalità.
La meiosi è quel tipo particolare di divisione cellulare che caratterizza il processo di
sporogenesi e si verifica ogni qual volta un organismo a propagazione sessuale deve
produrre gameti.
Si tratta perciò di un tipo di divisione cellulare comune a tutti gli organismi che, nel loro ciclo
vitale, presentano una fase sessuale più o meno evidente.
Nei vegetali la meiosi avviene in tessuti preventivamente differenziati, a partire da cellule
particolari che, a seconda che siano situate negli organi riproduttivi femminili o maschili,
prendono il nome, rispettivamente, di “megasporociti” o “cellule madri delle megaspore”
(macrospore) e di “microsporociti” o “cellule madri delle microspore”.
Prima di descrivere ciò che accade in meiosi è comunque opportuno ricordare quanto
precedentemente detto a proposito del numero di cromosomi contenuti in cellule aploidi e
diplidi ( pag. 51)
MEIOSI: aspetti più significativi
1 - Analogamente a quanto visto per la mitosi, anche in questo caso è identificabile una fase
preparatoria durante la quale avvengono fenomeni analoghi a quelli già descritti parlando del
ciclo cellulare: in particolare, avviene la duplicazione del DNA (una sola duplicazione).
2 – La meiosi è costituita da due divisioni cellulari che avvengono in successione e che sono
indicate come prima e seconda divisione meiotica (anche: meiosi 1 e meiosi 2).
3 – Ciascun cromosoma, all’inizio della meiosi, si presenta duplicato nei due filamenti
(cromatidi fratelli) tenuti insieme dal centromero.
4 – Nel corso della profase 1, i cromosomi omologhi, ciascuno diviso nei due cromatidi, si
appaiano molto strettamente, formando delle tetradi (strutture nelle quali sono individuabili i
quattro cromatidi), e dispongono i loro centromeri, uno di origine materna ed uno di origine
paterna, da una parte o dall’altra della piastra equatoriale in maniera del tutto casuale e
completamente indipendente per le diverse coppie.
5 – Sulla piastra equatoriale si contano pertanto n tetradi, ciascuna formata da due
centromeri, uno di origine materna ed uno di origine paterna, ai quali sono uniti, due a due, i
quattro cromatidi.
6 – In questa fase, mentre i cromatidi dei due cromosomi omologhi di ciascuna coppia sono
appaiati per tutta la loro lunghezza, si verifica quel fenomeno, denominato “crossing over”
per mezzo del quale hanno luogo scambi di materiale genetico (di tratti di DNA) tra cromatidi
di cromosomi omologhi.
7 – In anafase 1, n centromeri migrano ad un polo e n al polo opposto formando due cellule,
ciascuna con n centromeri, assortiti casualmente tra materni e paterni, ognuno dei quali
porta, ancora riuniti, due cromatidi.
20
8 – Si conclude così la prima divisione meiotica che, proprio perché comporta la riduzione a
metà del numero di centromeri (da 2n a n), prende il nome di “divisione riduzionale”.
9 – A questo punto, le due cellule di nuova formazione vanno incontro alla seconda divisione
meiotica che, conservando inalterato il numero di centromeri (da n a n) prende il nome di
“divisione equazionale”.
10 – In ognuna delle due cellule, analogamente a quanto visto per la mitosi, gli n centromeri,
in ciascuno dei quali sono riuniti i due cromatidi, si dispongono sulla piastra equatoriale in
maniera indipendente.
11 – In anafase 2 i due cromatidi che sono riuniti in ciascun centromero si separano, migrano
verso l’uno o l’altro dei due poli e si formano due cellule, ciascuna con n cromosomi.
12 – Non necessariamente gli n cromosomi di queste cellule sono tutti di origine materna o
tutti di origine paterna.
Alla metafase/anafase della prima divisione si è verificato infatti l’assortimento casuale dei
centromeri di origine materna e paterna. Il numero di combinazioni di cromosomi materni e
paterni che si originano dall’assortimento casuale è pari a 2 n.
N.B. = In precedenza (pag. 51) si è identificato come valore C (contenuto di DNA di una
cellula) la quantità di DNA di un genoma aploide. E’ opportuno osservare che nel corso del
ciclo cellulare il valore C si modifica e può essere anche molto diverso a seconda della fase
del ciclo e del tipo di divisione cellulare (mitosi o meiosi) alla quale ci si riferisce. Nella tabella
riportata qui sotto si è cercato di analizzare, in maniera sintetica, questa relazione.
CICLO 2n 2n
MITOSI
P
M
INTERFASE
A
M
4C
T
2C
2n
G1
2C
MEIOSI 1
S
G2
2C4C
4C
2n
P
M
A
MEIOSI 2
T
4C
2C
2n
n
P
M
A
2C
T
C
n
CICLO 2n n
Relazione tra ciclo cellulare, tipo di divisione, fase della divisione, livello di ploidia e
quantita’ di DNA (valore C) presente nella cellula.
21
Significato genetico della meiosi
1 – Nella meiosi si ha un’unica duplicazione del DNA che avviene nella fase S (Sintesi)
premeiotica.
Questo comporta che, partendo da una cellula diploide (2n), si giunga, dopo le due divisioni
meiotiche, a quattro cellule aploidi (macro o microspore) dalle quali, per successive divisioni
mitotiche, prenderanno origine i gameti.
La fecondazione (unione tra gameti ♂ e ♀) ripristina, negli organismi a propagazione
sessuale, la situazione diploide.
In questo modo viene garantita la conservazione del numero cromosomico tipico della
specie.
2 – Il Crossing-Over che si verifica durante la meiosi 1 provoca un riassetto dell’informazione
genetica portata da ciascun cromosoma.
Tale riassetto si verifica attraverso lo scambio fisico di tratti più o meno lunghi di cromatidio
tra cromatidi di cromosomi omologhi.
E’ per mezzo di questi scambi che avviene il fenomeno noto come “ricombinazione”.
3 – Nella metafase 1 i centromeri materni e paterni hanno uguale probabilità di situarsi da
una parte o dall’altra della piastra metafasica.
Le quattro cellule prodotte da ogni meiosi avranno pertanto cromosomi materni e paterni
assortiti in modo casuale.
E’ il fenomeno noto come “assortimento casuale”.
Il Crossing Over (C.O.) e l’assortimento casuale dei centromeri sono pertanto alla base della
ricombinazione genetica che caratterizza gli organismi a propagazione sessuale e
costituiscono i mezzi fondamentali per il mantenimento della variabilità genetica nelle
popolazioni naturali animali e vegetali.
La conseguenza dei due eventi è infatti che le quattro cellule che si originano da ogni meiosi
portano tutte un’informazione genetica diversa tra loro e da quella contenuta nella cellula
madre dalla quale si sono originate.
Il completamento della seconda divisione meiotica conclude il processo di sporogenesi dal
quale si originano le megaspore, negli organi riproduttivi femminili, e le microspore in quelli
maschili.
Alla sporogenesi segue la gametogenesi
22
GAMETOGENESI e FECONDAZIONE: generalità
Con il termine gametogenesi si indica il processo mediante il quale, in un individuo
sessuato, si formano i gameti, cioè le cellule o i nuclei responsabili del processo di
fecondazione.
I gameti si originano a partire dalle megaspore e dalle microspore e, analogamente a quanto
accade nella formazione di queste, i gameti ♀ e ♂ prodotti da un individuo portano
un’informazione genetica sempre diversa tra loro e rispetto a quella contenuta nei gameti
dalla cui unione quello stesso individuo si è generato.
Si suole anche dire, perciò, che i gameti che entrano in una generazione portano
un’informazione genetica diversa, o sono geneticamente diversi, rispetto a quelli che ne
escono.
Gametogenesi femminile = serie di eventi che portano alla formazione del gametofito
femminile (ovulo maturo o embriosacco) entro il quale è situato il gamete femminile
(cellula uovo):
- una delle quattro megaspore – le altre tre degenerano – va incontro a tre
successive divisioni mitotiche;
- si forma una struttura (ovulo o sacco embrionale) all’interno della quale sono
individuabili 8 cellule aploidi, o semplicemente 8 nuclei aploidi, che hanno nomi
differenti e sono collocate in posizione diversa;
- una cellula uovo (il vero e proprio gamete femminile), affiancata dalle due
sinergidi, si colloca nelle immediate vicinanze del micropilo o foro micropilare,
posto alla base dell’ovulo, dal quale entrano i due nuclei spermatici portati dal
tubetto pollinico
- tre cellule antipodali situate dalla parte opposta dell’ovulo
- due cellule polari (nuclei polari), destinate a fondersi per formare una cellula 2n,
situata al centro, che rappresenta il “nucleo secondario dell’endosperma”
Gametogenesi maschile = serie di eventi che portano alla formazione del gametofito
maschile (granulo pollinico bi- o trinucleato) entro il quale, in seguito ad una prima mitosi,
si formano due nuclei: il nucleo vegetativo ed un secondo nucleo il quale, a sua volta, si
divide per mitosi dando luogo a due nuclei generativi o spermatici, uno dei quali è il
gamete maschile vero e proprio destinato ad unirsi con la cellula uovo.
Quando si verifica l’unione del gamete maschile con il gamete femminile si ha la
fecondazione o gamia.
Nei vegetali superiori si verifica anche una seconda fecondazione che è data dall’unione del
secondo nucleo generativo (n) portato dal gametofito maschile con il nucleo secondario
dell’endosperma (2n) presente nel gametofito femminile. Si parla perciò di “doppia
fecondazione”.
A seconda che il gamete femminile e il gamete maschile che si uniscono nella fecondazione
siano prodotti dallo stesso individuo oppure da individui diversi, si parla di autofecondazione
o autogamia e di allofecondazione o fecondazione incrociata o allogamia.
Il tipo di gamia è condizionato dal sistema riproduttivo di una specie e condiziona a sua volta
la struttura genetica delle sue popolazioni.
23
MEGAGAMETOGENESI O GAMETOGENESI FEMMINILE
NEGLI OVULI
Cellula madre delle macrospore (2n)
MEIOSI
sporogenesi
4 macrospore
gametogenesi
1 sopravvive
mitosi 1
2 nuclei n
mitosi 2
4 nuclei n
mitosi 3
8 nuclei n
3 degenerano
ovulo maturo o embriosacco = gametofito femminile
Nel gametofito femminile sono riconoscibili:
1 cellula uovo (gamete femminile) situata in prossimità del micropilo
2 cellule (nuclei) sinergidi che affiancano la cellula uovo
2 nuclei polari al centro dell’ovulo
3 nuclei antipodali situati in posizione opposta alla cellula uovo
Al momento della fecondazione:
la cellula uovo, unendosi al primo nucleo spermatico, forma lo zigote (2n) dal quale,
per successive divisioni mitotiche, si sviluppa l’embrione (sporofito);
i due nuclei polari, fondendosi, danno origine al nucleo secondario dell’endosperma
(2n) il quale, unendosi al secondo nucleo spermatico, forma un nucleo triploide (3n) dal
quale, per mitosi, si sviluppa l’endosperma (tessuto di riserva).
24
MICROGAMETOGENESI O GAMETOGENESI MASCHILE
NELLE ANTERE
Tessuto archesporiale
Cellule archesporiali
sporogenesi
Cellula madre delle microspore (2n)
MEIOSI
4 microspore (n)
(tetrade)
gametogenesi
separazione delle microspore
nutrizione delle microspore
ingrossamento
(cellule del tappeto)
mitosi 1
2 nuclei
1 nucleo vegetativo (n)
accrescimento
tubetto pollinico
1 nucleo generativo (n)
mitosi 2
2 nuclei generativi (spermatici) (n)
gamete maschile
(1°nucleo spermatico)
2° nucleo spermatico
FECONDAZIONE
Seconda fecondazione
(nucleo secondario)
ZIGOTE (2n)
Endosperma (3n)
N.B. = Il granulo pollinico maturo (con 2 o 3 nuclei) rappresenta il gametofito maschile.
25
RIASSORTIMENTO CASUALE DEI CENTROMERI
Probabilità che alla meiosi di un organismo con numero cromosomico diploide 2n i
centromeri di origine paterna e materna si assortiscano secondo determinati
rapporti.
½ = probabilità che un determinato centromero paterno (o materno) vada ad un polo
particolare;
½ è anche la probabilità che l’omologo materno (o paterno) vada al polo opposto;
½ x ½ = ¼ = (½)2 = 1/22 = probabilità che due particolari centromeri paterni (o materni)
vadano allo stesso polo;
(½)2 = 1/22 = è anche la probabilità che i due omologhi materni (o paterni) vadano al polo
opposto;
½ x ½ x ½ x ………x ½ = (1/2)n = 1/2n = probabilità che tutti gli n centromeri paterni (o
materni) vadano ad un certo polo;
n volte
(1/2)n = 1/2n = è anche la probabilità che gli n centromeri omologhi materni (o paterni)
vadano al polo opposto.
Se 2n = 8 la probabilità che ad uno stesso polo migrino tutti i 4 centromeri di origine
paterna e al polo opposto vadano i 4 di origine materna è pari a 1/24 = 1/16 = 0,0625.
Probabilità che su n centromeri, di origine materna (o paterna), k vadano ad un
determinato polo e z vadano al polo opposto ( k+z = n).
Può essere calcolata espandendo il binomio (a + b )2 dove a = b = ½
Se n = 1 (una sola coppia di cromosomi omologhi) la probabilità per ciascuno dei due
centromeri di andare ad un certo polo è pari a ½ :
( a + b )1 = a + b
( ½ + ½ )1 = ½ + ½
Se n = 2 (due coppie di cromosomi omologhi):
(a + b )2 = a2 + 2ab + b2
( ½ + ½ )2 = ( ½ )2 + 2 x ½ x ½ + ( ½ )2 = ¼ + ½ + ¼
Se n = 4 (quattro coppie di cromosomi omologhi):
( a + b )4 = a4 + 4a3b + 6a2b2 + 4ab3 + b4
( ½ + ½ )4 = ( ½ )4 + 4 x (½)3(½) + 6 x (½)2(½)2 + 4 x (½)(½)3 + (½)4 =
1/16 + 4 x 1/16 + 6 x 1/16 + 4 x 1/16 + 1/16 = 1,00
La parte frazionaria degli addendi è uguale; la somma dei vari addendi è uguale all’unità;
la probabilità è allora determinata dai coefficienti dei vari addendi.
26
I diversi addendi rappresentano la probabilità che, rispettivamente:
4 centromeri paterni e 0 materni;
3 centromeri paterni e 1 materno;
2 centromeri paterni e 2 materni;
1 centromero paterno e 3 materni;
0 centromeri paterni e 4 materni
vadano ad un determinato polo.
I coefficienti si possono ricavare dallo sviluppo del triangolo di Tartaglia:
n° coppie
di omologhi
(n° aploide)
possibili
assortimenti (*)
1
1
2
3
4
5
6
1
1
1
1
1
1
2
3
4
5
6
1
1
3
6
10
15
n
1
4
10
20
1
5
15
1
6
1
ecc.
2 = 21
4 = 22
8 = 23
16 = 24
32 = 25
64 = 26
2n
(*) = n° di possibili assortimenti in base al numero di centromeri materni e paterni e alla
loro qualità (Se sono tre di un genitore e uno dell’altro, possono essere 3 paterni e un
materno oppure 3 materni e un paterno; nel primo caso, i tre paterni possono essere
123;124;134; 234 e il materno, rispettivamente, 4; 3; 2; 1.Analogamente, nel secondo
caso, i tre materni possono essere: 123; 124; 134; 234 e il paterno, rispettivamente, 4; 3;
2; 1).
Per n coppie di cromosomi omologhi è uguale a 2n e dà anche il numero di possibili
gameti, sia maschili che femminili (cellule uovo e granuli pollinici).
Ciascun gamete maschile ha uguali probabilità di unirsi con ciascun gamete femminile.
In totale, quindi, in un individuo con 2n = 8 (n = 4) il numero di unioni possibili è uguale
a 16 per ciascun gamete e a 16 x 16 per tutti i gameti; in generale, è 2n x 2n = 22n
Delle possibili unioni, solamente una delle 16 relative a ciascun gamete avviene però tra
gameti uguali. Il numero di unioni possibili tra gameti diversi, nel caso dell’esempio, è
pertanto pari a 15 per ciascun gamete e, in totale, visto che i tipi di gameti sono 16, è pari
a 16 x 15 = 240.
In generale, per n coppie di omologhi, sarà: 2n x ( 2n – 1 ) = 22n – 2n
Nel nostro esempio: 28 – 24 = 256 – 16 = 240
27
Sulla base di quanto detto, se un individuo AaBbCcDd si è originato dalla unione dei due
gameti ABCD e abcd, solamente due, tra le 16 combinazioni possibili rintracciabili sia nei
gameti maschili che in quelli femminili prodotti da tale individuo, sono uguali a quelle
parentali, la altre 14 sono completamente nuove e si originano per assortimento casuale.
Vale la pena notare che le 14 nuove combinazioni sono di gran lunga le più numerose.
Vale anche la pena osservare che il numero di nuove combinazioni diventa tanto più
elevato quanto maggiore è il numero di coppie di cromosomi omologhi coinvolte e che, per
n coppie di cromosomi omologhi, è pari a 2n - 2.
Solamente come conseguenza dell’assortimento casuale dei centromeri materni e paterni
si genera perciò nuova variabilità genetica ad ogni generazione di riproduzione sessuata.
Vi è però un’altra fonte di diversità tra le 16 combinazioni dell’esempio, sia tra loro che
rispetto alle due combinazioni parentali.
Nel corso della profase della prima divisione meiotica, durante l’appaiamento fra
cromosomi omologhi (sinapsi), avviene quel fenomeno indicato come “crossing over”
che consiste nello scambio di porzioni di DNA tra cromatidi di cromosomi omologhi.
Come conseguenza, anche con riferimento alle 16 combinazioni viste in precedenza, i
cromosomi che si assortiscono non sono identici a quelli parentali, ma rappresentano,
rispetto a questi, dei riassetti più o meno sostanziali dell’informazione genetica a seconda
della frequenza dei crossing over e, quindi, dell’entità degli scambi avvenuti.
Questo spiega anche perché sia più corretto parlare di assortimento casuale dei
centromeri (all’interno dei quali non avviene crossing over) piuttosto che dei cromosomi
materni e paterni i quali, come tali, in seguito alla ricombinazione provocata dal crossing
over, non esistono più.
Iniziando a parlare di propagazione sessuata è stato detto che essa rappresenta un fonte
continua di variabilità genetica perché, ad ogni generazione, durante il processo di
sporogenesi che precede la formazione dei gameti, si verifica un riassetto
dell’informazione genetica portata dagli individui che si riproducono.
Tale riassetto viene normalmente indicato come “ricombinazione genetica”, anche se
con questo termine dovrebbe essere indicato, più appropriatamente, lo scambio di
materiale genetico che avviene in seguito al crossing over.
Tanto l’assortimento casuale dei centromeri quanto il crossing over contribuiscono
comunque, ad ogni generazione, alla formazione di nuova variabilità genetica sulla quale
può intervenire la selezione naturale, nelle popolazioni naturali, o la selezione artificiale,
nelle popolazioni segreganti appositamente costituite dall’uomo attraverso l’incrocio.
Da ultimo, vale la pena osservare che tanto l’assortimento casuale quanto la
ricombinazione propriamente detta (crossing over) provocano il riarrangiamento
dell’informazione genetica, ma non danno luogo a variabilità genetica completamente
nuova (nuove sequenze). La fonte primaria di variabilità genetica “ex novo” è infatti
rappresentata dalle mutazioni che, attraverso meccanismi diversi, si originano
costantemente in ogni specie.
28
GENI E CARATTERI
Gene = sequenza codificante per un polipeptide.
Carattere = manifestazione osservabile dell’azione svolta, da solo o in collaborazione
(interazione) con altre molecole, da quel polipeptide.
Genotipo = L’insieme dei geni portati da un individuo: la sua informazione genetica
Fenotipo = può indicare o la manifestazione osservabile dell’azione di un gene e, allora, il
suo significato coincide con quello di carattere, oppure l’insieme dei caratteri
presentati da un individuo come risultante dell’espressione del suo genotipo.
Ad ogni carattere possono essere associati uno o più geni.
Non tutti i geni trovano corrispondenza in un carattere.
Molti caratteri, la maggioranza, sono la conseguenza dell’interazione tra più polipeptidi, più
proteine e, quindi, tra più geni.
I caratteri per i quali vale la corrispondenza gene
proteina
carattere sono
indicati come “caratteri monofattoriali”.
Quanto più precise diventano le conoscenze sulla espressione dei geni, tanto più ci si
rende conto che questa corrispondenza è quanto mai labile e che ogni carattere deve
essere considerato come la risultante dell’azione contemporanea di più geni, tanto da
poter essere considerato un “carattere polifattoriale”.
Ogni gene può avere due o più alleli. Di questi, di solito, uno è il più diffuso in natura e
viene indicato come “allele selvatico” o “wild type” ed è simboleggiato con “+”. Gli altri
alleli vengono indicati come “mutanti”.
Alleli = forme alternative dello stesso gene che non ne alterano la funzionalità ma ne
modificano l’espressione.
Locus (plurale: loci) = è la posizione (il sito) occupata dal gene sul cromosoma.
Allo uno stesso locus dei due cromosomi omologhi di un organismo diploide, possono
essere presenti:
due alleli uguali = omozigote
due alleli diversi = eterozigote.
Tra i due diversi alleli presenti ad un locus vi può essere un rapporto di:
dominanza = un allele sopprime l’espressione (la manifestazione )dell’altro;
assenza di dominanza = la presenza dei due alleli dà luogo ad un fenotipo
intermedio;
codominanza = entrambi gli alleli si esprimono e vengono sintetizzati i prodotti
genici (polipeptidi) codificati da entrambi.
29
GENETICA MENDELIANA
Le leggi fondamentali dell’ereditarietà furono formulate per la prima volta da Gregorio
Mendel (1865) e furono scoperte studiando il comportamento di una serie di caratteri
monofattoriali (controllati da un solo gene) ciascuno dei quali era controllato da due alleli
diversi tra i quali esiste un rapporto di dominanza.
Mendel scelse come pianta il pisello (Pisum sativum L.), individuò sette coppie di caratteri
alternativi e, per ciascuno di questi, si garantì, come prima cosa, di avere a disposizione
una linea (ceppo, famiglia) stabile e cioè in grado di conservare il carattere da una
generazione all’altra in seguito ad autofecondazione; in totale, perciò, aveva a
disposizione 14 linee.
Le sette coppie di caratteri alternativi erano le seguenti:
Seme rotondo
Seme con cotiledoni gialli
Fiore purpureo
Baccello rigonfio
Baccello verde
Fiori ascellari (sv. indet.)
Fusto normale
-
Seme raggrinzito
Seme con cotiledoni verdi
Fiore bianco
Baccello raggrinzito
Baccello giallo
Fiori apicali (sv. det.)
fusto accorciato (nano)
Eseguì tutti gli incroci, inclusi i reciproci – 14 in totale – tra linee portatrici dei caratteri
alternativi.
Esaminò la discendenza F1 di ciascun incrocio.
Da ciascun individuo della generazione F1 ottenne, per autofecondazione, la generazione
F2.
P1
(seme giallo)
X
P2
(seme verde)
F1
seme giallo
F2
3 semi gialli : 1 seme verde
DOMINANZA
SEGREGAZIONE
Da notare:
L’azione di uno dei due alleli sopprime quella dell’altro
Dominanza
Il carattere scomparso in F1 riappare in F2
Segregazione
I due alleli presenti in F1 si separano (ségregano) nei gameti e si riuniscono casualmente
nel corso della fecondazione per dare la generazione F2.
30
Se una linea riproduce sé stessa in autogamia vuol dire che porta due fattori (alleli) uguali
o, in altre parole, che è omozigote per quell’allele.
I due fattori che controllano i due caratteri alternativi devono essere diversi.
Quando si incrociano due omozigoti, si generano individui F1 nei quali sono presenti
entrambi i fattori (alleli): si generano, cioè, individui eterozigoti.
In questo caso, uno dei due alleli (allele dominante) impedisce la manifestazione
(l’espressione) dell’altro (allele recessivo).
Principio della Dominanza
Quando un individuo F1 si riproduce, i due alleli si separano (sègregano) e si generano
due tipi di gameti, sia maschili che femminili, ciascuno dei quali porta un fattore diverso.
Principio della Segregazione
Durante la fecondazione questi due tipi di gameti maschili e femminili si uniscono in
maniera casuale e si generano genotipi
omozigoti per un allele
eterozigoti
omozigoti per l’allele alternativo
in rapporto
1
2
1
Poiché un allele è dominante sull’altro, il genotipo omozigote per il fattore dominante e
quello eterozigote hanno fenotipo uguale.
Nella generazione F2 i due fenotipi alternativi saranno perciò osservabili in un rapporto di
3 (fenotipo dominante) : 1 (fenotipo recessivo)
Oltre che di segregazione tra alleli di una coppia, si parla perciò anche di segregazione dei
caratteri
Oltre a linee omozigoti per alleli che controllavano singole coppie di caratteri alternativi,
Mendel si preoccupò di costituire e di mantenere linee omozigoti per due alleli dominanti o
per due alleli recessivi di due coppie alleliche diverse.
Analogamente a quanto fatto per le coppie di caratteri alternativi prese una alla volta,
eseguì gli incroci tra linee omozigoti per due alleli di coppie alleliche diverse che, pertanto,
presentavano, in maniera stabile, abbinamenti di caratteri alternativi ad altri abbinamenti
(es. seme rotondo e con cotiledoni gialli vs. seme raggrinzito e con cotiledoni verdi).
Eseguì le osservazioni sulla discendenza F1 di ciascun incrocio.
Da ciascun individuo della generazione F1 ottenne, per autofecondazione, la discendenza
F2
31
P1
(seme rotondo – cotiledoni gialli)
X
P2
(seme raggrinzito – cotiledoni verdi)
seme rotondo – cotiledoni gialli
F1
F2
9
s. rotondo
cot. gialli
:
3
s. rotondo
cot. verdi
3
s.grinzoso
cot. gialli
1
s. grinzoso
cot. verdi
Ciascuna delle due coppie di caratteri alternativi sègrega in rapporto 3 : 1. Infatti:
(9 + 3) = 12 cotiledoni gialli : (3 + 1) = 4 cotiledoni verdi
(9 + 3) = 12 seme rotondo : (3 + 1) = 4 seme grinzoso
Quando un individuo F1 si riproduce, gli alleli alternativi che controllano una coppia di
caratteri (cotiledoni gialli – cotiledoni verdi) si separano (sègregano) in maniera del tutto
indipendente dagli alleli alternativi che controllano la seconda coppia di caratteri (seme
rotondo – seme grinzoso).
Si generano così 4 tipi di gameti ciascuno dei quali porta un allele che controlla un
carattere di una coppia e un allele che controlla un carattere dell’altra coppia.
Poiché il modo con il quale, nei gameti, gli alleli alternativi di una coppia si appaiano agli
alleli alternativi dell’altra coppia è del tutto casuale, i quattro tipi di gameti si formano in
proporzioni uguali e cioè, secondo il rapporto 1 : 1 : 1 : 1 .
Di questi quattro tipi di gameti, due sono uguali ai gameti prodotti dai parentali della F1
(gameti parentali) e due son diversi (gameti ricombinanti)
Il rapporto tra gameti parentali e gameti ricombinanti, in questo caso, è 1 : 1
Principio della Segregazione indipendente
Nella fecondazione, questi quattro tipi di gameti si uniscono tra loro in modo del tutto
casuale e si generano 16 combinazioni genotipiche le quali, per i rapporti di dominanza
esistenti entro ciascuna coppia di alleli, danno luogo a 4 classi fenotipiche che sono tra
loro in rapporto di 9 : 3 : 3 : 1 tra fenotipi dominanti per entrambi i caratteri (doppi
dominanti), dominanti per un carattere e recessivi per il secondo, recessivi per il primo e
dominanti per il secondo, recessivi per entrambi i caratteri (doppi recessivi).
32
In linea generale si può quindi affermare che in un diibrido (individuo eterozigote per due
coppie alleliche) la segregazione indipendente prevede che i quattro tipi di gameti possibili
si formino con uguale frequenza.
Alla fecondazione, data la casualità con la quale è previsto che avvengano le unioni tra
gameti maschili e gameti femminili, i diversi genotipi che si originano si formeranno con
frequenza pari al prodotto delle frequenze gametiche.
Come conseguenza, se tra i due alleli di ciascuna coppia vi è un rapporto di dominanza,
nella generazione F2 si formeranno quattro classi fenotipiche in rapporto di 9 : 3 : 3 : 1.
Esiste una procedura più immediata per verificare se due coppie alleliche sono
indipendenti oppure no: è quella indicata come “reincrocio” o “incrocio di prova” oppure,
con terminologia più tecnica, come “test cross”. Tale procedura consiste nell’incrocio tra il
diibrido e l’omozigote doppio recessivo e, in caso di segregazione indipendente, porta alla
formazione di una discendenza composta da quattro classi genotipiche (AaBb, Aabb,
aaBb, aabb), alle quali corrispondono altrettante classi fenotipiche, tra loro in rapporto di
1 : 1 : 1 : 1 che riproduce esattamente il rapporto tra i quattro possibili tipi di gameti.
Poiché tra le 4 classi gametiche si fa distinzione tra gameti parentali (uguali a quelli formati
dai due parentali omozigoti del diibrido) e gameti ricombinanti (diversi da quelli formati dai
due parentali omozigoti), è opportuno sottolineare che, stante la casualità con la quale i
cromosomi omologhi si distribuiscono alla prima metafase meiotica, le classi gametiche
parentali e quelle ricombinanti si formano con uguale frequenza.
Vale anche la pena osservare che, date due coppie alleliche A/a e B/b, dove con A e B
vengono indicati gli alleli dominanti e con a e b quelli recessivi, gli individui omozigoti per
entrambe le coppie alleliche possono essere AABB e aabb ma anche AAbb e aaBB;
indipendentemente dalle due situazioni, l’ibrido tra i due genitori omozigoti sarà sempre
AaBb e i gameti che questo forma saranno sempre AB, Ab, aB, ab e, con segregazione
indipendente, questi saranno sempre tra loro in rapporto di 1 : 1 : 1 : 1.
Se le due coppie alleliche considerate sono indipendenti (situate su due coppie di
cromosomi omologhi), il verificarsi del Crossing Over (C.O.) a carico dei cromatidi di una o
di entrambe le coppie di omologhi non altera le frequenze delle quattro classi gametiche
prodotte da un individuo. Tuttavia, è da osservare che in assenza di C.O. si originano due
soli tipi di gameti da ogni meiosi, mentre in presenza di C.O. da ogni evento meiotico si
originano quattro tipi di gameti, due parentali e due ricombinanti (vedi figura).
INTERAZIONI GENICHE
Tutte le considerazioni fatte fino ad ora presuppongono un rapporto di dominanza tra i due
alleli di ogni coppia presa in esame:
cotiledoni gialli cotiledoni verdi
seme liscio seme grinzoso
fiore purpureo fiore bianco
polline allungato polline rotondo ecc.
33
Dopo la riscoperta delle leggi di Mendel prese così corpo l’idea che tra i due alleli di una
coppia vi è una differenza funzionale in base alla quale uno dei due non svolge nessuna
azione (recessivo) mentre l’altro fa tutto (dominante)
Mano a mano che le ricerche genetiche progredivano, ci si rese conto che questa è
un’estrema semplificazione.
Vi possono infatti essere situazioni nelle quali le cose non vanno secondo quanto previsto
da un normale rapporto di dominanza in base al quale un allele è definito come dominante
se dà luogo alla stessa manifestazione fenotipica sia che si trovi in condizione omozigote
oppure in condiozione eterozigote:
GG = giallo
Gg = giallo
gg = verde
LL = liscio
Ll = liscio
ll = grinzoso
Gli scostamenti da questa situazione possono essere diversi.
Dominanza parziale o incompleta = il fenotipo dell’eterozigote è diverso da quello dei
due omozigoti. Se il fenotipo dell’eterozigote è intermedio tra quello dei due omozigoti si
parla di Semidominanza o dominanza intermedia.
Esempi:
Colore del fiore nella bocca di leone
Rosso (w+w+) x Bianco (ww)
Rosa (w+w)
F1
Rosa (w+w)
F2
x Rosa (w+w)
1 Rosso (w+w+) : 2 Rosa (w+w) : 1 Bianco (ww)
Colore del piumaggio nel pollo andaluso
Bianco
F1
Nero
Nero azzurrato
Nero azzurrato
F2
x
1 Nero
x
Nero azzurrato
2 Nero azzurrato 1 Bianco
Segregazione fenotipica in F2: 1 : 2 : 1
Spiegazione: L’intensità della pigmentazione è determinata dalla quantità di
prodotto sintetizzato dal gene del colore che si traduce in quantità di pigmento. Se w+ lo
produce e w no, gli omozigoti w+w+ hanno una quantità di prodotto doppia rispetto a quella
degli eterozigoti w+w e quindi mostrano un colore più marcato.
34
Codominanza = il fenotipo dell’eterozigote mostra entrambi i fenotipi dei due omozigoti.
Esempio:
Reazione antigene – anticorpo nel controllo del gruppo sanguigno
dell’uomo (Sistema M / N).
Il tipo di segregazione fenotipica è uguale a quello della dominanza incompleta.
Allelia multipla = un gene possiede alleli differenti dei quali, nello stesso genotipo,
possono esserne presenti solamente due per volta allo stesso locus.
Il fenotipo dell’eterozigote può essere uguale o diverso rispetto a quelli degli
omozigoti a seconda degli alleli coinvolti.
Esempi:
Colore della pelliccia nel coniglio himalayano
(4 alleli coinvolti)
Reazione antigene – anticorpo nei gruppi sanguigni dell’uomo
(sistema A / B / 0): tre alleli coinvolti.
Il gene C, come il gene I, prendono il nome di “geni polimorfici”.
Gli alleli del gene C o del gene I formano una “serie allelica”
Negli alleli di una serie allelica è quasi sempre riconoscibile l’allele selvatico, di
solito il più diffuso, indicato con il simbolo +.
Gli altri alleli rappresentano delle mutazioni dell’allele selvatico e, di solito, sono
recessivi rispetto a questo.
La dominanza e le sue varianti sono da interpretare come “interazioni intra-locus” o
interalleliche.
Vi sono però manifestazioni fenotipiche che sono da vedere come la risultante di
interazioni tra geni diversi (interazioni inter-locus): si parla di “azioni geniche
complementari” e/o di “epistasia”.
Esempi:
Forma della cresta dei polli
Colore del fiore del pisello
Capacità cianogenetica del trifoglio bianco
Forma del frutto della zucca
Colore del frutto della zucca
Forma della siliqua della Capsella
Pleiotropia = un gene svolge più di una funzione.
Esempio:
Colore del fiore e dell’ipocotile della soia
35
LA RICOMBINAZIONE
Definizioni:
- E’ definita come generazione F1 la discendenza di un incrocio tra due individui
omozigoti.
- E’ definita come generazione F2 la discendenza ottenuta per autofecondazione da un
individuo F1 o per incrocio tra due individui della generazione F1.
- E’ definito come test-cross l’incrocio tra un individuo F1 e un individuo omozigote
recessivo; il test-cross può perciò essere visto anche come l’incrocio di un individuo F 1 con
il parentale recessivo e, per questo, viene anche indicato come “reincrocio”.
-°-°-°-°-°-°Aspetti significativi della ricombinazione:
1 Due geni posti su cromosomi diversi segregano in maniera indipendente:
F2 = 9 : 3 : 3 : 1
Test-cross = 1 : 1 : 1 : 1 (Reincrocio)
2 Si formano 4 classi fenotipiche, due delle quali con fenotipo uguale ai parentali e due
con fenotipo nuovo.
3 Le prime vengono identificate come “classi parentali”, le seconde come “classi
ricombinanti”.
4 La ricombinazione tra geni materni e paterni, e quindi tra i caratteri da essi controllati, è
dovuta, in questo caso, al riassortimento casuale dei centromeri alla metafase della
prima divisione meiotica.
5 Il numero di classi fenotipiche che si formano nella segregazione di una generazione F 2
oppure di un test-cross dipende dal numero di coppie alleliche coinvolte e, se n è il
numero di coppie alleliche, il numero di classi fenotipiche è pari a 2n
6 Due geni che in F2 , oppure che con il test-cross, danno rapporti di segregazione
significativamente diversi dal rapporto 9 : 3 : 3 : 1 oppure, rispettivamente, dal rapporto 1 :
1 : 1 : 1, sono situati sullo stesso cromosoma (fanno parte del medesimo gruppo di
associazione o di concatenazione) e vengono indicati come “geni associati” o “geni
concatenati”.
7 La ricombinazione (in senso proprio) è dovuta, in questo caso, al verificarsi del
Crossing over (C. O.) tra i due geni (loci) considerati.
8 Lo scostamento rispetto al rapporto di 9 : 3 : 3 : 1 (o di 1 : 1 : 1 : 1) si manifesta con due
classi fenotipiche più numerose e due meno numerose del dovuto; nel caso del test-cross,
rispettivamente > di ¼ e < di ¼.
36
9 Nel caso del test-cross, le quattro classi fenotipiche sono comunque ugualmente
numerose due a due:
le due più numerose sono le classi “parentali”;
le due meno numerose sono le classi “ricombinanti”.
10 Lo scostamento rispetto al rapporto di segregazione di 1 : 1 : 1 : 1, tipico del test-cross,
è dovuto al fatto che il C.O. non si verifica in tutte le meiosi; infatti, poiché da ogni meiosi
nella quale avviene il C.O. si ottengono due gameti parentali e due ricombinanti, se il C.O.
avvenisse in tutte le meiosi si dovrebbe avere il 50% di gameti parentali e il 50% di gameti
ricombinanti. Viceversa, poiché il C.O. non si verifica in tutte le meiosi, i gameti parentali si
formano con frequenza maggiore rispetto ai ricombinanti. In questo caso è pertanto
evidente che si formeranno con maggior frequenza combinazioni che coinvolgono i gameti
parentali rispetto a quelle nelle quali entrano i gameti ricombinanti.
11 La frequenza con la quale avviene il C.O. tra due loci ne determina la frequenza di
ricombinazione la quale è misurata dal rapporto tra la somma dei ricombinanti e il totale
degli individui esaminati e non può mai superare il 50%.
12 La frequenza di ricombinazione tra due loci concatenati è caratteristica di quei due loci
e dipende dalla loro distanza; altrettanto, la frequenza di ricombinazione tra più geni
associati presi due a due dipende dalla loro posizione reciproca.
13 E’ possibile misurare la distanza tra due geni associati utilizzando la frequenza di
ricombinazione (due loci ricombinano con frequenza tanto maggiore quanto più sono
lontani).
14 Come unità di misura della distanza tra due loci viene assunta quella distanza per la
quale la frequenza di ricombinazione tra i due loci è pari all’1%; a questa unità viene dato il
nome di centimorgan (cm).
15 Le distanze tra una serie di loci concatenati sono additive; è quindi possibile stabilire
una successione logica e una posizione relativa dei diversi loci sul cromosoma in base
alla loro frequenza di ricombinazione relativa (A/B = 20%; B/C = 5%; A/C = 15%; la
posizione relativa è A---------------C-----B e le distanze relative saranno 15 cm tra A e C,
5 cm tra C e B e 20 cm tra A e B).
16 Questa operazione prende il nome di mappatura e consente di costruire le mappe
genetiche nelle quali, per ciascun cromosoma, sono indicate le posizioni dei geni rispetto
al centromero e tra loro.
17 Lo strumento fondamentale per la costruzione delle mappe genetiche è il test-cross il
quale può essere applicato ad una serie di geni prendendoli in considerazione due per
volta: il test-cross viene anche indicato, in questo caso, come “test a due punti”.
18 Con il test a due punti si arriva, come massimo, a misurare una distanza di 50 cm
corrispondente ad una frequenza di ricombinazione del 50% (il C.O. avviene in tutte le
meiosi).
37
Test del chi quadrato (2)
Il test del 2 è un test statistico di dispersione o di scostamento che, applicato all’analisi
genetica, mette a confronto una serie di valori osservati con una serie corrispondente di
valori attesi.
Un test statistico è una procedura che consente di calcolare la probabilità in favore
dell’ipotesi che le differenze (fra valori osservati e valori attesi, fra una serie di valori, fra un
valore e una serie di altri) siano dovute unicamente al caso (ipotesi nulla); in base al
valore della probabilità così calcolata si decide se rifiutare o accettare l’ipotesi nulla.
Applicato all’analisi genetica, il test del 2 è da vedere come un test di significatività e
viene utilizzato per verificare se un certa segregazione fenotipica, osservata in un test
cross o in una generazione F2, sia in accordo con la segregazione attesa in base
all’assortimento casuale dei centromeri materni e paterni (segregazione indipendente=
ipotesi nulla) oppure se debba essere attribuita al fatto che i loci considerati sono
associati.
La necessità di ricorrere ad un test statistico per stabilire questo, deriva dal fatto che, per
solo effetto del caso, i valori osservati in una segregazione indipendente non coincidono
mai con quelli attesi e si scostano da questi in maniera più o meno evidente.
In presenza di una segregazione che si scosta da quella attesa, le domande alle quali
rispondere sono allora:
-
siamo autorizzati a ritenere tale scostamento come dovuto esclusivamente al
caso oppure lo dobbiamo attribuire al fatto che i geni coinvolti sono associati?
oppure: qual è il livello di probabilità rispetto al quale possiamo ritenere lo
scostamento dovuto solamente al caso?
oppure, ancora: quanto grande deve essere lo scostamento tra valori attesi e
valori osservati perché lo si possa ancora ritenere dovuto solamente al caso?
In altri termini, queste domande possono anche essere formulate nel modo seguente:
-
siamo autorizzati a ritenere valida, e quindi ad accettare, l’ipotesi nulla?
qual è il livello di probabilità rispetto al quale possiamo ritenere valida l’ipotesi
nulla?
quanto grande deve essere lo scostamento tra valori attesi e valori osservati
perché l’ipotesi nulla possa essere ritenuta ancora valida?
Quando i valori osservati coincidono con i valori attesi il valore del 2 è uguale a 0 (2 = 0).
A mano a mano che aumenta la differenza fra valori osservati e attesi, il valore di 2
aumenta.
Per decidere se i valori osservati sono in accordo con quelli attesi, cioè se l’ipotesi nulla
può essere accettata, è necessario confrontare il 2 calcolato con una serie di valori
tabulari relativi alla sua distribuzione teorica calcolata per un numero diverso di gradi di
libertà e per livelli diversi di probabilità.
38
Gradi di libertà (g.l.) = numero di confronti indipendenti che possono essere fatti tra due
serie di dati o di valori.
Es. = Nella segregazione di un test cross condotto su un diibrido si osservano i
valori: 375; 75; 85; 365; totale = 900. Se si trattasse di segregazione indipendente, i valori
attesi sarebbero: 225; 225; 225; 225; totale = 900.
Se ora si mettono a confronto le due serie di valori e si calcolano le differenze algebriche
tra le prime tre coppie, si ha:
375 225 = 150; 75 225 = 150; 85 225 = 140.
Poiché le somme delle due serie di valori sono uguali, la somma delle differenze tra i valori
di una serie e i valori dell’altra deve essere uguale a 0. E’allora evidente che la differenza
tra i valori dell’ultima coppia deve essere uguale a 140; infatti: 365 225 = 140. L’ultima
differenza è quindi predeterminata nel momento in cui si conoscano le altre tre; il numero
di confronti indipendenti, in questo caso, è pari a tre e tre è anche il numero di gradi di
libertà.
I valori tabulari (teorici) di 2 sono quelli che, per puro effetto del caso, si verificano, per un
determinato numero di gradi di libertà, con una probabilità del 5% o dell’1%. Questi due
livelli di probabilità sono quelli ai quali si fa di solito riferimento nell’analisi genetica.
Il valore tabulare 2 = 11,341 relativo a 3 g.l. per un livello di probabilità dell’1% sta perciò
ad indicare che valori di 2 11,341 sono osservabili per puro effetto del caso solamente
in 1 caso su 100, mentre in 99 casi su 100 sono osservabili in virtù di una reale mancanza
di aderenza tra valori osservati e valori attesi. Un valore di 2 11,341 è cioè troppo raro
perché si verifichi per puro caso; l’ipotesi in base alla quale i valori osservati sono conformi
a quelli attesi (ipotesi nulla) è pertanto da scartare.
Viceversa, se 2 < 11,341, vi è più dell’1% di probabilità che lo scostamento tra valori
attesi e valori osservati sia dovuto al caso.
E’ forse opportuno osservare che i livelli di probabilità indicati nella tabella qui sotto
riportata sono da interpretare come la probabilità che si ha di sbagliare scartando l’ipotesi
nulla con valori di 2 uguali o superiori a quelli tabulari.
Valori teorici di 2
Gradi di
libertà
(g.l.)
1
2
3
4
5
6
Probabilità
Gradi di
libertà
(g.l.)
5%
1%
3,841
5,991
7,815
9,488
11,070
12,592
6,635
9,210
11,341
13,277
15,086
16,812
7
8
9
10
20
30
Probabilità
5%
1%
14,067
15,507
16,919
18,307
31,410
43,770
18,475
20,090
21,666
23,209
37,570
50,890
(xoss. xatt.)2
2 =
xatt.
39
MUTAZIONI
Viene definito come mutazione qualunque cambiamento stabile, e perciò ereditabile, del
materiale genetico e dell’informazione genetica che esso porta, non causato da
ricombinazione.
Un individuo, mono o pluricellulare, che porti una mutazione è un mutante.
Ogni mutazione stabile di un gene che ne modifichi la manifestazione fenotipica ma che
non ne alteri la funzionalità rappresenta un allele di quel gene.
In linea generale, le mutazioni di un gene rappresentano alleli recessivi del gene selvatico.
Una mutazione recessiva si verifica quando un gene perde la sua funzione originaria o,
meglio, quando il polipeptide che esso produce non è più in grado di svolgere la propria
funzione originaria. La mutazione recessiva è pertanto un tipico allele “perdita di funzione”.
Al contrario, una mutazione dominante si verifica quando un gene acquisisce una nuova
funzione o, meglio, quando il polipeptide che esso produce è in grado di svolgere una
funzione diversa da quella originaria. La mutazione dominante è pertanto un tipico allele
“acquisto di funzione”.
I mutanti sono quindi estremamente utili nello studio della funzione dei geni.
Se la mutazione si ripercuote direttamente sul carattere controllato da un certo gene e non
altera la vitalità dell’individuo che la porta, se ne può studiare l’ereditarietà secondo i
normali rapporti di segregazione fenotipica previsti dalla genetica mendeliana.
Vi sono però mutazioni che alterano più o meno profondamente il metabolismo di un
organismo compromettendone più o meno radicalmente la capacità di riproduzione o di
sopravvivenza. Di queste mutazioni è evidentemente difficile, se non impossibile, seguire
l’ereditarietà sulla base dei normali rapporti di segregazione in quanto gli omozigoti e, a
volte, gli eterozigoti per la mutazione si sottraggono all’osservazione.
Sulla base di queste considerazioni si può allora fare una distinzione tra:
Mutazioni visibili = alterano un aspetto del carattere che controllano
Forma del seme
(carattere)
liscio - grinzoso
(aspetto)
Colore dei cotiledoni
(carattere)
giallo - verde
(aspetto)
Colore dell’occhio
(carattere)
colori diversi
(aspetto)
Mutazioni sterili = esercitano un’azione negativa sulle capacità riproduttive degli individui
mutanti i quali presentano fertilità ridotta o nulla.
40
Mutazioni letali = alterano drasticamente una funzione vitale e determinano la morte degli
individui omozigoti per la mutazione.
Una misura quantitativa delle mutazioni può essere fornita da:
tasso di mutazione = è la probabilità che un determinato evento di mutazione
avvenga in un determinato tempo; di solito si fa riferimento alle generazioni, per cui si
parla di numero di mutazioni per gene (o per coppia di nucleotidi, o per sequenza) per
generazione.
frequenza di mutazione = è il numero di eventi di un particolare tipo di mutazione
espresso come quota di individui o di cellule mutanti nella popolazione di riferimento; si
parla perciò di 1 x 105 1 x 107 individui o cellule.
Rispetto alla localizzazione si fa la distinzione tra:
Mutazione somatica = interessa esclusivamente cellule somatiche; dà luogo ad aree
(tessuti, organi) mutate, le cosiddette “chimere”.
Mutazione della linea germinale = interessa le cellule dalle quali si origineranno i gameti
i quali ne saranno portatori e la trasmetteranno sia alle cellule somatiche sia a quelle
germinali degli individui che da essi si originano.
Rispetto all’origine, si fa distinzione tra:
Mutazioni spontanee = insorgono naturalmente, generalmente come conseguenza di
modificazioni delle normali situazioni fisiologiche della cellula che possono provocare
alterazioni nei meccanismi di duplicazione o di riparazione del DNA. Mutazioni spontanee
possono anche insorgere per azione di sequenze particolari (elementi trasponibili o
trasposoni).
Gli elementi trasponibili sono tratti di DNA che possono muoversi autonomamente da un
sito ad un altro all’interno del genoma integrandosi con questo e provocando modificazioni
nell’espressione e nell’attività dei geni localizzati nel sito di inserzione o nei siti adiacenti.
Secondo il tipo, un traspostone che si inserisce in una nuova posizione può lasciare una
copia di se tesso nel sito di origine oppure può escindersi direttamente. Nel sito di
inserzione, gli elementi integrati sono fiancheggiati da sequenze duplicate del DNA del sito
bersaglio.
Poiché gli elementi trasponibili provocano riarrangiamenti cromosomici più o meno estesi,
è evidente che essi possono interferire sia con l’espressione del gene nel quale si
inseriscono, sia con l’attività dei geni adiacenti al sito di inserzione. Si ritiene che gli
elementi trasponibili abbiano dato un notevole contribuito all’evoluzione del genoma, in
generale e di quello vegetale in maniera particolare.
Mutazioni indotte = derivano dal trattamento di cellule o tessuti con agenti mutageni
chimici o fisici che danneggiano in misura più o meno rilevante il DNA: la mutazione
insorge per la mancata riparazione di tale danno. La conseguenza di un trattamento
mutageno è un incremento significativo del tasso naturale di mutazione di una certa specie
o di un certo gene.
I mutageni chimici possono agire con meccanismi diversi: provocando una sostituzione di
basi, modificando chimicamente le basi, intercalandosi nella sequenza delle basi.
41
I mutageni fisici sono generalmente rappresentati da radiazioni ad alta energia
caratterizzate da potere penetrante più o meno elevato e in grado, quindi, di provocare
alterazioni più o meno pesanti nella struttura del genoma. Da questo punto di vista, viene
fatta una netta distinzione fra radiazioni non ionizzanti (radiazioni ultraviolette) e radiazioni
ionizzanti (raggi X, radiazioni β o radiazioni γ)
Rispetto all’entità della modificazione, si è soliti distinguere tra:
Mutazione genica = qualunque alterazione della sequenza codificante, inclusa la
sostituzione, l’inserzione o la delezione di una o più coppie di basi all’interno di una
sequenza codificante. Se l’alterazione riguarda un’unica coppia di basi, si parla di
mutazione puntiforme
Mutazione o aberrazione cromosomica = interessa un cambiamento nell’organizzazione
di uno o più cromosomi.
Mutazione genomica = riguarda un cambiamento del numero di cromosomi che
costituiscono il genoma di una specie.
Mutazioni puntiformi o geniche
Possono essere dovute a sostituzioni, inserzioni o delezioni di coppie di basi.
Nelle sostituzioni si fa distinzione tra:
mutazioni per transizione, quando una coppia purina-pirimidina è sostituita da
un’altra coppia purina-pirimidina ( AT
GC; GC
AT; TA
CG; CG
TA).
mutazioni per transversione, quando una coppia purina-pirimidina è sostituita da
una coppia pirimidina-purina (AT TA e viceversa, GC
CG e viceversa, AT
CG e
viceversa GC
TA e viceversa.
Le mutazioni per sostituzione possono avere effetto fenotipico diverso a seconda
dell’entità dell’errore di lettura che esse provocano.
mutazioni missenso = il cambiamento di una coppia di basi nel DNA provoca un
cambiamento nel codone dell’RNA con il risultato che nel polipeptide viene inserito un
aminoacido diverso da quello originario (selvatico).
La mutazione può provocare allora la sintesi di un polipeptide alterato e, quindi, di una
proteina funzionalmente diversa e meno efficiente.
E’ ciò che accade in seguito alla sostituzione di una coppia di nucleotidi nella sequenza
del gene per la -globina; tale fatto provoca la sostituzione di un acido glutammico con la
valina in posizione 6 della molecola della proteina. L’individuo omozigote per questa
mutazione ha le molecole di -emoglobina alterate ed è affetto da anemia falciforme
(globuli rossi di forma alterata anziché normale).
La mutazione missenso può anche non avere alcun effetto se la sostituzione di un
aminoacido con un altro lascia inalterata la funzionalità della proteina; si parla in questo
caso di mutazione neutra
42
La mutazione può anche non avere effetto fenotipico se:
a – la sostituzione di un aminoacido con un altro non comporta una profonda
alterazione funzionale nella proteina (può essere assimilata ad una mutazione neutra).
b – la sostituzione di una coppia di basi con un’altra provoca la formazione di un
codone che codifica per lo stesso aminoacido (degenerazione del codice genetico). SI
parla, in questo caso, di mutazione silente.
Mutazione nonsenso = si verifica quando la modificazione di una coppia di basi porta alla
formazione di un codone che non codifica per alcun aminoacido e cioè per un codone di
stop. In questo caso, com’è evidente, la sintesi del polipeptide si interrompe e si ha la
formazione di una proteina non funzionale.
Mutazioni frameshift = si verificano quando, per l’inserzione o la delezione di una coppia di
basi, si verifica uno scivolamento della fase di lettura che comporta inevitabilmente la
lettura di codoni diversi da quelli corretti. Se la mutazione interessa una sequenza
codificante per un polipeptide, questo significa che si possono generare polipeptidi più
corti o più lunghi del dovuto e, comunque, non funzionanti.
Esempio:
THE FAT CAT ATE THE BIG RAT
THE FTC ATA TET HEB IGR AT
A deleto
THE FAT CAT ATT ETH EBI GRA T
T inserito
La frase, dal punto di delezione o da quello di inserzione in poi, non ha più alcun
senso.
Gli effetti di una mutazione genica possono regredire (revertire o essere annullati) sia per
ripristino della sequenza modificata al suo stato originale (reversione vera), sia come
conseguenza di un nuovo evento di mutazione in un sito diverso da quello nel quale è
avvenuta la prima mutazione (mutazione soppressore).
Mutazioni cromosomiche
Sono cambiamenti della struttura dei cromosomi rispetto alla condizione normale.
La delezione comporta la perdita di un tratto di cromosoma e può essere provocata da
calore o da radiazioni, in particolare da quelle ionizzanti. Le sue conseguenze dipendono
dal tipo di geni o dalle parti di geni che vengono perduti. Un individuo eterozigote per una
delezione può essere normale se il cromosoma non alterato porta un allele normale, ma
se porta un allele recessivo con effetto negativo l’effetto può essere letale. Se la delezione
interessa la zona del centromero, si forma un cromosoma acentrico che verrà perso nel
corso della meiosi.
La duplicazione implica il raddoppiamento di un tratto di cromosoma (in posizione diversa
o a tandem) con conseguente alterazione dell’appaiamento tra cromosomi omologhi nel
corso della meiosi. In linea di massima si ritiene che le duplicazioni siano sopportate
meglio delle delezioni e sembra accertato che abbiano avuto un ruolo importante
43
nell’evoluzione dei geni multipli che svolgono funzioni correlate (famiglie geniche). Un
esempio può essere quello dei geni dell’emoglobina nell’uomo.
L’inversione può essere pericentrica (include il centromero) o paracentrica (non
comprende il centromero). In linea generale l’inversione non comporta la perdita di
materiale genetico, anche se può comportare delle conseguenze se la rottura del
cromosoma avviene all’interno di un gene. L’inversione può essere omozigote oppure
eterozigote.
Nel primo caso non vi sono conseguenze a livello meiotico, in quanto l’appaiamento tra
omologhi avviene normalmente.
Nel secondo caso, invece, la presenza dell’inversione provoca appaiamento irregolare,
con formazione di cromosomi acentrici e dicentrici che vanno persi, e assenza di
ricombinazione nel segmento invertito, tanto che l’inversione viene anche identificata
come un soppressore della ricombinazione.
La traslocazione comporta il cambiamento di posizione di tratti di cromosoma, senza
aumento o perdita di materiale genetico, analogamente all’inversione. Si possono
verificare due tipi di traslocazione:
-
traslocazione intracromosomica = implica lo spostamento di un tratto
cromosomico da un punto all’altro dello stesso cromosoma;
-
traslocazione intercromosomica = comporta lo spostamento di un tratto
cromosomico da un cromosoma ad un altro; in questo caso si fa distinzione fra:
-
traslocazione reciproca, quando due cromosomi si
scambiano porzioni di cromosoma;
-
traslocazione non reciproca, quando lo spostamento
avviene in una sola direzione.
Le conseguenze delle mutazioni o aberrazioni cromosomiche si riflettono, in linea
generale, sulla gametogenesi e, quindi, sulla fertilità degli individui che ne sono affetti. Tali
conseguenze, tuttavia, in particolare per quelle mutazioni che non comportano la perdita di
informazione genetica (duplicazioni, inversioni , traslocazioni) sono sostanzialmente
diverse a seconda che la mutazione sia in condizione omozigote oppure eterozigote.
Nel primo caso, a meno di interruzioni di sequenze nei siti di rottura del cromosoma,
l’informazione genetica non viene né persa né alterata e la mutazione può anche non
avere conseguenze dannose.
Nel secondo caso, invece, la mutazione comporta sempre difficoltà di appaiamento tra
cromosomi omologhi durante la meiosi e, quindi, un’alterazione più o meno marcata nella
produzione dei gameti (gameti non funzionanti, gameti sbilanciati), che può significare o
una riduzione netta della fertilità dell’individuo mutato oppure la manifestazione di
alterazioni fenotipiche più o meno importanti nella sua discendenza.
44
Mutazioni genomiche
Un organismo o una cellula sono definiti euploidi quando presentano un assetto
cromosomico completo o un multiplo esatto di assetti cromosomici completi. Questa
condizione è definita come euploidia.
Un organismo o una cellula sono definiti aneuploidi quando presentano un assetto
cromosomico incompleto, o ridondante, per la mancanza o la presenza in eccesso di uno
o più cromosomi singoli.La condizione è definita come aneuploidia.
Variazioni nel numero di cromosomi che portano ad una situazione di aneuploidia possono
verificarsi per mancata o irregolare disgiunzione di una o più coppie di cromosomi
omologhi in prima o seconda divisione meiotica nel corso della gametogenesi.
Le situazioni aneuploidi che si possono verificare sono diverse e qui di seguito ne sono
indicate alcune.
Diploidia (2n) = situazione normale
1
2
3
4
Monosomia (2n – 1) = perdita di un singolo cromosoma da una
qualunque coppia di cromosomi omologhi
Nullisomia (2n – 2) = perdita di una coppia di cromosomi
omologhi
1
2
3
4
Trisomia (2n + 1) = acquisizione di un singolo cromosoma. Vi
sono 3 copie dello stesso cromosoma
1
2
3
4
Doppia Monosomia (2n + 1 + 1) = perdita di un cromosoma da
ciascuna didue coppie di cromosomi omologhi.
1
2
3
4
Tetrasomia (2n + 2) = acquisizione di due cromosomi di una
stessa coppia di omologhi. Vi sono 4 copie dello stesso
cromosoma
1
2
3
4
ecc.
45
Tutte le forme di aneuploidia sono letali o sub-letali per gli animali, mentre sono molto
meglio sopportate dalle piante nelle quali sono state utilizzate, in passato, come un
importante mezzo di ricerca per studiare la funzione dei geni situati su singoli cromosomi.
Forme aneuploidi sono anche osservabili, più frequentemente che in quelle animali, nelle
popolazioni naturali di diverse specie vegetali.
Un organismo o una cellula vengono definiti euploidi quando presentano uno o più assetti
cromosomici in eccesso rispetto alla situazione diploide normale. Si fanno ricadere tra gli
euploidi anche le situazioni monoploidi nelle quali è presente un unico assetto
cromosomico aploide (n).
A titolo di esempio sono indicate qui di seguito alcune tra le situazioni euploidi più comuni.
Assetto normale (diploide = 2n)
1
2
3
4
Monoploidia (n) coincide con l’aploidia (*)
1
2
3
4
Triploidia (3n)
1
2
3
4
Tetraploidia (4n)
1
2
3
4
ecc.
(*) = Il termine “aploide” è di solito usato per descrivere la situazione dei gameti, nei quali
è presente un numero di cromosomi pari alla metà di quello proprio delle cellule
somatiche.
La situazione monoploide è rara e, tra gli animali, è presente in api, vespe, formiche, in
individui provenienti da uova non fecondate.
Nelle piante, individui monoploidi possono essere ottenuti artificialmente mediante la
coltura di antere o di microspore oppure di ovuli non fecondati. Gli individui monoploidi
46
(aploidi) possono essere vantaggiosamente utilizzati per ottenere i cosiddetti aploidi
diploidizzati, o diplo-aploidi, mediante trattamento con colchicina dell’apice vegetativo,
caratterizzato dalla presenza di cellule meristematiche in attiva divisione, e conseguente
raddoppiamento del numero cromosomico (n
2n). Il trattamento con colchicina,
alcaloide inizialmente identificato nella specie Colchicum autunnale, ha infatti l’effetto di
bloccare la formazione del fuso mitotico nel corso della divisione cellulare. In questo modo
si possono ottenere individui completamente omozigoti anche partendo da piante
eterozigoti.
La situazione polipoide è invece molto comune nelle specie vegetali dove ha
rappresentato uno strumento di notevolissima importanza dal punto di vista evolutivo.
Circa la metà dei generi di piante conosciute contiene specie polipoidi e sono polipoidi i
due terzi delle graminacee. La moltiplicazione del genoma è perciò da ritenere un evento
mutazionale piuttosto comune tra le angiosperme ed ha contribuito in modo marcato alla
speciazione, soprattutto tra le specie arboree.
Una conseguenza abbastanza generalizzata della poliploidia è l’aumento delle dimensioni
cellulari che, in generale, si traduce in un aumento di dimensioni dell’organismo. Le specie
polipoidi tendono ad essere più grandi delle loro omologhe diploidi e questo implica, di
solito, la tendenza a produrre semi, frutti, radici, foglie di maggiori dimensioni e, quindi, a
fornire una più consistente produzione utile.
E’ presumibilmente per questo motivo che esempi di poliploidia sono molto frequenti tra le
specie coltivate le quali, per circa due terzi, sono da ritenersi polipoidi (2n > 18). Tra le
piante coltivate è inoltre abbastanza agevole rintracciare quelle che sono individuate come
“serie polipoidi” che comprendono specie con numeri di cromosomi che, per ciascuna
serie, sono multipli di uno stesso numero cromosomico di base. In qualche caso si tratta di
specie a diverso grado di evoluzione o a diverso tipo di utilizzazione e che raccordano
molto chiaramente le specie coltivate attuali con quelle di più antica domesticazione o con
le specie ancestrali.
A seconda dell’origine degli assetti cromosomici, si fa distinzione tra:
autopoliploidia = quando tutti gli assetti cromosomici derivano dalla medesima
specie;
allopoliploidia = quando gli assetti cromosomici derivano da specie diverse.
Nei poliploidi non sono rari fenomeni più o meno accentuati di sterilità in quanto, alla
meiosi, possono formarsi gameti scompensati (n ± 1) che, unendosi o tra loro o con
gameti normali, possono formare zigoti scarsamente vitali, o completamente non in grado
di sopravvivere, oppure sterili.
La sterilità è particolarmente frequente negli autopoliploidi nei quali, la presenza di serie di
cromosomi uguali comporta difficoltà di appaiamento degli omologhi alla prima divisione
meiotica che possono causare anche perdita di tratti di uno o più cromosomi con perdita di
informazione genetica e la conseguente formazione di gameti non vitali.
In molte specie (es. graminacee) questi fenomeni sono superati o con la propagazione
vegetativa oppure con meccanismi che comportano la formazione di semi senza che vi
sia unione tra gameti (apomissia).
47
GAMETOGENESI NORMALE
GAMETOGENESI ALTERATA
gameti ridotti (n = 5)
gameti non ridotti (2n = 10)
AUTOPOLIPLOIDIA OMOZIGOTE
genitore diploide
gameti non ridotti
2n = 10
autotetraploide omozigote
2n = 10
4n = 20
AUTOPOLIPLOIDIA ETEROZIGOTE
genitore 1 (2n= 10)q
gameti non ridotti (2n = 10)
genitore 2 (2n=10)
gameti non ridotti (2n = 10)
autopoliploide eterozigote (4n = 20)
48
Tra gli autopoliploidi, inoltre, è opportuno distinguere fra omozigoti e eterozigoti (vedi
figura).
In questo caso, il termine “omozigote” sta ad indicare che nella costituzione genetica
dell’autopoliploide non vi è unione tra gameti di individui diversi. Il termine “eterozigote”
indica invece che la situazione polipoide si è generata con il contributo di individui diversi.
I due termini non fanno quindi riferimento al tipo di alleli presenti nel polipoide.
autopoliploidi omozigoti = si originano per unione di gameti non ridotti prodotti
dallo stesso individuo oppure in seguito a duplicazione del numero di cromosomi in
cellule somatiche (meristematiche) di un individuo originatosi per autofecondazione, come
conseguenza di irregolarità nella mitosi analoghe a quelle indotte dal trattamento con
colchicina.
autopoliploidi eterozigoti = si originano dall’unione di gameti non ridotti prodotti da
individui diversi della stessa specie, oppure in seguito a duplicazione del numero di
cromosomi in cellule somatiche (meristematiche) di un individuo originatosi per incrocio.
Livelli di poliploidia particolari (3n, 5n, 6n e superiori si sono probabilmente originati per
unione di gameti non ridotti (2n) con gameti normali (n) o per duplicazione di livelli di
ploidia intermedi: 2n + n = 3n; 3n + 2n = 5n; 3n x 2 = 6n, ecc.
Una annotazione a parte meritano gli autopoliploidi in quanto presentano un particolare
modello di ereditarietà che è indicato come “eredità tetrasomica” per distinguerlo da quello
osservabile in una specie diploide e che è indicato come “eredità disomica”.
Vale anche la pena ricordare che nei vegetali esistono e sono stati identificati geni che
controllano l’appaiamento cromosomico alla meiosi e dalla cui espressione può quindi
dipendere la possibilità della regolare formazione dei gameti.
I poliploidi di nuova formazione, sia omozigoti che eterozigoti, presentano generalmente
un’accentuata variabilità per la maggior parte delle loro caratteristiche morfologiche e
fisiologiche e, proprio per questo, sono generalmente caratterizzati da una maggiore
capacità adattativa rispetto ai corrispondenti diploidi da cui derivano. E’ ovvio che tale
variabilità sarà tanto più elevata quanto maggiore è la diversità genetica dei genomi che si
uniscono.
Come si è detto, gli allopoliploidi si originano per unione di gameti prodotti da individui di
specie diverse. Anche in questo caso l’unione può essere tra gameti maschili e femminili
non ridotti oppure tra un gamete non ridotto di una specie e un gamete normale dell’altra,
oppure, ancora, per unione di gameti normali delle due specie e successivo
raddoppiamento del numero di cromosomi dell’ibrido interspecifico sterile.
In ogni caso, gli allopoliploidi presentano un livello di fertilità superiore a quello degli
autopoliploidi in quanto ciascuno dei due assetti cromosomici provenienti dalle due specie
ha la possibilità di appaiarsi normalmente alla meiosi.
Non è quindi casuale che tra le specie coltivate i casi di autopoliploidia siano
estremamente rari mentre, al contrario, l’allopoliploidia è di gran lunga la più diffusa e ad
essa è riconosciuto un ruolo estremamente importante per la loro evoluzione.
49
Emblematica, da questo punto di vista, è la storia evolutiva dei generi Triticum, Brassica e
Fragaria.
Anche gli allopoliploidi possono essere costituiti artificialmente facendo ricorso alla
ibridazione interspecifica tra individui 2n. Da questa è possibile ottenere un ibrido il quale,
pur essendo in molti casi in grado di sopravvivere e vegetare regolarmente, risulta però
sterile proprio per l’impossibilità dei cromosomi delle due specie di appaiarsi regolarmente
nel corso della meiosi. Il trattamento con colchicina dell’apice vegetativo, inducendo il
raddoppiamento del numero di cromosomi, ripristina la regolarità del processo meiotico
e,quindi, anche la fertilità dell’ibrido.
Esistono anche almeno due esempi di specie nuove costituite artificialmente con questa
procedura, Triticale e Tritordeum derivate, rispettivamente dall’incrocio Triticum x Secale e
Triticum x Hordeum.
Un fenomeno diverso, che ha contribuito moltissimo all’evoluzione di alcune specie
coltivate e che implica comunque l’ibridazione interspecifica è l’introgressione. Con
questo processo, ad una iniziale ibridazione interspecifica tra due specie più o meno
lontane fa seguito una serie di ulteriori incroci dell’ibrido su una delle due specie parentali
con conseguente ricostituzione della maggior parte del genoma della specie ricorrente e
con l’acquisizione di una parte più o meno rilevante del genoma dell’altra. Esempi di
questo si hanno nel mais (Zea mais) e nel girasole (Helianthus annuus).
GAMETOGENESI NORMALE (gameti ridotti)
Specie A
pianta 2
Specie A
pianta 1
Specie B
pianta 1
GAMETOGENESI ALTERATA (gameti non ridotti)
Autotetraploide eterozigote
Allotetraploide
50
IL SISTEMA RIPRODUTTIVO NEI VEGETALI
Il sistema riproduttivo dei vegetali può essere definito come “insieme di strutture e di
processi che predispongono ad un certo tipo di impollinazione e determinano il tipo
di gamia”
Si parla di autoimpollinazione quando il polline prodotto dall’organo maschile (antera) di
un fiore cade, o viene portato, sull’organo femminile (pistillo) dello stesso fiore o di un altro
fiore dello stesso individuo.
Si parla invece di alloimpollinazione quando il polline di un fiore cade, o viene portato,
sul pistillo del fiore di un individuo diverso da quello sul quale il polline è maturato.
Analogamente, si parla di autofecondazione o di autogamia quando si uniscono gameti
maschili (♂) e femminili (♀) prodotti dallo stesso individuo e di allofecondazione o
fecondazione incrociata o allogamia quando si uniscono gameti ♂ e ♀ prodotti da
individui diversi.
Il fiore di una pianta può essere completo o incompleto e, in relazione alla presenza o
meno di tutti gli organi demandati a svolgere la funzione riproduttiva, un fiore può essere
classificato come
ermafrodita (♀) = se è provvisto di stami e pistilli;
staminato o maschile (♂) = se è provvisto solamente di stami;
pistillato o femminile (♀) = se è provvisto solamente di pistilli.
Con la stessa terminologia vengono indicati, rispettivamente, gli individui e le popolazioni
di individui anche se, in questo caso, la classificazione si complica con denominazioni
nuove.
Singola pianta:
ermafrodita = solamente con fiori ermafroditi
monoica = con fiori maschili e femminili
androica = solamente con fiori maschili
ginoica = solamente con fiori femminili
andromonoica = con fiorio ermafroditi e maschili
ginomonoica = con fiori ermafroditi e femminili
trimonoica = con fiori ermafroditi, maschili e femminili
Gruppo di piante (popolazione o specie):
ermafrodita = solamente con piante ernmafrodite
monoica = solamente con piante monoiche
dioica = con piante androiche e ginoiche
androdioica = con piante ermafrodite e androiche
ginodioica = con piante ermafrodite e ginoiche
subdioica = con piante ermafrodite, androiche e ginoiche
51
Una delle classificazioni adottabili per le specie a fiore, che mette in relazione il tipo di
struttura fiorale con il tipo di gamia, può essere la seguente:
Specie dioiche = fiori unisessuati
I fiori ♂ e ♀ sono portati da individui diversi (es. canapa, spinacio,
luppolo, asparago)
Allogamia esclusiva; autogamia impossibile
Specie monoiche
dicline = fiori unisessuati (♂ e ♀) sullo stesso individuo (es. mais, ricino)
Allogamia prevalente; autogamia possibile
monocline = fiori ermafroditi (specie ermafrodite). Vengono distinte in:
dicogame = stigmi e antere dello stesso fiore maturano in tempi
diversi
protandre (proterandre) = le antere maturano prima degli
stigmi
protogine (proterogine) = gli stigmi maturano prima delle
antere
Allogamia favorita; autogamia non esclusa
omogame = stigmi e antere dello stesso fiore maturano
contemporaneamente
casmogame = fiori aperti alla maturazione del polline e
dello stigma
Autogamia favorita
cleistogame = fiori chiusi alla maturazione del polline e
dello stigma
Autogamia esclusiva
Fatta eccezione per le specie dioiche, in tutte le altre situazioni possono verificarsi casi di
autoincompatibilità e/o maschiosterilità che possono condizionare più o meno il tipo di gamia al
quale la pianta è predisposta in base alla sola struttura fiorale e ai tempi di maturazione degli organi
sessuali.
52
POPOLAZIONI ALLOGAME: Generalità
ALLOGAMIA = è il sistema riproduttivo in base al quale la fecondazione avviene tra
gameti maschili e femminili prodotti da individui diversi.
Si definisce come allogama una specie nella quale il sistema riproduttivo abituale è
l’allogamia.
Se si ammette che in una popolazione di una specie allogama l’unione tra gameti maschili
e femminili avvenga in maniera casuale, vuol dire che ogni gamete femminile ha uguali
probabilità di unirsi con ogni gamete maschile in circolazione al momento della
impollinazione.
Questo vuole anche dire che ogni individuo ha uguali probabilità di unirsi con ciascuno
degli altri individui della popolazione o, in altre parole, che non vi sono limiti alla casualità
delle unioni.
Questa situazione corrisponde a quella che, con termine appropriato, viene indicata come
panmissia e rappresenta la situazione di riferimento per ricavare indicazioni sulla struttura
genetica di una popolazione allogama.
Se si considera un locus con due alleli alternativi A1/A2, in una popolazione saranno
presenti, con frequenza diversa, individui che, a quel locus, potranno essere di genotipo
A1A1, A1A2, A2A2.
Quando questa popolazione si riproduce, la frequenza con la quale, nella popolazione,
vengono prodotti gameti A1 e A2 dipenderà dalla frequenza con la quale i genotipi A1A1,
A1A2, A2A2 sono presenti nella popolazione stessa.
La frequenza con la quale, al momento della riproduzione, avvengono le unioni tra gameti
A1, tra gameti A1 e A2, o tra gameti A2, dipenderà perciò dalla frequenza con la quale i
gameti A1 e A2 vengono prodotti e, in definitiva, dalla frequenza dei genotipi A1A1, A1A2,
A2A2.
Si può perciò anche dire che la frequenza con la quale, in una generazione, sono presenti
genotipi A1A1, A1A2, A2A2 dipende dalla frequenza con la quale, nella generazione
precedente, si sono formati gameti A1 e A2 e, quindi, dalla frequenza con la quale quegli
stessi genotipi erano presenti.
Questa situazione ha conseguenze genetiche importanti in quanto:
-
condiziona la struttura genetica delle popolazioni allogame naturali
determinandone, nel contempo, la risposta adattativa alle sollecitazioni della
selezione naturale e, quindi, l’evoluzione;
-
determina le modalità con le quali la selezione artificiale deve intervenire per
modificare geneticamente tali popolazioni così da migliorarne il comportamento
come risposta ai fattori colturali.
53
Prendiamo in considerazione una coppia all’elica A/a e immaginiamo di realizzare
l’incrocio tra due individui omozigoti AA e aa.
P1 AA
x
F1
Aa
P2 aa
Dall’incrocio otteniamo una popolazione di individui tutti di genotipo Aa.
Se ora immaginiamo di allevare questa popolazione e di lasciare che si riproduca in
allogamia, cosa succede?
Tutti gli individui della popolazione produrranno gameti A e gameti a, sia maschili che
femminili e, per quanto visto parlando di meiosi, i due tipi di gameti saranno prodotti in
uguali proporzioni: 50% A e 50% a.
Se ora indichiamo con
p = frequenza dei gameti A
q = frequenza dei gameti a
p = f(A)
q = f(a)
possiamo anche dire che tanto i gameti maschili quanto quelli femminili conterranno
gameti A e a con uguale frequenza e cioè:
p = q = 0,5
per cui,
p+q=1
In allogamia , questi gameti si uniranno in modo casuale e il risultato sarà il seguente:
gameti
♂
gameti
♀
A
p = 0,5
a
q = 0,5
A
p = 0,5
AA
p2 = 0,25
Aa
pq = 0,25
a
q = 0,5
Aa
pq = 0,25
aa
q2 = 0,25
Dall’unione casuale dei gameti maschili e femminili si origina perciò una popolazione nella
quale i genotipi AA, Aa, aa sono presenti con frequenza
0,25 AA + 0,5 Aa + 0,25 aa = 1,00
o, usando i simboli p e q,
p2 + 2pq + q2 = 1
54
Esempio numerico
Prendiamo in considerazione sempre la coppia allelica A/a.
1) - A = “fiore rosso”;
a = “fiore bianco”
2) - assenza di dominanza; questo significa che l’osservazione fenotipica
consente di riconoscere i genotipi Aa che saranno a “fiore rosa”.
3) - prendiamo in considerazione 120 individui estratti a caso dalla
popolazione.
La classificazione di questi 120 individui in base al colore del fiore porta al seguente
risultato:
60 individui con fiore rosso
AA
48 individui con fiore rosa
Aa
popolazione (1)
12 individui con fiore bianco
aa
Ipotesi:
Queste sono frequenze fenotipiche assolute alle quali corrispondono uguali frequenze
genotipiche assolute.
Calcolo delle frequenze fenotipiche e genotipiche relative:
60 / 120 = 0,5 = f(AA) = P
fiore rosso
48 / 120 = 0,4 = f(Aa) = H
fiore rosa
12 / 120 = 0,1 = f(aa) =Q
fiore bianco
Se questa poopolazione si riproduce, produce gameti A e a; con quale frequenza?
60 individui AA
120 loci
120 loci A
168 loci A
48 loci A
48 individui Aa
96 loci
48 loci a
72 loci a
12 individui aa
120 individui
24 loci
240 loci
24 loci a
240 loci
La frequenza con la quale saranno prodotti gameti A e a sarà allora data da:
frequenza dei loci A = 168 / 240 = 7 / 10 = 0,7 = frequenza dei gameti A = f(A) = p
frequenza dei loci a = 72 / 240 = 3 / 10 = 0,3 = frequenza dei gameti a = f(a) = q
Poiché ogni locus è occupato da un gene, si può anche dire che frequenze geniche e
frequenze gametiche coincidono.
Allo stesso risultato si sarebbe giunti utilizzando le frequenze genotipiche assolute:
(60 + 24) / 120 = 84 / 120 = 0,7 = f(A) = p ; (12 + 24) / 120 = 36 / 120 = 0,3 = f(a) = q
oppure le frequenze genotipiche relative:
0,5 + (0,4 / 2) = P + ( H / 2) = 0,7 = f(A) = p ; 0,1 + (0,4 / 2) = Q + (H / 2) = 0,3 = f(a) = q
55
Date queste frequenze gametiche (geniche), la probabilità di unione tra due gameti A, un
gamete A e un gamete a o due gameti a sarà data dal prodotto delle frequenze con le
quali i gameti A e a sono presenti sulla totalità dei gameti prodotti dalla popolazione.
Allora:
gameti
♂
gameti
♀
A
a
A
p = 0,7
a
q = 0,3
p = 0,7
p2
AA
= 0,49
Aa
pq = 0,21
q = 0,3
Aa
pq = 0,21
aa
q2 = 0,09
Dall’unione casuale dei gameti maschili e femminili si origina pertanto una popolazione
nella quale i tre genotipi AA, Aa, aa sono presenti con frequenza:
0,49 AA + 0,42 Aa + 0,09 aa = 1,00
(popolazione 2)
o, usando i simboli p e q,
p2 AA + 2pq Aa + q2 aa = 1,00
Se ora si calcolano le frequenze geniche (gametiche) in questa popolazione, si ha:
p = f(A) = 0,49 + ½ 0,42 = 0,49 + 0,21 = 0,7
q = f(a) = 0,09 + ½ 0,42 = 0,09 + 0,21 = 0,3
Questo vuole dire che, passando dalla popolazione 1 alla popolazione 2 si sono modificate
le frequenze genotipiche, ma non si sono modificate le frequenze geniche e quindi, se
questa popolazione si riproduce, si forma un’altra popolazione nella quale le frequenze
genotipiche saranno sempre:
0,49 (AA) + 0,42 (Aa) + 0,09 (aa) = 1,00
dove p = 0,7 e q = 0,3
In generale, si può allora dire che se in una popolazione nella quale le unioni
avvengono a caso (popolazione panmittica) sono presenti due alleli, A e a con
frequenze, rispettivamente, p = f(A) e q = f(a) tali che p + q = 1,00, le frequenze
genotipiche sono:
p2 (AA) + 2pq (Aa) + q2 (aa) = 1,00
Questa situazione, definita come “situazione di equilibrio” viene raggiunta in
un’unica generazione di riproduzione e si mantiene indefinitamente finché
permangono le condizioni di unioni casuali, di assenza di mutazioni e di migrazioni
preferenziali e di assenza di selezione. La popolazione è indicata come
“popolazione in equilibrio Hardy-Weimberg”.
56
POPOLAZIONI AUTOGAME: Generalità
AUTOGAMIA = è il sistema riproduttivo in base al quale la fecondazione avviene tra
gameti maschili e femminili prodotti dallo stesso individuo.
Si definisce come autogama una specie nella quale il sistema riproduttivo abituale è
l’autogamia.
In una popolazione autogama ciascun individuo si riproduce pertanto in maniera
indipendente dagli altri individui della popolazione. Questa situazione ha conseguenze
genetiche importanti in quanto determina un progressivo spostamento della popolazione
verso lo stato di completa omozigosi. Questo significa che una popolazione autogama,
non essendovi, almeno in linea teorica, alcuno scambio di materiale genetico tra gli
individui che la costituiscono, si avvicina progressivamente alla situazione nella quale tutti
gli alleli presenti sono fissati in condizione omozigote e, quindi, tutti gli individui possono
essere considerati degli omozigoti completi.
Come punto di partenza si può immaginare una popolazione di individui F1 ottenuti
dall’unione tra due parentali omozigoti per due alleli alternativi: AA e aa. Da tale unione si
originerà una popolazione di tutti individui Aa: 100% eterozigoti
Se questa popolazione si riproduce in autogamia, ogni individuo genera una discendenza
F2 nella quale individui AA, Aa, aa sono presenti in rapporto 1 : 2 : 1. Nel complesso,
quindi, la popolazione F1 genera una popolazione F2 nella quale saranno presenti individui
AA, Aa, aa in rapporto di 1 : 2 : 1 e nella quale individui omozigoti ed eterozigoti saranno
presenti in uguali proporzioni: ½ omozigoti e ½ eterozigoti.
Immaginiamo ora che questa popolazione F2 vada incontro ad una successiva
generazione di riproduzione in autogamia e che tutti gli individui della popolazione,
indipendentemente dal loro genotipo, siano ugualmente fertili. Ogni individuo AA originerà
una discendenza F3 tutta composta di individui AA, ogni individuo aa originerà una
discendenza F3 di tutti individui aa e ogni individuo Aa originerà una discendenza F3 nella
quale individui AA, Aa, aa saranno presenti in rapporto di 1 : 2 : 1. Poiché la fertilità degli
individui viene supposta uguale, la quota di popolazione che si origina dagli individui
omozigoti (pari a ½) sarà comunque costituita da individui omozigoti (1/2 omo), mentre la
quota di popolazione che si origina dagli individui eterozigoti (pari a ½) sarà a sua volta
costituita da una metà, pari a ¼ del totale, di individui omozigoti (1/4 omo) e da una metà,
pari a ¼ del totale, di individui eterozigoti (1/4 etero). Nell’insieme, si origina quindi una
popolazione F3 nella quale individui omozigoti ed eterozigoti saranno presenti in rapporto
di 3 : 1: ¾ omozigoti e ¼ eterozigoti.
Se questo ragionamento viene ripetuto passando dalla generazione F3 alla generazione F4
con un’ulteriore generazione di autogamia, sempre supponendo un’uguale fertilità degli
individui indipendentemente dal loro genotipo, ci si rende conto che i ¾ della popolazione
che si originano dagli individui omozigoti saranno comunque costituiti da individui
omozigoti (3/4 = 6/8 omo), mentre ¼ della popolazione che si origina dagli individui
eterozigoti sarà costituito da una metà, pari a 1/8 del totale, di individui omozigoti (1/8
omo) e da una metà, pari a 1/8 del totale, di individui eterozigoti (1/8 etero). Nell’insieme
si origina quindi una popolazione F4 nella quale individui omozigoti ed eterozigoti saranno
presenti in rapporto di 7 : 1: 7/8 omozigoti e 1/8 eterozigoti.
57
Le considerazioni ora fatte possono essere generalizzate e sintetizzate nella tabella
seguente:
Tab. 1: Modificazione delle frequenze genotipiche in seguito a successive
autofecondazioni di una popolazione originatasi da un individuo eterozigote per una
coppia allelica.
Genotipi
Eterozigoti
Omozigoti
1/2
-
1
1
-
1
-
1/4
1/2
1/4
1/2
1-1/2 = 1/2
F3
1/4+1/8
1/4
1/4+1/8
1/4=1/22
1-1/22 = 3/4
F4
3/8+1/16
1/8
3/8+1/16
1/8=1/23
1-1/23 = 7/8
F5
7/16+1/32
1/16
7/16+1/32
1/16=1/24
1-1/24 = 15/16
.
.
.
.
.
.
.
.
.
.
.
.
Fm+1
1/2 (1-1/2m)
1/2m
1/2(1-1/2m)
1/2m
1-1/2m=(2m-1)/2m
Generazione
A1A1
A1A2
A2A2
P
1/2
-
F1
-
F2
Sulla base di queste considerazioni è allora possibile esprimere i rapporti di segregazione
tra omozigoti ed eterozigoti nella forma indicata alla colonna 3 della tabella alla pagina
successiva (Tabella 2).
Si può cioè concludere che, ad ogni generazione, la popolazione è composta da individui
omozigoti ed eterozigoti che stanno tra loro in rapporto (2m-1) : 1 su una popolazione di
dimensioni pari a (2m-1) + 1 = 2m.
Da qui la conferma che la frequenza degli omozigoti nella popolazione dopo m
generazioni di autofecondazione è pari a (2m-1)/2m come indicato nella tabella 1.
I rapporti suindicati possono essere verificati direttamente a livello fenotipico se ci si trova
in condizioni di assenza di dominanza (additività), dominanza parziale, o sovradominanza,
cioè quando, comunque, la classe fenotipica corrispondente agli individui eterozigoti è
individuabile.
58
In condizioni di dominanza completa, in corrispondenza delle diverse generazioni il
rapporto di segregazione fenotipica si modificherà come risulta dalla tabella 2.
E’ evidente che, in tale tabella, la differenza tra le due classi, che è pari a
(2m + 1) - (2m - 1) = 2,
rimane costante come valore assoluto ma va via via diminuendo come valore relativo,
all’aumentare delle generazioni di autogamia. In altri termini, al crescere delle generazioni
di autofecondazione, la frequenza degli eterozigoti tende a 0 e, parallelamente, il rapporto
tra individui con fenotipo dominante e quelli con fenotipo recessivo tende a 1.
Tab. 2 : Modificazione dei rapporti di segregazione genotipica tra omozigoti ed eterozigoti
e di segregazione fenotipica in condizioni di assenza di dominanza e di dominanza
completa a seguito di successive autofecondazioni di una popolazione F2 originatasi da un
individuo eterozigote per una coppia allelica.
Generazione
n°
gener.
autof.
Rapporto di
segregazione
genotipica
Rapporti di segregazione fenotipica
Totale
Omo : Etero
-
1
Assenza di dominanza
Dominanza completa
Dom.
Int.
Rec.
Dom.
Rec.
-
1
-
1
-
Totale
F1
-
F2
1
1 = 21-1 : 1
21
1
:
2
:
1
3
:
1
22
F3
2
3 = 22-1 : 1
22
3
:
2
:
3
5
:
3
23
F4
3
7 = 23-1 : 1
23
7
:
2
:
7
9
:
7
24
F5
4
15 = 24-1 : 1
24
15 :
2
: 15
17
:
15
25
F6
5
31 = 25-1 : 1
25
31 :
2
: 31
33
:
31
26
F7
6
63 = 26-1 : 1
26
63 :
2
: 63
65
:
63
27
.
.
.
.
.
.
.
.
.
Fm+1
m
2m-1
.
.
.
: 1
2m
2m-1 : 2
.
.
.
: 2m-1
2m+1
:
.
2m-1
2m
59
Ciò significa che se si prende in considerazione la popolazione che si origina per
successive autofecondazioni partendo da un unico individuo F1(A1A2) generato
dall’ unione tra due individui omozigoti per due alleli alternativi (A1A1 x A2A2), tale
popolazione, dopo m generazioni (con m sufficientemente grande) sarà composta
da tutti individui omozigoti i quali, se i due alleli A1 e A2 sono indifferenti ai fini della
sopravvivenza (non conferiscono una diversa fitness agli individui che li portano)
tenderanno a ripartirsi in misura uguale tra i due possibili genotipi A1A1 e A2A2.
Analoghe considerazioni possono essere svolte se il discorso viene esteso a due o più
coppie alleliche. In questo caso, tuttavia deve essere fatta una netta distinzione tra coppie
alleliche indipendenti e coppie alleliche associate.
Se le coppie alleliche coinvolte sono due (A1A2 e B1B2) e sono tra loro indipendenti (sono
situate su due coppie di cromosomi omologhi), ad ogni generazione di autogamia nella
discendenza compaiono, oltre agli omozigoti completi (A1A1B1B1, A1A1B2B2, A2A2B1B1,
A2A2B2B2), anche gli omozigoti o eterozigoti parziali (A1A2B1B1, A1A2B2B2, A1A1B1B2,
A2A2B1B2) e gli eterozigoti (A1A2B1B2).
Volendo calcolare il rapporto con il quale omozigoti completi, omozigoti/eterozigoti parziali
e eterozigoti sono presenti nella popolazione con il procedere delle generazioni di
autofecondazione, è necessario partire dalla considerazione che, per una coppia allelica, il
rapporto tra omozigoti ed eterozigoti dopo m generazioni di autofecondazione è pari a
(2m-1) : 1.
Poiché le due coppie alleliche sono indipendenti, è evidente che la quota di omozigoti per
una coppia potrà essere indifferentemente omozigote o eterozigote per l’altra.
La frequenza con la quale si formeranno omozigoti completi, omozigoti/eterozigoti parziali
ed eterozigoti completi, dipenderà allora dalla frequenza relativa con la quale le due
situazioni genotipiche (omo o etero) relative a ciascuna coppia saranno presenti nella
popolazione e cioè dalla casualità con la quale i 2m-1 omozigoti e l’1 eterozigote di una
coppia si assortiranno con i 2m-1 omozigoti e con l’1 eterozigote dell’altra coppia.
Tale frequenza è allora pari al prodotto tra i due rapporti omo/etero relativi alle due coppie
e cioè all’espressione:
[(2m –1) : 1]2
che, sviluppata, è pari a:
(2m –1)2 : 2(2m –1) : 1
Su questa base si può allora ricavare un’espressione generale che consenta di calcolare
la composizione della popolazione dopo m generazioni di autofecondazione allorché siano
coinvolte n coppie alleliche.
Come si è già visto parlando di una sola coppia allelica, la composizione della popolazione
dopo m generazioni di autofecondazione è data dalla espressione
[(2m-1) + 1]
che vale per ciascuna coppia allelica.
60
La composizione della popolazione rispetto a due coppie alleliche indipendenti sarà allora
data da:
[(2m-1) + 1] [(2m-1) + 1] = (2m-1)2 + (2m-1) + (2m-1) + 1 = (2m-1)2 + 2(2m-1) + 1 = [(2m-1) + 1]2
Tab. 3: Modificazione delle frequenze genotipiche in seguito a successive
autofecondazioni di una popolazione F2 originatasi da un individuo eterozigote per due
coppie alleliche.
Generazione
N° generazioni
di
autogamia
Omozigoti
completi
Rapporti
Omozigoti
parziali
F1
Totale
Eterozigoti
-
0
0
1
1
F2
1
1
2
1
22
F3
2
9
6
1
(22)2
F4
3
49
14
1
(23)2
F5
4
225
30
1
(24)2
.
.
.
.
.
.
.
.
.
.
.
.
Fm+1
m
(2m-1)2
2(2m-1)
1
(2m)2
Ammettendo segregazione indipendente, entro ciascuno dei due gruppi più frequenti
(omozigoti completi e omozigoti/eterozigoti parziali) i quattro genotipi possibili saranno
presenti con uguale frequenza pari, rispettivamente, a ¼ della frequenza propria di
ciascun gruppo.
Anche in questo caso, comunque, vale quanto precedentemente detto a proposito di una
coppia allelica:
se si prende in considerazione la popolazione che si origina per successive
autofecondazioni partendo da un unico individuo F1(A1A2B1B2) generato dall’unione
tra due individui omozigoti per due coppie indipendenti di alleli alternativi (A1A1B1B1
x A2A2B2B2), tale popolazione, dopo m generazioni (con m sufficientemente grande),
sarà composta da tutti individui omozigoti i quali, se gli alleli A1, A2, B1 e B2 sono
indifferenti ai fini della sopravvivenza (non conferiscono una diversa fitness agli
individui che li portano) tenderanno a ripartirsi in misura uguale fra i quattro
possibili genotipi A1A1B1B1, A1A1B2B2, A2A2B1B1, A2A2B2B2.
61
100
80
60
40
omo comp
20
0
omo parz
F1
F2
eterozigoti
F3
F4
F5
Fig. 1: Variazione del rapporto percentuale tra genotipi omozigoti completi,
omozigoti/eterozigoti parziali e eterozigoti completi al procedere delle generazioni di
autofecondazione partendo da un ibrido eterozigote per due coppie alleliche.
Se le due coppie alleliche controllano due coppie di caratteri alternativi, la distribuzione
fenotipica della popolazione rispetto ai due caratteri considerati dipenderà, anche in
questo caso, dal grado di dominanza espresso entro ciascuna coppia allelica.
Se, infatti, per ciascuna delle due coppie alleliche vale un rapporto di dominanza completa
(A1>A2 e B1>B2), la popolazione si suddividerà in quattro classi fenotipiche: una sarà la
classe di maggior frequenza e sarà rappresentata dal fenotipo doppio dominante; due
saranno mediamente frequenti, saranno presenti con frequenze tra loro uguali e saranno
rappresentate, rispettivamente, dal fenotipo dominante per il carattere controllato dalla
prima coppia e quello recessivo per il carattere controllato dalla seconda o viceversa; la
quarta classe sarà la meno frequente e sarà rappresentata dal fenotipo doppio recessivo.
Se, invece, tra gli alleli entro ciascuna delle due coppie considerate vi è una situazione
d'assenza di dominanza (additività), dominanza parziale, o di sovradominanza, nella
popolazione saranno individuabili nove classi fenotipiche, quattro delle quali attribuibili ai
quattro omozigoti completi (A1A1B1B1; A1A1B2B2; A2A2B1B1; A2A2B2B2), quattro ai quattro
omozigoti (eterozigoti) parziali (A1A1B1B2; A1A2B1B1; A1A2B2B2; A2A2B1B2) e una
all’eterozigote completo (A1A2B1B2).
Le stesse considerazioni valgono per un numero indefinito di coppie alleliche indipendenti.
Se, infatti, si prendono in considerazione 3 coppie alleliche e si ripete il ragionamento, è
evidente che la frequenza con la quale si formeranno genotipi omozigoti completi,
omozigoti parziali ed eterozigoti completi dipenderà dalla frequenza con la quale i (2 m – 1)
omozigoti e l’1 eterozigote per ciascuna coppia si assortiranno con i (2m –1) e l’1
eterozigote di ciascuna delle altre due coppie.
62
La composizione della popolazione sarà allora data da:
[(2m-1) + 1]3
e per n coppie alleliche indipendenti l’espressione generale che ci consente di calcolare la
composizione della popolazione dopo m generazioni di autofecondazione è data da:
[(2m-1) + 1]n
(1)
Questa espressione, se opportunamente utilizzata, non consente solamente di calcolare il
rapporto tra omozigoti completi, omozigoti parziali ed eterozigoti, ma anche di calcolare,
ad ogni generazione, la composizione della popolazione relativamente alla frequenza di
ciascuna classe di omozigoti parziali per n-1, n-2, n-3,.......n-(n-1) coppie alleliche e il
livello di omozigosi ( frequenza dei loci in condizione omozigote ) raggiunto, nel suo
insieme, dalla popolazione. Questa possibilità può essere illustrata con un esempio.
Esempio.
Supponiamo di voler calcolare, dopo 5 generazioni di autofecondazione, la composizione
della popolazione che si origina da una generazione F1 eterozigote per 4 coppie alleliche
indipendenti; perciò, m = 5 e n = 4.
Applicando l’espressione (1), si ha:
[(25-1) + 1]4 = (25-1)4 + 4(25-1)3 + 6(25-1)2 + 4(25-1) + 1 = 314 + 4x313 + 6x312 + 4x31 + 1 =
= 923521 + 4x29791 + 6x 961 + 4x31 + 1 = 923.521 + 119.164 + 5.766 + 124 + 1 = 1.048.576
In questo polinomio, ciascun membro si riferisce ad una determinata situazione genotipica
ed esprime la frequenza con la quale tale situazione è presente all’interno della
popolazione. Così:
(25 – 1)4 = 314
=
923.521 omo. per 4 coppie all’eliche (c.a.)
4(25 – 1)3 = 4 x 313
=
119.164 omo. per 3 c.a.; etero. per 1 c.a.
6(25 – 1)2 = 6 x 312
=
5.766 omo. per 2 c.a.; etero. per 2 c.a.
4(25 – 1) = 4 x 31
=
124 omo. per 1 c.a.; etero. per 3 c.a.
1
=
Totale = (25)4
=
1 etero. per 4 coppie alleliche
__________
1.048.576 individui = 4.194.304 loci
63
Il livello di omozigosi o di eterozigosi (quota di loci in condizione omozigote o
eterozigote, rispettivamente) della popolazione nel suo insieme può essere allora calcolato
moltiplicando il numero di genotipi presenti in ciascuna classe per il numero di loci
omozigoti o eterozigoti proprio di ogni classe e sommando tra loro i valori così ottenuti:
n° loci omozigoti
923.521 x 4 = 3.694.084
n° loci eterozigoti
-
119.164 x 3 =
357.492
119.164 x 1 = 119.164
5.766 x 2 =
11.532
5.766 x 2 = 11.532
124 x 1 =
124
__________________
4.063.232
124 x 3 =
372
1x4=
4
___________________
131.072
Complessivamente, dei 4.194.304 loci disponibili nella popolazione, 4.063.232, pari al
96.875% sono perciò in condizione omozigote e solamente 131.072, pari al 3.125%, sono
in condizione eterozigote.
Il valore del 96.875% rappresenta quindi il livello di omozigosi della popolazione nella
quale è presente una quota di individui omozigoti completi pari all’ 88.078%.
Le considerazioni svolte fino a questo momento sugli effetti genetici dell’autogamia
porterebbero a concludere che una popolazione autogama, lasciata a sé stessa, si sposta
gradualmente verso una situazione di completa omozigosi e che, pertanto, essa può
essere considerata come costituita da un insieme di individui omozigoti.
Ora, è necessario precisare che si definisce come “linea pura”, la discendenza
omozigote ottenuta per autofecondazione da un individuo omozigote. Una linea pura è
pertanto costituita da individui tutti geneticamente identici tra loro e all’individuo che li ha
generati.
Poiché un individuo omozigote che si riproduce per autogamia genera una discendenza di
individui tutti omozigoti, geneticamente identici tra loro e all’individuo che li ha generati, è
evidente che quando in una popolazione un individuo ha raggiunto la situazione
omozigote, esso genera una linea pura.
Poiché questo accade per tutti gli individui della popolazione, una popolazione autogama
può allora essere considerata come costituita da tante linee pure che convivono nella
popolazione senza scambiarsi materiale genetico.
Questa situazione, vera in linea teorica, non trova riscontro nella realtà per almeno tre
motivi: l’insorgenza di mutazioni spontanee, il tasso naturale di incrocio, la presenza di loci
associati.
64
Come si è detto parlando di mutazioni, ogni gene è caratterizzato da un determinato tasso
di mutazione (probabilità che si verifichi un certo tipo di mutazione in funzione del tempo;
es. ad ogni generazione) e ogni popolazione è caratterizzata da una certa frequenza di
mutazione (numero di eventi di mutazione di un certo tipo che si verificano in una
popolazione di cellule o di individui). Una volta che un certo genotipo abbia raggiunto la
situazione omozigote in seguito a ripetute autofecondazioni, non è detto che in tale
situazione rimanga in perpetuo.
Quando si è parlato di relazione tra strutture fiorali e tipo di gamia si è inoltre visto che
l’allogamia e l’autogamia assolute rappresentano delle eccezioni e che la maggior parte
delle specie vegetali è caratterizzata da strutture fiorali che consentono sempre, anche se
in diversa misura, tanto l’autofecondazione quanto la fecondazione incrociata. Ciò significa
che anche in una specie ad autogamia prevalente esiste sempre la possibilità che si
verifichino unioni tra individui diversi con la conseguente formazione di individui eterozigoti
che daranno luogo a generazioni segreganti.
Nel considerare gli effetti genetici dell’autogamia si è fatto riferimento a coppie alleliche
indipendenti per le quali la segregazione dipende dall’assortimento casuale dei cromosomi
(centromeri) materni e paterni nella profase della prima divisione meiotica. Sappiamo
tuttavia che questa non è la situazione generale e che esistono loci associati la cui
ricombinazione dipende dalla loro distanza sul cromosoma e cioè dalla frequenza con la
quale, fra tali loci, avvengono eventi di Crossing Over. Un individuo eterozigote per due o
più coppie alleliche genera pertanto una discendenza nella quale individui omozigoti
saranno presenti in misura inferiore rispetto a quanto previsto dalla segregazione
indipendente.
Una popolazione di una specie autogama la possiamo quindi considerare caratterizzata da
un elevato tasso di omozigosi, ma anche da una frequenza più o meno elevata di
eterozigosi in relazione alla frequenza di mutazione, al controllo esercitato dalla struttura
fiorale sul tipo di gamia ed alla frequenza di ricombinazione o, in altri termini, all’intensità
del linkage tra loci associati.
Il considerare una popolazione autogama come costituita da linee pure conviventi è quindi
un’astrazione del tutto teorica anche se molto conveniente specialmente quando si devono
elaborare strategie di miglioramento genetico.
Se una popolazione è caratterizzata da un certo livello di autogamia, si dice che essa
presenta anche un certo livello di inbreeding. L’inbreeding può infatti essere definito come
quel sistema di unione o quella procedura riproduttiva che prevede l’unione tra individui
imparentati o, detto in termini genetici, che presentano alleli comuni.
Il livello di inbreeding è infatti misurato dal “Coefficiente di inbreeding”, che è indicato
con F e che rappresenta la probabilità che ad una certa generazione si formino individui
omozigoti per alleli identici (repliche dello stesso allele).
E’ allora evidente che la forma più stretta di inbreeding è data dall’autofecondazione di
individui omozigoti nella quale i gameti maschili e femminili prodotti dallo stesso individuo
sono tra loro identici e, unendosi, danno luogo ad un inbreeding pari a 1.
La presenza, in una popolazione di un livello di autogamia più o meno elevato comporta
quindi, per quella stessa popolazione, un corrispondente livello di inbreeding che, ad una
certa generazione, possiamo indicare con F.
65
Si può dimostrare che in una popolazione panmittica nella quale sussista un livello di
inbreeding pari ad F e ad un locus siano presenti due alleli alternativi A e a con frequenza
rispettiva p e q, tale che p + q =1, gli omozigoti si formano con frequenza maggiore di
quanto previsto dalla legge di Hardy-Weimberg, tale incremento è proporzionale ad F ed è
pari a pqF per ciascuno dei due alleli alternativi presenti a quel locus.
L’espressione di pag 56 relativa alla situazione di equilibrio può quindi essere riscritta nel
modo seguente:
(p2 + pqF) AA + (2pq – 2pqF) Aa + (q2 + pqF) aa = 1
Questa espressione rappresenta la situazione generale di equilibrio in base alla quale, se
F = 0 (panmissia), nella popolazione saranno presenti i tre genotipi AA, Aa, aa con
frequenza rispettiva di p2, 2pq, q2 e, se F = 1(autogamia assoluta di materiale
omozigote), nella popolazione saranno presenti solamente genotipi AA e aa con
frequenza rispettiva di (p2 + pqF) e (q2 + pqF). In un caso e nell’altro le frequenze con le
quali i diversi genotipi sono presenti nella popolazione dipendono dai valori di p e q e cioè
dalla frequenza dei due alleli.
66