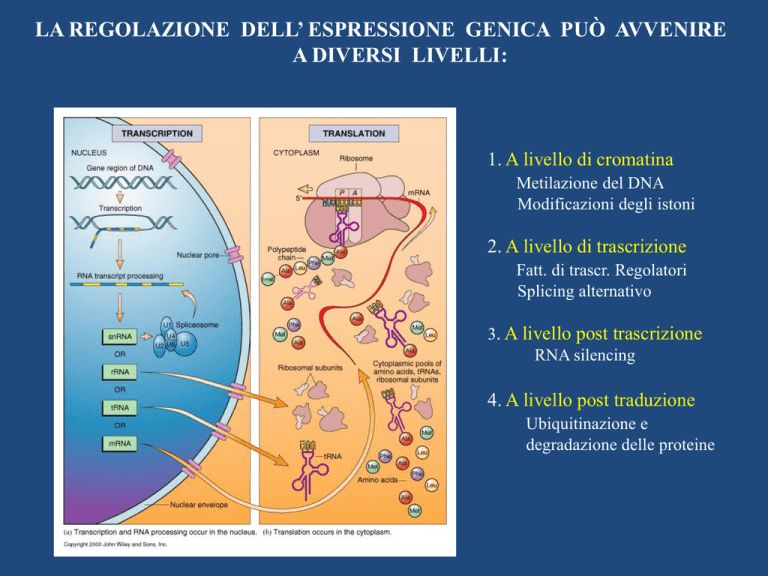
LA REGOLAZIONE DELL’ ESPRESSIONE GENICA PUÒ AVVENIRE
A DIVERSI LIVELLI:
1. A livello di cromatina
Metilazione del DNA
Modificazioni degli istoni
2. A livello di trascrizione
Fatt. di trascr. Regolatori
Splicing alternativo
3. A livello post trascrizione
RNA silencing
4. A livello post traduzione
Ubiquitinazione e
degradazione delle proteine
1. REGOLAZIONE DELL’ ESPRESSIONE GENICA A
LIVELLO DI CROMATINA
1a: METILAZIONE DEL DNA
Le basi del DNA possono essere metilate
per azione della DNA metilasi con
formazione della 5-metilcitosina
1b : MODIFICAZIONI DEGLI ISTONI
Acetilazione e deacetilazione istoni
Fosforilazione e defosforilazione degli istoni
Metilazione e demetilazione degli istoni
Sono modificazioni chimiche che avvengono
preferenzialmente sugli amminoacidi LISINA e
SERINA e ARGININA
Influenzano la attività del DNA associato agli istoni
LE ESTREMITA’ N-TERMINALI DEGLI ISTONI HANNO
MOLTI SITI DI MODIFICAZIONE
LE MODIFICAZIONI DEGLI ISTONI CONTROLLANO L’ATTIVITA’ DEI GENI
L’acetilazione degli
istoni favorisce la
trascrizione
La metilazione degli istoni
reprime la attività dei geni
L’ESTREMITA N-TERMINALE DELL’ ISTONE H3
PU0’ TROVARSI IN STATI ALTERNATIVI
I siti bersaglio sono la
lisina in posizione 9 e la
serina in posizione 10
Cromatina
Attiva
Cromatina
Inattiva
2. REGOLAZIONE DELL’ ESPRESSIONE GENICA A LIVELLO DELLA
TRASCRIZIONE
2a regolazione della trascrizione svolta dai Fattori di Trascrizione Regolatori
può portare alla attivazione o alla repressione del gene
FATTORI DI TRASCRIZIONE REGOLATORI e FATTORI DI
TRASCRIZIONE BASALE
MOTIVI SRTUTTURALI DEI DIVERSI TIPI DI FATTORI DI
TRASCRIZIONE REGOLATORI
I fattori di trascrizione regolatori sono proteine
classificate in base alla struttura
Zinc Finger (struttura a dita di Zn)
Helix-turn-helix (Struttura a elicaripiegamento-elica)
Leucin zipper (struttura a cerniera di
leucina che facilita la formazione di dimeri)
Helix-loop-helix (struttura a elica-ansa-elica
che facilita la formazione di dimeri)
I FATTORI DI TRASCRIZIONE REGOLATORI SONO PROTEINE
CAPACI DI LEGARSI AL DNA
Hanno un dominio che può
legarsi alla doppia elica di
DNA
Hanno anche un dominio
capace di interagire con i
Fattori basali della
trascrizione o con la
RNApolII o con altri FT
(o con molecole effettrici
come gli ormoni)
FATTORI DI TRASCRIZIONE REGOLATORI CHE SI
LEGANO AGLI ENHANCER E AI SILENCER
Esercitano una azione continuativa sulla trascrizione
Gli attivatori si legano agli Enhancer
e i repressori si legano ai silencer
interferendo con l’azione degli
attivatori
In genere agiscono nella regolazione
tessuto-specifica
TFIID, B, A, F, E e H sono i fattori
basali della trascrizione
FATTORI DI TRASCRIZIONE REGOLATORI CHE SI LEGANO A
SPECIFICHE SEQUENZE DI REGOLAZIONE DEL PROMOTORE
Agiscono in modo transiente per il tempo necessario alla risposta della pianta
Esempio. alcuni fattori di trascrizione rispondono agli stimoli esterni come disidratazione e interni
come ABA
Struttura di un promotore a monte di un gene codificante una deidrina di girasole.
PROMOTORE DI UN GENE CHE CODIFICA UNA METALLOTIONEINA
Schematic illustration of the cold response network in Arabidopsis.
MYBRS = MYB recognition sequence
MYCRS= MYC recognition sequence
HOS1= ubiquitin E3 ligase
2. REGOLAZIONE DELL’ESPRESSIONE GENICA A LIVELLO DELLA
TRASCRIZIONE
2b SPLICING ALTERNATIVO
4. REGOLAZIONE DELL’ ESPRESSIONE GENICA A
LIVELLO POST TRADUZIONE
UBIQUITINAZIONE E DEGRADAZIONE DELLE PROTEINE
IL PROCESSO DI UBIQUITINAZIONE
Sono necessari 4 enzimi : E1, E2, E3 e E4
E1, Ubiquitin activating enzyme
E2, Ubiquitin conjugating enzyme
E3, Ubiquitin ligase
E4, Ubiquitin chain factor
DEGRADAZIONE DELLA PROTEINA UBIQUITINATA
I proteosomi sono grandi complessi proteici presenti in tutti gli eucarioti e in alcuni
batteri la cui funzione è quella di degradare le proteine non più necessarie o
danneggiate
3. REGOLAZIONE DELL’ESPRESSIONE GENICA A
LIVELLO POST-TRASCRIZIONALE
RNA INTERFERENCE
Esperimenti precedenti
Jorgensen e coll in Petunia (1990): trasformazione con un gene per la pigmentazione viola anziché
ottenere fiori viola ottennero fiori bianchi (o al max variegati).
Spiegarono il risultato con la CO-soppressione : sia il gene endogeno che il transgene venivano
soppressi
In Neurospora è stato osservato un fenomeno simile alla co-soppressione detto Quelling
L’RNA interference è stato scoperto in Caenorabditis elegans (1998) facendo esperimenti con RNA
antisenso per bloccare un certo gene. Videro che il gene veniva bloccato anche dall’RNA senso di
controllo.
Se iniettavano un RNA ibrido senso-antisenso ottenevano un forte silenziamento del gene endogeno
Studi successivi hanno studiato in dettaglio il Silenziamento genico post-trascrizionale mediato dai
piccoli RNA non codificanti. E’ stato scoperto
The small RNAs world
RNA SILENCING
FILMATO
C. C. Mello e A. Fire hanno vinto il Premio Nobel nel 2006 per la
scoperta dell’RNA Interference o silencing (1998 Nature)
La regolazione della trascrizione avviene anche degradando gli RNA già prodotti
E’ un meccanismo di regolazione dell’espressione genica Post-trascrizionale
Si basa sulla produzione di microRNA, mi RNA, e di short interfering RNA, i siRNA
piccoli RNA non codificanti complementari all’RNA trascritto da silenziare
I piccoli RNA non codificanti sono RNA di 21- 26 nucleotidi che sono stati identificati in
quasi tutti gli eucarioti , nelle piante identificati per la prima volta nel 2002
Nelle piante l’RNA silencing ha un ruolo di difesa dai virus patogeni ed è anche un
metodo per silenziare geni specifici
I micro RNA (miRNA) SONO CODIFICATI DA GENI DETTI GENI miRNA
Negli Animali
L’RNA trscritto dai geni miRNA forma una struttura
a doppio filamento imperfetto che viene processata
da un enzima detto DROSHA (RNAsi II) nel nucleo
Il pre miRNA viene riconosciuto dal DICER e
tagliato: si forma un miRNA a doppio filamento di
21bp che si lega ad un complesso di proteine , così si
forma il RISC (RNA Induced Silencing Complex)
Il complesso miRNA-RISC apre il doppio filamento
e uno solo dei due filamenti rimane con il RISC
Il miRNA-RISC forma un ibrido con l’RNA
messaggero nella regione 3’ UTR e ne inibisce la
traduzione
Oppure taglia l’RNA messaggero da silenziare
I bersagli dei mi RNA sono mRNA prodotti da vari
geni
I siRNA negli animali
siRNA
Si formano a partire
da lunghe molecole di
RNA a doppio
filamento di diversa
origine.
DICER
RISC,RISC-si RNA
che può tagliare gli
mRNA complementari
o bloccarne la
traduzione come i
miRNA.
Si ritiene che prima si sia evoluto il sistema dei siRNA per difesa dai virus
(e dai trasposoni) poi il sistema dei microRNA per regolare la espressione
genica di specifici geni
I microRNA DELLE PIANTE
Anche nelle piante sono
codificati dai geni miRNA
Si trascive un miRNA
precursore che forma una
struttura secondaria stemloop che poi viene
processata per ottenere il
miRNA maturo
( sequenza in grassetto)
In Arabidopsis sono stati trovati 117 geni miRNA
In vite 140 organizzati in 28 famiglie
I microRNA DELLE PIANTE
Distribuzione dei geni miRNA in diverse specie
miRNA families
Arabidopsis thaliana
46
Oryza sativa
46
Populus trichocarpa
33
Zea mays
18
Sorghum bicolor
16
Glycine max
11
Medicago truncatula
10
Saccharum officinarum
6
miRNA genes
117
178
213
97
72
22
16
16
Molti geni miRNA diversi
possono codificare lo stesso
miRNA maturo. Questi geni
son raggruppati in famiglie.
(Griffiths-Jones et al.,2006)
I miRNA sono coinvolti nei processi dello sviluppo della pianta,
nella organogenesi, nella risposta della pianta a stress ambientali
e stress da patogeni, nella risposta a ormoni .......
I microRNA DELLE PIANTE
Biogenesi e meccanismo di azione
•
I geni miRNA sono trascritti dalla RNApol II.
•
La struttura secondaria dell’RNA viene processata da
una proteina tipo DICER (DCL 1) in più passaggi
fino alla produzione di duplex miRNA-miRNA*
•
Il duplex miRNA-MiRNA passa nel citoplasma
•
Il duplex si apre e il miRNA siassocia al RISC. Si
forma il coplesso miRNA-RISC che taglia l’mRNA o
ne impedisce la traduzione.
I siRNA nelle PIANTE
• 1. I siRNA che derivano da RNA trascritti da transgeni o da virus patogeni
Gli RNA trascritti da virus o dai transgeni vengono resi a doppio filamento da
RDR1/6
RNA Dependent RNApolimerasi 1/6
I lunghi RNA a doppio filamento (in blu) sono tagliati in corti filamenti di 21 nucleotidi
(in rosso) da enzimi DICER- like .
•
•
a
b
VIA a: i piccoli si RNA sono caricati dal RISC
(associati con AGO4)
Il complesso si lega allo stesso RNA da cui si è
originato, lo taglia quindi lo silenzia.
I siRNA nelle PIANTE
• 2. I si RNA che derivano da Elementi Trasponibili e da Sequenze Eterocromatiche
Alcuni Autori li chiamano heterochromatic si RNA hc-siRNA
Biogenesi
dei hc-si RNA
DNA
RNApol IV
RISC
RNA
RDR
RISC
Bioge
nesi
dei
hc-si
RNA
Dicer-like
Elementi
Trasponibili
Dicer-like
I siRNA prodotti possono seguire due vie:
1° via
2° via
La via del silenziamento degli mRNA prodotti dai trasposoni stessi per bloccarli
La via del rimodellamento della cromatina attraverso:
la metilazione degli istoni H3K4 e H3K9 (Istone H3, lisina in posizione 9 e 4)
la metilazione del DNA , detta metilazione del DNA dipendente da RNA
RISC
1° via
Dicer-like
Elementi
Trasponibili
2° via
RISC
FINE