www.bioelearning.it - gruppo Metabolismo - Giovanni Aglialoro
Credits
animazioni tratte da:
* David Krogh “Biologia” Le Monnier - cd allegato
**
“Biologia della Cellula” cd di Federico Tibone e BioMEDIA Associates – Zanichelli ed.
libri consultati:
David Krogh “Biologia” Le Monnier
R. Torchio e altri “Biologia” – ed. Bulgarini
Cecie Starr “Biologia” – ed. Garzanti Scuola
Nelson - Cox “I principi di Biochimica di Lehninger” - ed. Zanichelli
[in rosso ho descritto i link, che fanno aprire le animazioni o verso altre pagine]
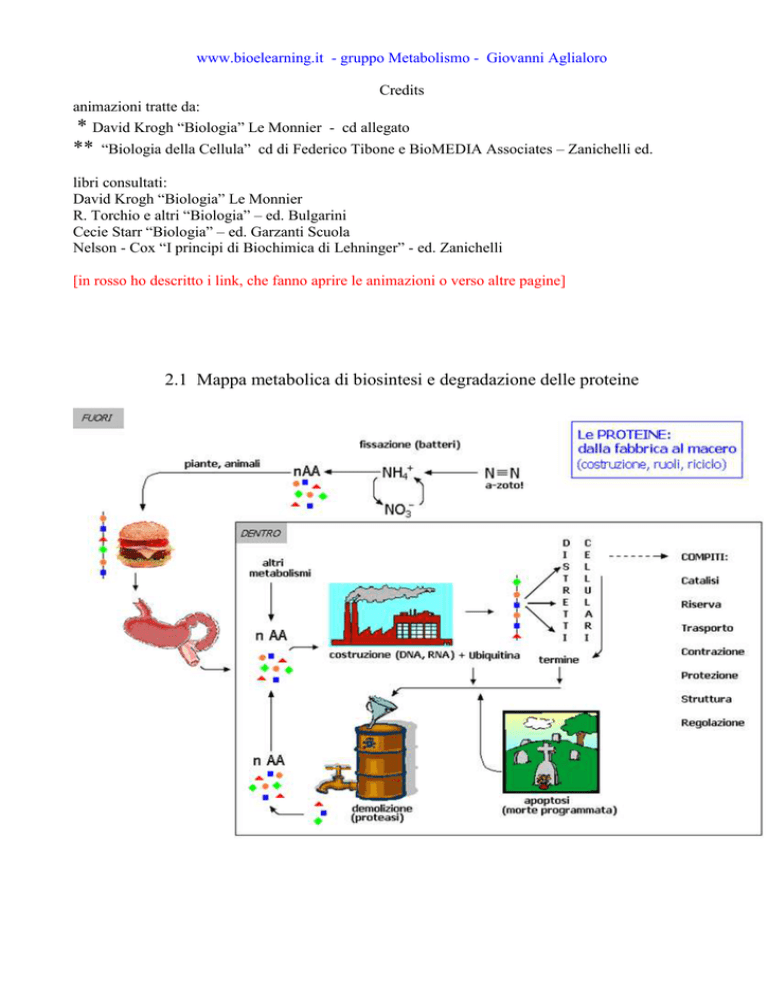
2.1 Mappa metabolica di biosintesi e degradazione delle proteine
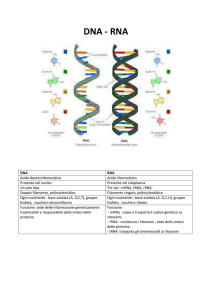
2.2 Biosintesi: DNA, mRNA, tRNA, AA, ribosomi
Tante proteine con solo 20 amminoacidi - Quando si legano una dopo l’altra più molecole di
amminoacidi si ottiene un polipeptide [animaz. polipeptide.swf ] che ripiegandosi su se stesso assume
una particolare conformazione tridimensionale; il risultato è una proteina.
La conformazione conferisce alla proteina la propria funzione specifica, in altre parole la capacità di
legarsi ad altre molecole.
RNA – Nella costruzione delle proteine sono coinvolte tre diverse classi di RNA, prodotte nella
trascrizione del DNA. Una, la più diffusa, è costituita da RNA messaggero, o mRNA, l’unico che porta
codificate le istruzioni per la sintesi delle proteine.
Un’altra classe è costituita da RNA ribosomiale, o rRNA, fondamentale componente dei ribosomi.
La terza classe, infine, è costituit da RNA di trasporto, o tRNA, che ha la funzione specifica di
consegnare ai ribosomi gli amminoacidi , uno per uno e nell’ordine specificato dall’mRNA.
La sintesi delle proteine [filmato sintesi_proteica.mov] – Come si può vedere dal filmato si tratta di un
processo biochimicamente complesso che, prima di venir esaminato più dettagliatamente, conviene
riassumere nel modo che segue.
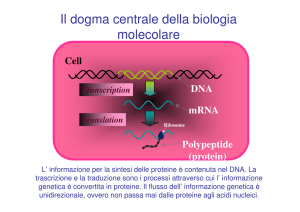
Un tratto del DNA contenuto nel nucleo della cellula si srotola separando i due filamenti; l’informazione
contenuta (ovvero la sequenza di basi azotate del filamento “stampo”) viene trascritta in una molecola di
mRNA. L’mRNA quindi fuoriesce dal nucleo della cellula verso i ribosomi ove viene letta la sequenza
che lo costituisce; man mano gli amminoacidi, presenti nel citosol e trasportati dal tRNA, vengono legati,
nell’ordine indicato dal mRNA, a formare la catena polipeptidica. Quando la costruzione è terminata essa
si stacca dal ribosoma e si ripiega su se stessa a formare la proteina. Nell’animazione seguente [animaz.
sintesi_proteica.swf] il processo viene suddiviso in due sequenze che tra poco esamineremo: trascrizione
(transcription, in nucleus) e traduzione (translation, in cytoplasm).
DNA trascrizione RNA traduzione Proteina
Innanzitutto per la sintesi di un polipeptide devono venir soddisfatte due condizioni: (1) il gruppo
carbossilico di ciascun amminoacido deve essere attivato per facilitare la formazione del legame peptidico
e (2) un legame si deve stabilire tra ciascun amminoacido e il codone corrispondente sull’mRNA.
Entrambi i requisiti vengono soddisfatti tramite l’attacco dell’amminoacido al tRNA corrispondente.
Durante questo stadio, che avviene nel citosol, non sui ribosomi, ciascuno dei 20 amminoacidi viene
legato covalentemente a un tRNA specifico utilizzando energia fornita dall’ATP.
Queste reazioni sono catalizzate da enzimi chiamati amminoacil-tRNA sintetasi.
Trascrizione - Questa fase permette la sintesi dell’mRNA; in altre parole, dal DNA (o meglio da una
regione particolare del filamento) l’informazione genetica passa all’ mRNA. La semielica di DNA che
agisce da stampo è denominata filamento stampo (o anti-senso), l’altra invece è il filamento non stampo
(o con senso). Meno del 10% del DNA umano è trascritto in mRNA e, a sua volta, solo una porzione di
quest’ultimo viene tradotta in proteine. L’mRNA neoformato (anche precursore o trascritto primario) è
costituito da porzioni dette esoni (codificanti) intervallate da altre, introni, non codificanti.
La trascrizione inizia in corrispondenza di siti specifici del DNA stampo, chiamati promotori, particolari
sequenze situate vicino al punto d’inizio della sintesi dell’mRNA. Alcune proteine (fattori di specificità,
repressori, attivatori) posizionano l’enzima RNA polimerasi sullo stampo di DNA e favoriscono il suo
legame con il promotore. L’animazione relativa ai geni regolatori spiega il meccanismo di repressione attivazione. [animaz. geni_regolatori.swf] L’enzima si sposta poi lungo il filamento di DNA
aggiungendo e unendo fra loro i vari nucleotidi. L’mRNA, una volta terminato, prima di uscire dal
nucleo viene escisso da enzimi specifici, ovvero vengono eliminate le porzioni di introni saldando tra loro
quelle costituite da esoni, in modo da ottenere quello che si chiama un mRNA maturo.
Traduzione – L’arrivo dell’mRNA ai ribosomi segna l’inizio del secondo stadio, ovvero la traduzione
[animaz. traduzione.swf] delle informazioni, codificate nella sequenza di basi dell’mRNA, in sequenza di
amminoacidi. I ribosomi sono organuli cellulari formati da due sub-unità composte da proteine e rRNA;
quest’ultimo permette la traduzione di ciascuna tripletta in un amminoacido.
Nel citoplasma esiste un pool di molecole di amminoacidi e tRNA liberi. Ogni molecola di tRNA ha un
sito di legame specifico per un amminoacido, oltre ad avere una tripletta di nucleotidi (anticodone, in blu
nella fig.) in grado di appaiarsi in modo complementare con un particolare codone dell’mRNA (in viola
nella fig.).
[figura tratta da http://www.gwu.edu/~darwin/BiSc150/One/codon.gif]
Queste triplette servono al posizionamento automatico dell’amminoacido, “agganciato” al tRNA, nella
sequenza corretta specificata dall’mRNA. Nel momento in cui inizia la sintesi il ribosoma è ancora
separato nelle sue due sub-unità. Quando alla minore si lega il tRNA d’inizio (start, che porta
l’amminoacido metionina) anche la sub-unità maggiore si chiude sull’mRNA. A questo punto un
secondo tRNA, con l’amminoacido corrispondente al secondo codone dell’mRNA, giunge al ribosoma. I
due amminoacidi si legano quindi fra loro (legame peptidico) e questo procedimento prosegue.
Una volta che il tRNA ha portato l’amminoacido e questo si è legato al precedente esso può
abbandonare il ribosoma ed essere impiegato per il trasporto di un altro amminoacido dello stesso tipo.
Questa traduzione procede fino a quando compare nell’mRNA un codone di terminazione (stop); esso
viene riconosciuto da molecole proteiche dette fattori di rilascio che determinano il distacco del tRNA
dalla catena polipeptidica ormai formata. Il ribosoma, ora, si separa nuovamente nelle due sub-unità in
attesa di una nuova sintesi. E’ importante ricordare che solitamente l’mRNA è letto da più ribosomi
contemporaneamente, che formano il cosiddetto polisoma. In tal modo si possono produrre più molecole
successive della stessa proteina. Impiegando sempre l’identica molecola di mRNA.
C’è da dire ancora che per acquisire la sua forma biologicamente attiva, il nuovo polipeptide deve
ripiegarsi nella sua conformazione tridimensionale. Prima o dopo il ripiegamento il polipeptide può
subire modificazioni enzimatiche , come rimozione di qualche amminoacido terminale o aggiunta di
gruppi (acetile, fosfato, metile, carbossile o altri).
Il codice genetico ovvero perché “triplette”? - Sappiamo che DNA e RNA “scrivono” mediante 4
“lettere”, cioè i nucleotidi, mentre gli amminoacidi che si trovano nelle proteine sono venti. Era quindi
impossibile che ogni nucleotide codificasse un singolo amminoacido, altrimenti il tutto si ridurrebbe a
quattro possibilità. Anche provando a ipotizzare un messaggio scritto con una sequenza di due nucleotidi
alla volta, il conto non torna in quanto 4x4 =16, con ancora quattro amminoacidi di troppo rispetto alle
“parole” per codificarli. Con sequenze di tre nucleotidi si hanno 4x4x4 =64 “parole”, numero ora
ridondante di “parole”, difatti nella realtà ci sono più triplette che codificano lo stesso amminoacido,
come si può vedere dalla tabella.
Il codice genetico.
La tabella riporta i 64 codoni (triplette di basi nel mRNA) e gli amminoacidi corrispondenti.
2a
base
U
C
A
G
3a
base
U
UUU fenilalanina
UUC fenilalanina
UUA leucina
UUG leucina, start
UCU serina
UCC serina
UCA serina
UCG serina
UAU tirosina
UAC tirosina
UAA stop
UAG stop
UGU cisteina
UGC cisteina
UGA stop
UGG triptofano
U
C
A
G
C
CUU leucina
CUC leucina
CUA leucina
CUG leucina, start
CCU prolina
CCC prolina
CCA prolina
CCG prolina
CAU istidina
CAC istidina
CAA glutammina
CAG glutammina
CGU arginina
CGC arginina
CGA arginina
CGG arginina
U
C
A
G
A
AUU isoleucina, start2
AUC isoleucina
AUA isoleucina
AUG metionina, start1
ACU treonina
ACC treonina
ACA treonina
ACG treonina
AAU asparagina
AAC asparagina
AAA lisina
AAG lisina
AGU serina
AGC serina
AGA arginina
AGG arginina
U
C
A
G
G
GUU valina
GUC valina
GUA valina
GUG valina, start2
GCU alanina
GCC alanina
GCA alanina
GCG alanina
GAU ac. aspartico
GAC ac. aspartico
GAA ac. glutammico
GAG ac. glutammico
GGU glicina
GGC glicina
GGA glicina
GGG glicina
U
C
A
G
1a
base
1
Il codone AUG serve per codificare la metionina ma anche come codone di avvio. Il primo
codone AUG lungo il filamento di mRNA è il punto in cui la traduzione ha inizio.
2
Questi sono codoni di avvio per i soli procarioti.
La traduzione inizia in corrispondenza di un codone di avvio (start) ma, a differenza del codone di
termine, questi non è sufficiente per avviare il processo di sintesi; in prossimità del codone di avvio
devono infatti anche trovarsi alcune sequenze tipiche che permettono all'mRNA di legarsi ai ribosomi.
Il codone di avvio più noto è AUG, che codifica anche la metionina. Altri codoni di avvio sono CUG,
UUG e, nei procarioti, GUG e AUU.
Molti codoni sono ridondanti, cioè due o più codoni corrispondono allo stesso amminoacido. Questa
ridondanza è limitata alla terza base del codone - ad esempio, sia GAA che GAG codificano la
glutammina. Un codone è detto quattro volte degenere se qualsiasi nucleotide nella sua terza posizione
codifica lo stesso amminoacido (ad esempio, UCA, UCC, UCG e UCU, tutti corrispondenti alla serina); è
detto due volte degenere se solo due delle quattro basi nella sua terza posizione codificano lo stesso
amminoacido (ad esempio AAA ed AAG, corrispondenti alla lisina). Nei codoni due volte degeneri, i
nucleotidi equivalenti nella terza posizione sono sempre o due purine (A/G) o due pirimidine (C/T).
La ridondanza rende il codice genetico meno vulnerabile alle mutazioni causali. Un codone quattro volte
ridondante può subire qualsiasi mutazione alla sua terza posizione ed un codone due volte ridondante può
subire una delle tre possibili mutazioni alla sua terza posizione senza che l'amminoacido da esso espresso
- e quindi la struttura della proteina in cui l'amminoacido verrà inserito - cambi. Inoltre, dato che le
mutazioni per transizione (da una purina all'altra o da una pirimidina all'altra) sono più probabili delle
mutazioni per transversione (da purina a pirimidina o viceversa), l'equivalenza tra purine o tra pirimidine
nei codoni due volte degeneri aggiunge un'ulteriore resistenza. Sono solo due gli amminoacidi codificati
da un unico codone: metionina, codificata da AUG (che è anche codone di avvio) e triptofano, codificato
da UGG.
Diverse variazioni al codice genetico standard sono state trovate nei mitocondri. Anche i protozoi ciliati
presentano qualche modifica: in loro (come anche in alcune specie di alga verde) UAG e, spesso, UAA
codificano la glutammina e UGA codifica la cisteina. In alcune specie di lievito CUG codifica la serina.
In altre specie di batteri ed archeobatteri i codoni di arresto codificano invece amminoacidi non comuni:
UGA codifica la selenocisteina e UAG la pirolisina. È possibile che vi siano altri amminoacidi nonstandard la cui codifica è ancora ignota. A dispetto di queste variazioni, i codici genetici usati da tutte le
forme di vita della Terra sono molto simili. Dato che i codici genetici possibili e potenzialmente adatti
alla vita sono molti, la teoria dell'evoluzione fa pensare che questo codice genetico sia andato a definirisi
molto presto nella storia della vita su questo pianeta.
Esempio di trascrizione - traduzione di triplette: dal DNA all’amminoacido
DNA (filam. non stampo)
DNA (filam. stampo)
mRNA
amminoacido
TTG
AAC
UUG
TTA
AAT
UUA
TGT
ACA
UGU
AGC
TCG
AGC
ACT
TGA
ACU
leu
leu
cys
ser
thr
Volendo convertire triplette di DNA in amminoacidi si può visitare:
http://www.geneseo.edu/~eshamb/php/dna.php
o, ancora, per interagire con il Codice Genetico:
http://web.rhul.ac.uk/biological-sciences/Archives/Warren/gencode/