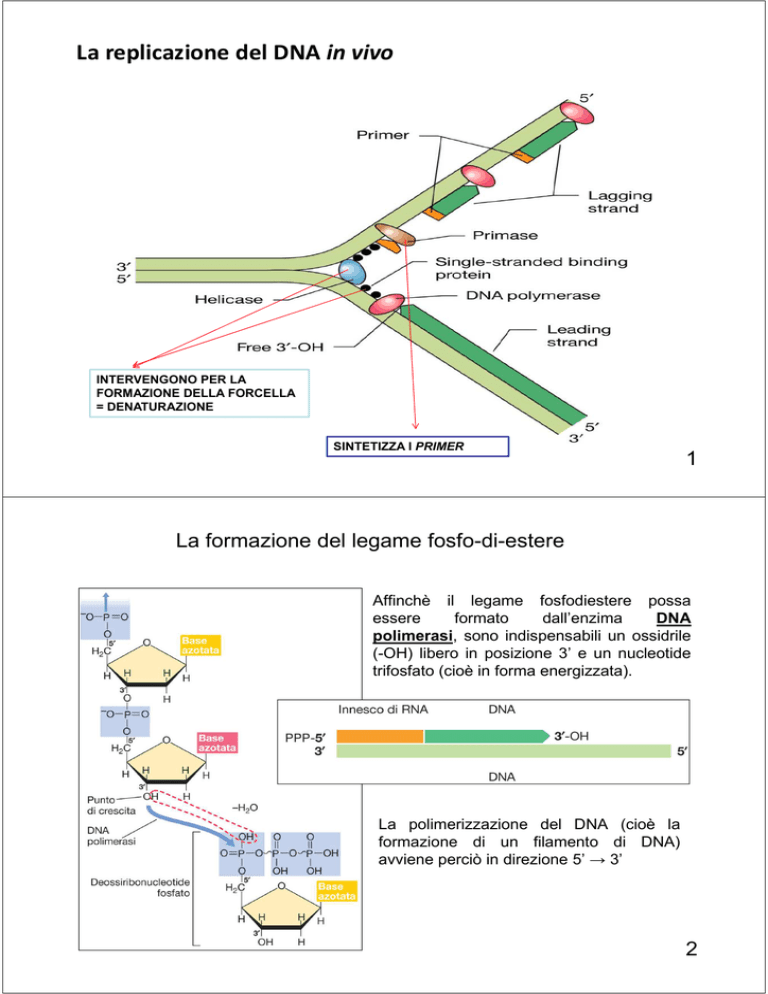
La replicazione del DNA in vivo
INTERVENGONO PER LA
FORMAZIONE DELLA FORCELLA
= DENATURAZIONE
SINTETIZZA I PRIMER
1
La formazione del legame fosfo-di-estere
Affinchè il legame fosfodiestere possa
essere
formato
dall’enzima
DNA
polimerasi, sono indispensabili un ossidrile
(-OH) libero in posizione 3’ e un nucleotide
trifosfato (cioè in forma energizzata).
La polimerizzazione del DNA (cioè la
formazione di un filamento di DNA)
avviene perciò in direzione 5’ → 3’
2
LA REAZIONE A CATENA
DELLA POLIMERASI
- Kary Mullis ideò e mise a punto la PCR (Polymerase Chain
Reaction) tra il 1983 ed il 1985
- Riproduce in vitro la reazione di replicazione del DNA
(per chi non si ricordasse come avviene la replicazione del DNA in una cellula, si
consiglia di ripassare l’argomento. C’è del materiale sul tema in una precedente
lezione).
- Permette di replicare (riprodurre) indefinitamente una specifica
regione di DNA
- Affinché la PCR avvenga sono necessari:
▪ il DNA templato (stampo);
▪ gli oligonucleotidi iniziatori (primer);
▪ desossinucleotidi trifosfato (dNTP);
▪ la DNA polimerasi
Ve ne sono di diversi tipi (x es. Taq polimerasi)
▪ un ciclizzatore termico (PCR termocycler)
3
Taq polimerasi
È l’enzima DNA-polimerasi utilizzato per le reazioni di PCR
Si chiama in questo modo perché è stato originariamente isolato dal
batterio termofilo Thermus aquaticus
4
IL TERMOCICLATORE (detto anche
ciclatore termico) per PCR
5
LE TRE FASI DELLA PCR
DNA (cromosomale, plasmidico, etc…)
A
B
1. DENATURAZIONE
(94 – 96 °C per 30 s / 1 min)
A
B
2. APPAIAMENTO (tra 45 e 65 °C circa per 30 s / 1 min)
(annealing)
A
B
3. ESTENSIONE
(extension)
(72 °C per 10 s / 10 min o più)
A
B’
A’
B
6
La costruzione dei primers
5’ 3’ -
- 3’
- 5’
Primer per amplificazione specifica (solitamente di lunghezza da 18 a 22 mer)
Primer FORWARD: 5’- AAGATCTTCTTACTCAGT - 3’
Primer REVERSE: 5’- CATGGACCGGTACAGTAGGT- 3’
7
I CICLI DELLA PCR
- Da una singola molecola di templato, sono prodotti circa 1
miliardo di frammenti copia di DNA in 30 cicli:
N.B.: con la comune Taq polimerasi si riescono ad amplificare
fino a circa 3000 – 3500 bp; con Ext-Taq fino anche a 40000 bp!
(Animazione: http://www.sumanasinc.com/webcontent/anisamples/molecularbiology/pcr.html)
8
LE APPLICAZIONI DELLA PCR
- Nelle biotecnologie: semplificazione del processo di clonaggio
di geni singoli per le più dettagliate analisi biochimiche
- In medicina: per una rapida diagnosi di infezioni virali o per la
diagnosi prenatale di malattie congenite
- Permette di raccogliere informazioni da campioni biologici
contenenti anche scarsissime tracce di DNA importanti
applicazioni in campo giuridico o in archeologia, paleontologia
e antropologia (x es. per lo studio delle migrazioni umane)
- In microbiologia: di fondamentale importanza per la
tassonomia e la tipizzazione molecolare di isolati microbici
9
RICAPITOLANDO:
10
Verifica dell’avvenuta amplificazione:
• gel elettroforesi
• controllo negativo
C.N.
MARKER di
frammenti di DNA
lineare a peso
molecolare noto
11
La tassonomia e l’identificazione batterica
La tassonomia si occupa della classificazione, cioè della
IDENTIFICAZIONE e della NOMENCLATURA degli organismi viventi.
L’unità tassonomica di base è la SPECIE, che è definibile nell’ambito delle
batteriologia come una collezione di ceppi simili che differiscono sufficientemente
da altri gruppi di ceppi da giustificare il riconoscimento come unità tassonomica
a se stante (si veda in seguito una definizione ‘’più concreta’’ di specie batterica).
Ogni specie batterica possiede un CEPPO TYPE, cioè un ceppo di riferimento che
riassume in se tutte le caratteristiche fenotipiche e genotipiche dei ceppi della specie
che rappresenta. Dove posso trovare il ceppo type di ogni specie? I ceppi type si
trovano nelle «ceppoteche», cioè nelle collezioni di colture cellulari. Le collezioni di
colture di riferimento, in Europa e negli Stati Uniti, sono le seguenti:
Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH (DSMZ),
(German Collection of Microorganisms and Cell Cultures), organizzazione no-profit
(per es.: Bifidobacterium bifidum DSM 20456T)
N.B.: SONO LO
STESSO CEPPO!
American Type Culture Collection (ATCC), centro di ricerca privato no-profit
(per es.: Bifidobacterium bifidum ATCC 11863T)
12
Come si identifica un batterio
L’identificazione tassonomica di un ceppo batterico si basa sulla
valutazione di CARATTERISTICHE FENOTIPICHE e GENOTIPICHE e
sul confronto di queste caratteristiche con quelle dei ceppi type.
CARATTERISTICHE
FENOTIPICHE
1. Morfologiche
2. Fisiologiche
3. Colturali
4. Nutritive
5. Biochimiche
6. Bio-strutturali
GENOTIPICHE
Analisi degli acidi nucleici
(in particolare della loro
sequenza nucleotidica)
Lo studio delle caratteristiche fenotipiche per l’identificazione di un batterio è la via
tradizionale seguita da decenni da quella che è definita MICROBIOLOGIA CLASSICA.
Ad essa si è ora aggiunta (e in molti casi sostituita) la microbiologia basata sulle
tecniche di BIOLOGIA MOLECOLARE, che prevede lo studio di DNA e RNA.
13
La caratterizzazione fenotipica
Le prime caratteristiche da considerare sono le più generali, al fine di ridurre subito il
numero delle possibilità tra cui ricercare…
A. CARATTERISTICHE MORFOLOGICHE
1. micromorfologia: forma, dimensione, disposizione delle cellule (catenelli,
aggregati), presenza di endospore;
2. macromorfologia: aspetto della colonia;
3. reazione di Gram
4. mobilità
B. METABOLISMO ENERGETICO
Opera respirazione aerobia, oppure fermentazione; è anaerobio facoltativo…
C. CARATTERISTICHE COLTURALI E NUTRITIVE
Primariamente, viene considerata la TIPOLOGIA DI FONTE DI CARBONIO: organica
o inorganica? Quali molecole possono essere usate? È importante ottenere un profilo
di utilizzazione del carbonio. A tal fine, si prepara un TERRENO COLTURALE DI
BASE, cioè un terreno in cui è stata completamente tolta la fonte dell’elemento in
esame (in questo caso il carbonio). Trasferisco questo terreno di coltura in alcune
provette e in ciascuna aggiungo una fonte di carbonio diversa (per esempio glucosio,
ribosio, mannosio, etc…). Quindi inoculo ogni provetta con lo stesso numero di cellule
del ceppo in esame e metto ad incubare. Se il batterio cresce significa che ha saputo
usare quella specifica fonte di carbonio.
14
N.B.: se, ad esempio, sto studiando un batterio lattico, posso valutare la capacità di
utilizzare la fonte di carbonio da parte del ceppo in esame aggiungendo al terreno di
coltura un indicatore di pH. Se il ceppo utilizza la fonte di carbonio produce acido e
quindi il colore dell’indicatore di pH cambia.
N.B.: esistono in commercio kit per poter testare rapidamente numerose fonti di
carbonio in contemporanea, come ad esempio il sistema di gallerie API (Analytical
Profile Index). Nello specifico, le gallerie API consistono in una serie di microtubi
contenenti substrati disidratati x evidenziare attività enzimatiche o fermentazioni di
zuccheri; i microtubi vengono inoculati con una sospensione batterica che
ricostituisce i substrati. Le reazioni prodotte durante il periodo di incubazione si
traducono in reazioni colorate immediate o rilevati dall’aggiunta di reattivi.
Esempio di uso del kit API 50 CHL
= il batterio non utilizza il substrato
= il batterio è in grado di utilizzare il substrato
Controllo
negativo
Ribosio
http://aulavirtual.usal.es/aulavirtual/demos/microbiologia/unidades/LabV/API/api.html
15
C. CARATTERISTICHE COLTURALI E NUTRITIVE
Oltre alla tipologia di fonte di carbonio possono essere studiate: la fonte di azoto, i
fattori di crescita (vitamine), i parametri ambientali (pH, temperatura).
D. CARATTERISTICHE FISIOLOGICHE E BIOCHIMICE
Tipicamente, si studia il pattern enzimatico; per es.:
1. catalasi;
2. amilasi;
3. proteasi;
4. lipasi;
5. ureasi.
E. CARATTERISTICHE CHIMICO-STRUTTURALI
Tipicamente, si studiano:
1. la composizione chimica della parete cellulare;
2. gli acidi grassi nella membrana cellulare
16
Identificazione preliminare di un ceppo batterico attraverso
colorazione di Gram e morfologia cellulare
17
Esempio di identificazione preliminare di bastoncini Gram positivi
Resistenza alla
perdita di colore
con acidi dopo
colorazione
18
C. CARATTERISTICHE COLTURALI E NUTRITIVE
Oltre alla tipologia di fonte di carbonio possono essere studiate: la fonte di azoto, i
fattori di crescita (vitamine), i parametri ambientali (pH, temperatura).
D. CARATTERISTICHE FISIOLOGICHE E BIOCHIMICE
Tipicamente, si studia il pattern enzimatico; per es.:
1. catalasi;
2. amilasi;
3. proteasi;
4. lipasi;
5. Ureasi.
E. CARATTERISTICHE CHIMICO-STRUTTURALI
Tipicamente, si studiano:
1. la composizione chimica della parete cellulare;
2. gli acidi grassi nella membrana cellulare
A questo punto si avrebbero già molte informazioni per determinare la
specie del ceppo batterico allo studio; spesso, però, non si riesce a
ottenere un risultato definitivo (non si riesce ad avere la certezza); si
passa allora allo studio del GENOTIPO
lo studio del genotipo è l’unico
che mi può dare la certezza in merito all’identificazione tassonomica di
specie di un ceppo batterico.
19
Bergey's manual of systematic bacteriology
“MANUALE DI BERGEY”: è il punto di
riferimento per la tassonomia dei
procarioti
Contiene la lista completa delle specie
procariotiche e la loro classificazione e
nomenclatura
20
La caratterizzazione genotipica
Il concetto di specie batterica
- Negli organismi superiori le SPECIE sono definite come gruppi di individui in grado
di accoppiarsi in natura o potenzialmente in grado di farlo (ovvero interfecondi, cioè
individui che si possono accoppiare e dare origine a prole fertile), e che sono ben
distinti dagli altri gruppi per quanto concerne l’attività riproduttiva
NON
APPLICABILE AI BATTERI
Nei batteri la SPECIE è definita come un insieme di ceppi isolati
in habitat diversi ed in tempi diversi che posseggono (queste
sono REGOLE PRATICHE!):
• una elevata similarità fenotipica, con almeno una proprietà
distintiva nei confronti delle altre specie
• una elevata omologia genetica (riassociazione molecolare
DNA/DNA >70%)
• una elevata omologia filogenetica (omologia di sequenza del
gene codificante per il 16S rRNA maggiore del 98%)
21
DA RICORDARE!
La riassociazione molecolare DNA/DNA (detta anche omologia DNA/DNA o
ibridazione DNA/DNA) è una prova di laboratorio che prevede i seguenti passaggi:
1. Isolamento del DNA totale del ceppo batterico allo studio;
2. Isolamento del DNA totale estratto da un ceppo di riferimento (solitamente il
ceppo type di una specie);
3. Rottura meccanica dei due DNA
4. Miscela dei due DNA
5. Denaturazione termica del campione con misura della densità ottica (a 260 nm)
6. Lento raffreddamento del campione con misura della densità ottica (in questa fase
avvien l’ibridazione, cioè il DNA dei due campioni si riassociano fra di loro)
curva di
ipercromicità
OD260
% riassociazione
La percentuale di omologia
si stabilisce analizzando la
curva di riassociazione (detta
di ibridazione) che avviene
durante il raffreddamento,
fase in cui i DNA dei due
ceppi riassociano fra di loro.
Tale curva varia varia a
seconda di quanto le
sequenze di DNA sono simili
tra loro
nm
OD Tr=0.
OD iniz.
RISCALDAMENTO
RAFFREDDAMENTO
h
22
Al fine di identificare un ceppo batterico a livello di specie si utilizzano geni conservati
(cioè presenti in tutti) dei procarioti che possono essere usati come orologi molecolari
in grado di esprimere la filogenesi dei batteri, il più noto di questi geni è il:
gene codificanti per la subunità ribosomale 16S (16S rRNA)
Esso è un gene appartenente all’operone ribosomale batterico
(~1,5 kb)
5’ -
(tRNA)
16S
23S
5S
- 3’
regione spaziatrice intergenica (Internally Transcribed Spacer)
I geni 16S, 23S e 5S rRNA codificano per le subunità di RNA costituenti i ribosomi
batterici; l’operone ribosomale è posseduto da tutti gli organismi viventi:
Type
Size
Subunità più grande (rRNA)
Subunità piccola (rRNA)
PROCARIOTA
70S
5S (120 nt), 23S (2906 nt)
16S (1542 nt)
EUCARIOTA
80S
5S (121 nt), 5.8S (156 nt), 28S (5070 nt)
18S (1869 nt)
Il gene che codifica per il 16s rRNA è una regione ALTAMENTE CONSERVATA tra
le differenti specie di procarioti (cioè subisce mutazioni di sequenza molto lentamente
nel corso dell’evoluzione, poiché è di importanza primaria per la vita della cellula).
È considerato un ottimale “MOLECULAR CLOCK”, un OROLOGIO MOLECOLARE,
cioè è adatto per stabilire le distanze filogenetiche (evolutive) tra i diversi gruppi
tassonomici batterici.
23
24
25
N.B.: diverse regioni del DNA possiedono una diversa predisposizione ad “accettare”
le mutazioni… così, per avere un elevato potere discriminante, utile a
distinguere tra due organismi strettamente correlati tra loro, sceglierò regioni a
più alta variabilità (meno conservate)
Il gene 16S rRNA nell’identificazione batterica
La determinazione della sequenza del gene 16S rRNA è diventata molto
diffusa in microbiologia come un’alternativa rapida ed accurata ai metodi di
identificazione batterica basati sul fenotipo
Oltre a regioni estremamente conservate, il gene 16S rRNA contiene regioni
IPERVARIABILI che possono rappresentare delle sequenze di
riconoscimento di tipo specie specifico, utili per l’identificazione dei batteri
L’analisi della sequenza del gene 16S rRNA, quindi, può fornire
informazioni per diversi livelli tassonomici, fino al livello di specie, e
permette di tracciare relazioni filogenetiche tra i microrganismi
Sulla base delle sequenze note del gene 16S rRNA, sono stati disegnati degli
oligonucleotidi definiti primer universali, che permettono di amplificare via PCR
questo gene virtualmente da tutti i batteri
26
27
Il sequenziamento del gene 16SrDNA
Il sequenziamento viene generalmente condotto da tecnici specializzati e fornito
sotto forma di cromatogramma. Una sequenza parziale delle prime 500 bp del
gene, può essere sufficiente per determinare la specie di appartenenza del ceppo
allo studio, tramite comparazione in banca dati con le sequenze note riportate
(l’intero gene 16S rRNA è lungo circa 1500 bp). Però, talvolta, per una
identificazione più sicura, è necessario sequenziare l’intero gene.
Ricordiamo che due ceppi appartenenti alla stessa specie devono possedere una
% omologia 16S rDNA >97-98%
28
Lo studio della filogenesi batterica
Confrontando la sequenza del gene 16S rRNA dei diversi batteri è possibile
quantificare la distanza filogenetica, cioè, determinare a che punto dell’evoluzione due
specie batteriche si sono differenziate
Algoritmi matematici allineano le sequenze dei geni 16S rRNA e ne determinano il
grado di similarità, che viene espresso attraverso un DENDROGRAMMA DI
SIMILARITÀ o ALBERO FILOGENETICO
29
30
Albero filogenetico delle divisioni batteriche basato sull’analisi delle sequenze
del 16S rRNA della maggior parte delle specie batteriche note
Gram +
Firmicutes
Bifidobatteri
Batteri Lattici e Batteri Sporigeni
Lactobacillus
Streptococcus
Lactococcus
Pediococcus
Leuconostoc
Oenococcus
Bacillus
Clostridium
Gram +
Batteri Enterici o Enterobatteri
Escherichia
Salmonella
Shigella
Yersinia
Enterobacter
Gram -
31
I gruppi tassonomici
La tassonomia batterica è in continuo aggiornamento
per conoscere a la
filogenesi di un qualsiasi gruppo tassonomico e per ottenere informazioni sulla
tassonomia aggiornata e valida, si può fare riferimento alla National Center for
Biotechnology Information (NCBI): http://www.ncbi.nlm.nih.gov/taxonomy
Vale per tutti gli organismi viventi, non solo per i batteri!
È sufficiente scrivere il nome di un qualsiasi gruppo tassonomico per conoscere la
sua filogenesi aggiornata. Per esempio, se volessimo sapere di più della famiglia
Enterobacteriaceae:
32
Un altro interessante sito per ottenere informazioni su un qualsiasi gruppo
microbico è MicrobWiki: https://microbewiki.kenyon.edu/index.php/MicrobeWiki
33
33
FINE DEL CORSO
34



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)





![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)


