Chimica Biologica
A.A. 2010-2011
Fosforilazione
ossidativa
Marco Nardini
Dipartimento di Scienze Biomolecolari e Biotecnologie
Università di Milano
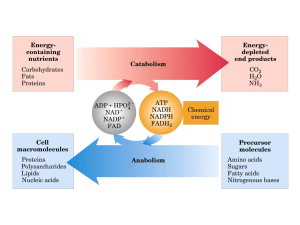
Macromolecole Biologiche
Fosforilazione ossidativa
Teoria Chemiosmotica
ΔG = 2.3 RT (pH(matrice) - pH(spazio itermembr)) + ZFΔψ
- se
H+ è trasportato da una regione negativa ad una positiva Δψ >0
⇒ il trasporto di H+ dalla matrice allo spazio intermembrana (contro il
gradiente protonico) è un processo endoergonico
⇒ lo scarico del gradiente protonico è esoergonico
Fosforilazione ossidativa
Macromolecole Biologiche
Teoria Chemiosmotica
NADH + H+ + ½O2 → NAD+ + H2O
ΔG’°= -220 kJ/mol
ΔG = RT ln(C2/C1) + zF ΔΨ
ΔG = 2.3RT ΔpH + F ΔΨ
Forza proton-motrice
2 componenti:
differenza di [H+]
differenza di potenziale elettrico
ΔΨ = 0.15-0.2 V (potenziale di membrana)
ΔpH = 0.75
ΔG = 20 kJ/mol (H+)
10H+N → 10 H+P
ΔG = +200 kJ/mol
⇒ l’energia libera dell’ossidazione del NADH da parte dell’O2 viene
efficientemente convertita in gradiente elettrochimico di H+
Fosforilazione ossidativa
Macromolecole Biologiche
Teoria Chemiosmotica
- in condizioni fisiologiche l’energia libera per la sintesi di un ATP è
~40-50 kJ/mole
⇒ almeno 2 H+ devono tornare indietro nella matrice mitocondriale
per generare tale energia libera
- l’ATP sintasi utilizza
tale energia libera per la
fosforilazione dell’ADP
Fosforilazione ossidativa
Macromolecole Biologiche
Teoria Chemiosmotica
- per ogni coppia di e- trasportata dal NADH all’O2 attraverso la catena
respiratoria sono traslocati 10 H+ dal lato N a quello P della membrana
interna
⇒ il ritorno di 10 H+ dal lato P a quello N rende disponibile ~200 kJ di energia libera
ADP + Pi + nH+P →ATP + nH+N
Quindi l’accoppiamento tra la reazione:
NADH + H+ + ½O2 → NAD+ + H2O
e la reazione:
ADP + Pi →ATP
è mediato dal trasporto vettoriale di H+ nelle due direzioni attraverso la
membrana interna rispettivamente contro e a favore del gradiente
elettrochimico
Fosforilazione ossidativa
Macromolecole Biologiche
ATP sintasi
“pompa protonica” o “F1F0-ATPasi” (complesso V)
F0: proteina transmembrana ⇒ fino a 8 tipi di subunità
F1: proteina periferica di membrana (solubile) ⇒ 5 tipi di subunità
- F1 solubilizzato idrolizza ATP ma non lo sintetizza (⇒ ATP-asi)
- F1 proiettato all’interno della matrice
F1
stelo ~50 Å
F0
Fosforilazione ossidativa
Macromolecole Biologiche
Componente F1
subunità α3β3γδε
α
β
γ
subunità α e β:
- 20% identiche in aa
- α e β stesso fold ed
arrangiamento alternato
- solo β catalizza la sintesi di ATP
20 Å
subunità γ:
- elica C-terminale di 90 Å
F1-ATP sintasi da mitocondri di cuore bovino
- stelo ~30 Å (coil-coil antiparallelo eliche N-term e C-term)
Fosforilazione ossidativa
Macromolecole Biologiche
Componente F0
subunità a1b2c9-12 (E. coli)
Subunità ε
subunità c:
2 eliche che formano 2 anelli
concentrici transmembrana
subunità a:
5 eliche transmembrana
subunità b:
- singola elica transmembrana
- dominio polare (dimero) che contatta
le subunità δα3β3 di F1
Subunità c
Asp 61
deprotonato
Asp 61
protonato
Macromolecole Biologiche
Fosforilazione ossidativa
Complesso F1-c10 da lievito
Macromolecole Biologiche
Fosforilazione ossidativa
ATP sintasi: meccanismo del cambiamento di
legame
- sintesi di ATP mediante translocazione di protoni (dallo spazio
intermembrana alla matrice mitocondriale) da parte dell’ATP sintasi
3 tappe:
1) translocazione di protoni promossa da F0
2) formazione catalitica del legame fosfoanidridico dell’ATP da parte di F1
3) accoppiamento della dissipazione del gradiente protonico con la sintesi
di ATP, che necessita l’interazione di F1 con F0
Meccanismo del cambiamento di legame:
- F1 possiede 3 protomeri catalitici (subunità αβ) interagenti fra loro ma
ognuno dei quali con un diverso stato conformazionale:
stato L (loose), stato T (tight), stato O (open)
Macromolecole Biologiche
Fosforilazione ossidativa
ATP sintasi: meccanismo del cambiamento
di legame
stato L: substrati e prodotti si legano debolmente alla subunità αβ di F1
stato T: substrati e prodotti si legano saldamente
stato O: substrati e prodotti non si legano
- l’ATP viene sintetizzato solo nello stato T
- l’ATP viene rilasciato solo nello stato O
- l’energia libera rilasciata durante la translocazione dei protoni viene
utilizzata per interconvertire questi 3 stati conformazionali
Macromolecole Biologiche
Fosforilazione ossidativa
ATP sintasi: meccanismo del cambiamento
di legame
1) ADP e Pi si legano debolmente alla subunità ab di F1 nello stato L
2) - conversione del sito L in sito di legame T dovuto ad un cambio
conformazionale promosso dal rilascio di energia libera
- concomitante conversione del sito T (contenente ATP legato) a sito O
e sito O in sito L
3) sintesi di ATP nel sito T, dissociazione di ATP dal sito O
L’energia libera fornita dal flusso di protoni facilita principalmente il
rilascio dell’ATP sintetizzato dall’enzima, cioè la transizione T→O
Macromolecole Biologiche
Fosforilazione ossidativa
Meccanismo rotatorio
“cambiamenti di legame” promossi dalla rotazione relativa fra α3β3 e le
altre parti della F1F0-ATPasi
in bianco
residui
apolari
subunità γ
⇒ libera rotazione di γ in α3β3 fisso (dovuta ad assenza di interazioni polari
o di legame a H fra γ e α3β3 nel poro)
⇒ variazioni in α3β3 correlate alla posizione di γ (movimento ad “albero
a camme”)
Fosforilazione ossidativa
Macromolecole Biologiche
Meccanismo rotatorio
statore: α3β3, l’unità ab2 (e δ per E.coli)
rotore: anello di subunità c, subunità γ (e ε per E.coli)
modello di F1FO-ATPasi
da E.coli
in E.coli: spazio periplasmico
nel mitocondrio: spazio intermembrana
in E.coli: citoplasma
nel mitocondrio: matrice
Fosforilazione ossidativa
Macromolecole Biologiche
Meccanismo rotatorio
- i protoni si legano probabilmente ad Asp 61 di una subunità c
- alterazione conformazione subunità c ⇒ spinta contro subunità a ⇒ rotazione
- i protoni entrano in un canale
idrofilico tra la subunità a e l’anello c
e si legano ad una subunità c
- vengono rilasciati all’interno della
matrice attraverso un secondo canale,
dopo che l’anello c ha eseguito quasi
un giro completo
- F1F0-ATPasi genera 3 ATP per giro
Asp 61
deprotonato
Asp 61
protonato
in lievito (10 subunità c) ⇒ 0.3 ATP per ogni H+ che passa dall’esterno
all’interno della membrana interna
Fosforilazione ossidativa
Macromolecole Biologiche
Meccanismo rotatorio
evidenza diretta
- filamento di actina biotinilato e resa fluorescente
- streptavidina che lega fortemente la biotina (4 siti di legame)
in E.coli la F1F0 -ATPasi può agire anche in modo inverso
esperimento di idrolisi di ATP (5 mM MgATP)
Glu 2Glu
→ Cys
2 → Cys
con microscopio a fluorescenza
- rotazione actina in senso antiorario
- la subunità γ ruota per incrementi di ~120°
Fosforilazione ossidativa
Macromolecole Biologiche
Rapporto P/O
relazione stoichiometrica fra sintesi di ATP (P) e quantità di
ossigeno ridotto (O), cioè il numero di moli di ATP per atomo
di ossigeno ridotto, ovvero per coppia di elettroni trasportata
- la catena di trasporto degli elettroni genera il gradiente protonico ai capi
della membrana mitocondriale interna
- lo scarico del gradiente protonico è accoppiato con la sintesi di ATP
⇒ è possibile esprimere la quantità di ATP sintetizzato in termini di
molecole di substrato ossidate
esperimenti su mitocondri isolati:
ossidazione di una molecola di NADH ⇒ sintesi di ~ 3 ATP
ossidazione di una molecola di FADH2 ⇒ sintesi di ~ 2 ATP
ossidazione di una molecola di TMPD ⇒ sintesi di ~ 1 ATP
TMPD (tetrametil-p-fenilenediamina):
dona 2e- dire direttamente al Complesso IV
forma ossidata
Macromolecole Biologiche
Fosforilazione ossidativa
Rapporto P/O e teoria chemiosmotica
i rapporti P/O determinati sperimentalmente sono compatibili con la
teoria chemiosmotica
- il flusso di 2 elettroni attraverso i Complessi I, III, e IV risulta nella
translocazione di 10 H+ nello spazio intermembrana
- l’influsso di 10 H+ attraverso la F1F0-ATPasi (10 subunità c negli
eucarioti) porta ad una completa rotazione del rotore formato dall’anello
c e dalla subunità γ rispetto allo sferoide α3β3 ⇒ sintesi di ~ 3 ATP
Macromolecole Biologiche
Fosforilazione ossidativa
Rapporto P/O e teoria chemiosmotica
- il flusso di 2 elettroni attraverso il Complesso II (bypass del Complesso I)
risulta nella translocazione di 6 H+ nello spazio intermembrana
⇒ rotazione di 2/3 del rotore di F1F0-ATPasi ⇒ sintesi di ~ 2 ATP
- il flusso di 2 elettroni attraverso il Complesso IV risulta nella translocazione di
2 H+ nello spazio intermembrana
⇒ rotazione di 1/3 del rotore di F1F0-ATPasi ⇒ sintesi di ~ 1 ATP
Macromolecole Biologiche
Fosforilazione ossidativa
Rapporto P/O e teoria chemiosmotica
- il gradiente protonico può essere parzialmente dissipato dal
(1) ritorno non specifico di H+ nella matrice
(2) consumo di H+ per altri scopi
es: trasporto di Pi nella matrice dal citosol attraverso
simporto Pi-H+ ⇒
⇒ 4 protoni consumati per la sintesi di un ATP
rapporti P/O ~2.5, ~ 1.5, ~ 1
Respirazione
Macromolecole Biologiche
Bilancio energetico
- glicolisi: 1 glucosio viene convertito in
2 molecole di piruvato
⇒ 2 ATP + 2 NADH (~7 ATP)
- complesso piruvato deidrogenasi:
2 molecole di piruvato
convertite in 2 acetil-CoA
⇒ 2 NADH (~5 ATP)
5 ATP
2 ATP
5 ATP
- per ogni ciclo si generano ~ 10 ATP
⇒ 2 giri del ciclo dell’acido citrico
(uno per gruppo acetile) (~20 ATP)
15 ATP
3 ATP
2 ATP
In condizioni aerobiche 1 glucosio può potenzialmente promuovere la
sintesi di ~32 ATP. Solo 2 ATP in condizioni di anaerobiosi