Danio rerio (Zebrafish)
Adulto = lunghezza 3 cm
- Acque tropicali India e Sud-Est Asia
- Fecondazione esterna, uovo ed embrione
trasparenti
- Sviluppo molto rapido: dopo 24 h dalla
fertilizzazione tutti gli organi sono formati
- Dopo 3 gg dalla fertilizzazione esce dall’uovo
- Dopo 3-4 mesi raggiunge maturità sessuale
- Una femmina produce fino a 200 uova/settimana
Uova fecondate a diversi stadi
dello sviluppo
Danio rerio (Zebrafish)
Tavares B. et al. 2013
Danio rerio (zebrafish)
Vantaggi
- Sviluppo rapido e alto numero di uova
- Facilità di osservare lo sviluppo di tutti gli organi
- Rappresentatività di tipi cellulari, organi e sistemi fisiologici
- Facilità di manipolazione genetica e sperimentale
- Bassi costi di mantenimento
- Eccellente potenzialità per esperimenti High- Throughput
- Disponibilità di oltre 1000 linee transgeniche o mutanti (www.zfin.org)
Svantaggi
- Duplicazione dell’intero genoma (avvenuto nei teleostei) (ma in alcuni casi una copia è
diventata pseudogene)
- I mutanti embrionali sopravvivono anche in presenza di grosse malformazioni (morte
in utero nei mammiferi)
- Difficoltà di studio comportamenti cerebrali complessi
Pubblicazioni
2004-2013
532.000 topo
361.000 ratto
54.000 cane
34.000 Drosophila
15.000 zebrafish
13.000 C. elegans
Progenitore comune:
340 mln anni fa
APPROCCI PER LO STUDIO DELLA RELAZIONE
GENE-FENOTIPO
GENETICA CLASSICA (FORWARD): osservazione di un fenotipo mutante
spontaneo, individuare il gene in cui si è verificata la mutazione e
determinare la proteina codificata
Approcci:
- inducendo mutazioni a caso nel genoma e cercando il gene associato a
fenotipo
- con inserzione casuale di sequenze retrovirali o trasposoni
GENETICA INVERSA (REVERSE): conoscendo una molecola (sequenza di
DNA, RNA o una proteina) cercare di capire la funzione del gene
nell’organismo
Approcci:
- mutagenesi specifica nel gene d’interesse
- creando fenocopie: stesso fenotipo mediante interferenza mRNA
antisenso (mutazione non trasmissibile ad altre cellule o nella linea
germinale)
Il 70% dei geni umani ha almeno un ortologo in Zebrafish; alcuni geni
assenti; alcuni molto differenziati
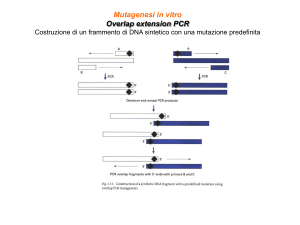
Strategie per la mutagenesi in Zebrafish
Fino al 2000: Forward genetics con mutagenesi casuale indotta: radiazioni gamma
(aberrazioni troppo grandi che coinvolgono più geni), sostanze mutagene (EMS o ENU),
integrazione casuale di sequenze trasponibili o retrovirus
Dal 2000 in poi: Reverse genetics con gene targeting
Produzione di mutazioni random nel genoma
Trattamento di cellule staminali degli spermatogoni con radiazioni
ionizzanti (raggi X) o mutageni chimici
Etil-metan-sulfonato
Aggiunge gruppi etilici a guanina o timina e
determina appaiamenti non corretti.
Ciclo successivo cambiamento coppia di basi
Produzione di mutazioni random nel genoma
N-etil-N-nitrosourea
Modifica la coppia A-T
Anni ‘90: caratterizzati circa 4000 fenotipi con alterazioni embrionali per:
Gastrulazione, sviluppo di somiti e strutture cranio-facciali, cervello, apparato
cardiovascolare
PRODUZIONE DI MUTAZIONI IN ZEBRAFISH
Genetica classica (Forward genetics)
- Produzione di mutazioni random nel genoma con sostanze mutagene (spermatozoi
con mutazione m X cellule uovo +)
- Individuazione fenotipi mutanti (+/m) (F1) (mutazione in eterozigosi)
- Incrocio di fenotipo mutante (+/m) X (+/+) wt
- Analisi fenotipica della progenie (F2) e identificazione del gene candidato con
marcatori genetici disponibili (microsatelliti, RFLP, SNP) (analisi di linkage):
positional cloning
- Clonaggio del gene (da librerie genomiche) e sequenziamento
- Eventuale selezione individui e incroci (+/m) X (+/m) per generare linee omozigoti
m/m (F3)
- Analisi del fenotipo
Mutagenesi casuale attraverso trattamento con ENU
Individuazione del locus mutato
ENU sciolto in acqua
Mutazioni random
c. germinali
Si stima una mutazione per genoma
Se fenotipo rilevabile: incroci con +/+ ,
analisi di linkage con marcatori polimorfici;
identificare il locus mutato in
librerie DNA genomico
Incrociati più volte tra di
loro per produrre omozigoti
In caso di fenotipicamente normali
si incrociano con wt per individuare
geni con effetti materni su precoci
stadi embrionali
Esempio di linkage in un albero genealogico
m+
++
m+
m+ ++
m+
++
m+ ++
m = allele fenotipo mutato; + = allele wildtype
Locus A = marcatore polimorfico
m+
Mutagenesi casuale per integrazione casuale retrovirale
Individuazione del locus mutato
Solo alcuni embrioni
avranno c. germinali
mutate = founder
founder X wt
o founder X founder
Linked-Mediated PCR
sequenziamento
intensivo per
identificazione del
locus con mutazione
PCR INVERSA
Linked-mediated (LM) PCR
DNA polimerasi
Sequenza vettore
retrovirale
Prodotto PCR clonato e sequenziato
PCR INVERSA
Mutagenesi inserzionale casuale: strategia per l’identificazione del tratto di DNA in
cui si è inserita la sequenza esogena (trasposone o retrovirus)
Mutanti per inserzione retrovirale
WT
A = mutaz. dominante:
Pinne molto lunghe
C= corpo curvo, edema
Intorno al cuore
B = bubble brain
D= ritardo sviluppo e
locomozione
Alterazioni struttura
occhio
J = difetti di pigmentazione
Ingrandimento
embrione J
PRODUZIONE DI MUTAZIONI IN ZEBRAFISH
Genetica inversa (Reverse genetics)
Negli ultimi 15 anni: gene targeting di geni candidati attraverso:
Mutagenesi inserzionale
- Utilizzo di transgeni (ricombinazione sequenze omologhe)
nelle ES (knock-in) (metodo non efficiente in zebrafish)
- Utilizzo di vettori retrovirali
- Utilizzo di sequenze trasponibili
Silenziamento genico
- RNA interference (RNAi) con dsRNA (poco efficiente)
- RNAi con “morfolini” antisenso
- Nucleasi a “dita di zinco” (ZFN) geneticamente modificata
- TALENS (Transcription activator-like TAL effector nucleases)
- CRISPR-Cas9 (Clustered Regularly Interspersed Short
Palindromic repeats)
- TILLING (Targeting Induced Local Lesions IN Genomes)
Metodi di gene targeting in Zebrafish
RNA interference con oligo antisenso modificati con morfolino
ZNF = DNA binding protein: Nucleasi a “dita di zinco” (ZFN)
TALENs = TALE proteina da un patogeno di una pianta composta da unità ripetute che si
legano a nucleotidi specifici
CRISPR-Cas9 = Sistema utilizzato da batteri per digerire DNA esogeno.
Clustered Regularly Interspaced Short Palindromic Repeats associata alla nucleasi Cas
In Zebrafish non viene applicato il gene-target knock-out per ricombinazione omologa
Studio degli effetti dell’RNA interference con produzione in vitro di
trascritti antisenso
RISC: RNA-induced silencing complex
Silenziamento genico in Zebrafish con morfolino
Studio degli effetti in malattie con alleli loss-of-function o dominanti negativi
- RNA interference con microiniezione di dsRNA non efficaci, interferenze aspecifiche
quindi tossicità per l’embrione
- RNA interference con oligo morfolino antisenso: le basi non attaccate allo zucchero
ribosio ma ad un anello di morfolino; legami fosforodiamidati (non fosforodiesterici) tra
le basi.
Struttura del morfolino
- Microiniezione in embrione stadio 1/2 cellule
morfolini 25 nt
- Molto stabili
- Si legano al 5’ del trascritto inibiscono traduzione
oppure
- Si legano ai siti di splicing provocando splicing
alternativo
Svantaggi: effetto transitorio (3-5 gg). Solo studio
a livello embrionale
Possibile co-iniezione di mRNA geni ortologhi umani
Nucleasi a “dita di zinco” (ZFN) geneticamente
modificata
Genome editing con nucleasi a “dita di zinco” (ZFN)
geneticamente modificata
Urnov FD et al. 2010
FokI: nucleasi batterica isolata inizialmente dal batterio Flavobacterium
okeanokoites;
Composta da 2 domini separati: un dominio N-terminale (DNA-binding
domain) e un dominio C-terminale per il taglio del DNA (DNA-cleavage
domain)
Il DNA-binding domain riconosce una sequenza non palindromica e il DNAcleavage domain taglia il DNA in modo non specifico ad una distanza di 9-13
nucleotidi a valle del sito di legame.
L’enzima FokI è attivo solo come dimero: due molecole di FokI si legano a
ciascun DNA-binding domain e poi dimerizzano in modo che i due domini
DNA-cleavage formino una endonucleasi funzionale
Genome editing con nucleasi a “dita di zinco” (ZFN)
geneticamente modificata
Microiniezione di coppie di
mRNA che tradotti produrranno
catena polipeptidica con
domini zinc-finger e nucleasi FokI
Deriva dal gene FokI che
codifica endonucleasi
batterica di restrizione
non specifica
Assemblaggio di domini zinc-finger
specifici di una regione genica
Sistemi naturali di riparo della
rottura della doppia elica
Unione non omologa delle
estremità = Non-homologous
end joining
Sistema con
ricombinazione
omologa
Genome editing con nucleasi a “dita di zinco” (ZFN)
geneticamente modificata
Genome editing con nucleasi a “dita di zinco” (ZFN)
geneticamente modificata
Metodo di gene targeting per inserire mutazione: conversione da allele B ad allele A
(DNA esogeno come stampo)
INATTIVAZIONE GENICA MEDIANTE ZNF (o TALE) GENETICAMENTE MODIFICATE
Coppia di mRNA microiniettati
Attivazione sistemi di riparazione
che genera mutazioni
Individuazione DNA mutati:
-alterazione del taglio di enzimi
nel gene target;
-digestione con enzimi che
tagliano ssDNA per presenza di
mismatch (CEL I o T7
endonucleases), oppure
-Analisi di mismatch
(heteroduplex) e
sequenziamento
CRISPR-CAS9 = Clustered Regularly Interspaced Short Palindromic Repeats
associata alla nucleasi Cas9 (COMPLESSO RIBONUCLEOPROTEICO)
Strutturale naturale
RNA-Guided Endonuclease technology. Doppio RNA: crRNA (crispRNA) unito alla sequenza
target e trascrRNA (trans activating crRNA) .
crRNA ibrido: lega la sequenza genica target e ibrida per complementarietà una sequenza di
trascrRNA. Il complesso riconosciuto e tagliato dalla nucleasi Cas 9
Necessaria la presenza di Protospacer Adjacent Motif (PAM) Sequence. Riparo del DNA con
mutagenesi
CRISPR-CAS9 = Clustered Regularly Interspaced Short Palindromic Repeats
associata alla nucleasi Cas9 (COMPLESSO RIBONUCLEOPROTEICO)
Co-iniezione di RNA sintetici (naturalmente
presenti in batteri) e mRNA per la nucleasi
Cas9 in uova fecondate (o in vettori di
espressione)
gRNA = RNA guida composto in parte da crRNA
e trascrRNA
Introduzione diretta in cellule dei complessi ribonucleoproteici. Nature Protocols 10, 1842–
1859 (2015) doi:10.1038/nprot.2015.117Published online 22 October 2015
Genome targeting con la tecnica TILLING
Metodi di gene targeting in Zebrafish
ESEMPI DI INATTIVAZIONE GENICA CON TECNICA ZNF
ESEMPI DI CORREZIONE O INSERZIONE GENICA CON TECNICA ZNF