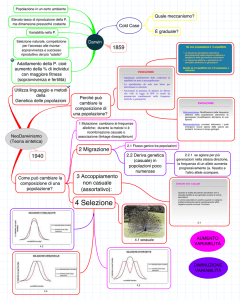
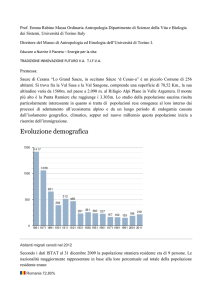
Dall’idea al prodotto
Come impostare una ricerca scientifica
Genetica medica
•Studi di associazione (geni/malattie)
•Screening genetico
•Malattie multifattoriali (es. QTL)
•Malattie mitocondriali
•Analisi pre-impianto
Genetica Forense
•Identificazione individuale
•Test di paternità/maternità
Genetica Evoluzionistica
•Struttura delle popolazioni
•Effetti della microevoluzione sulle popolazioni
•Storia delle popolazioni
ANTROPOLOGIA MOLECOLARE
È il campo dell’antropologia che fa inferenze sull’evoluzione umana
analizzando la variabilità biologica inter- e intra-specifica a livello
molecolare
ANTROPOLOGIA CULTURALE
ARCHEOLOGIA
LINGUISTICA
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Analisi
statistiche
Scelta
marcatori
Ricerca
finanziamenti
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
L'Italia è uno dei paesi europei più ricchi di biodiversità, sia animale
che vegetale del bacino Mediterraneo e dell’Europa in generale.
Numero di specie di mammiferi
Numero di specie di mammiferi endemici
Tundra alpina
Zona arida mediterranea
Questo elevato grado di diversità, biologica e
culturale, è anche osservabile a livello della
variabilità genetica delle popolazioni umane?
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Analisi
statistiche
Scelta
marcatori
Ricerca
finanziamenti
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
34 VARIABILI
28 LOCALITÀ
PIAZZA ET AL. 1988
Mappe sintetiche delle 3 prime componenti
principali
•Chiaro gradiente Nord-Sud (più evidente per
l’Italia meridionale)
Relazione con differenze morfologiche tra
Centro-Nord e Sud Italia
•Gradiente Est-Ovest in Sicilia
Popolamento preistorico
Dominazione Normanna
Componente principale 1 (27% variabilità)
34 VARIABILI
28 LOCALITÀ
PIAZZA ET AL. 1988
Mappe sintetiche delle 3 prime componenti principali
Popolamento Etrusco
Roma e Napoli : movimenti migratori
dall’entroterra (2000 B.P. e 500 B.P.)
Componente principale 2 (18% variabilità)
34 VARIABILI
28 LOCALITÀ
PIAZZA ET AL. 1988
Mappe sintetiche delle 3 prime componenti principali
Zona scura: centro della regione abitata
dai Liguri
Zona chiara: Centro della regione
abitata dai Piceni
Componente principale 3 (14% variabilità)
34 VARIABILI
28 LOCALITÀ
PIAZZA ET AL. 1988
Somma delle tre prime mappe sintetiche
La somma delle tre Componenti
principali chiarisce i risultati osservati
per le singole componenti.
Popolamento antico
Preromanico
Barbujani et al. 1995
12 popolazioni (5 Sardegna)
1072 individui
12 RFLP
BamH I
Hae II
Msp I
Ava II
3 siti
3 siti
2 siti
4 siti
Barbujani et al. 1995
Frequenze
aplotipiche
gene diversity totale = 0,471
Within =0,444 Between=0,027
La diversità mitocondriale in Italia può essere
attribuita a differenze tra località (<6%)
Gli aplotipi più comuni sono gli stessi e con frequenze simili
per tutte le località campionate.
Separazione recente da
un pool genico
ancestrale
geneticamente
eterogeneo
Barbujani et al. 1995
Correlazione spaziale per 4 classi di distanze
7 loci mostrano correlazione
spaziale
Capelli et al. 2007
12 località
699 campioni
17 SNP’s e 10 STR
Capelli et al. 2007
Aplogruppo più comune Hg R1*
Eccetto West Calabria (J2)
80%
Capelli et al. 2007
Capelli et al. 2007
Analisi di mescolamento
Stima del contributo genetico di due popolazioni sorgente alla popolazione presa in
esame
Iberia (popolamento paleolitico)
Anatolia (transizione neolitica)
Tutti
Sud
Nord
Boattini et al., 2013
SPCA (Spatial analysis of Principal Components)
Prima componente: struttura lungo l’asse Nordest-Sudovest
Seconda componente: strutturazione della Sardegna
Cromosoma Y
Boattini et al., 2013
SPCA (Spatial analysis of Principal Components)
Prima componente: strutturaizone della Sardegna
Seconda componente:struttura lungo l’asse Nordest-Sudovest
mtDNA
Il cline nordest-sudovest indicherebbe la presenza di due distinti
processi di neolitizzazione in Italia
La diversità della popolazione Sarda che sarebbe stata meno
influenzata dai flussi genici che hanno interessato il resto della
penisola e la Sicilia
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Analisi
statistiche
Scelta
marcatori
Ricerca
finanziamenti
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
•Diversi lavori che hanno preso in considerazione le popolazioni
aperte dell’Italia
•Diversi lavori che hanno investigato la variabilità genetica delle
popolazioni soggette a fattori di isolamento geografico e/o
culturale
Nessuno studio che abbia investigato in maniera sistematica la
variabilità genetica delle popolazioni Italiane nel loro
complesso
•Analizzare la struttura genetica di numerose popolazioni
Italiane allo scopo di valutare possibili associazioni tra fattori
geografici e linguistici e isolamento genetico
•Comparare il grado di variabilità genetica delle popolazioni
Italiane con quelle Europee, prendendo in considerazione sia
quelle “aperte” che quelle isolate
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Analisi
statistiche
Scelta
marcatori
Ricerca
finanziamenti
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
PROGETTI DI INTERESSE NAZIONALE (PRIN)
PRIN 2007
Isolating the isolates: analisi dei fattori geografici e culturali
della variabilità genetica umana
PRIN 2009
La Biodiversità umana in Italia: patterns microevolutivi
Prof. Davide Pettener
Università di Cagliari
Prof. Giuseppe Vona
Prof. Carla Calò
Prof. Giovanni Destro Bisol
Università di Pisa
Prof. Giorgio Paoli
Dr. Sergio Tofanelli
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Analisi
statistiche
Scelta
marcatori
Ricerca
finanziamenti
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
• Molecola circolare di 16569bp
• Regione codificante
• 37 geni (13 proteine, 22 tRNA, 2rRNA)
• Regione di controllo (Hrv 1 e 2)
• 58 milioni di bp
• 86 geni che codificano per
23 diverse proteine
• Regioni pseudoautosomiche
(5% del cromosoma)
MtDNA
e cromosoma Y PRO E CONTRO
PRO
CONTRO
mtDNA
Cromosoma Y
Eredità uniparentale materna
Eredità uniparentale paterna
Eredità uniparentale
Assenza di ricombinazione
NRY
Taglia effettiva bassa
SNPs:
- HVR (evol. veloce)
- regione codificante (evol.
lenta)
Soggetto a deriva
-SNPs (evol. lenta)
-STRs (evol. veloce)
Forte strutturazione geografica
Piccole dimensioni
Molte copie per cellula
Strutturazione geografica
Aplotipo
Combinazione di varianti alleliche lungo un
cromosoma o segmento cromosomico contenente loci
strettamente associati tra loro
ATTTCCCCTAGGTAGA
ATTTGCACTAGGCAGA
Aplotipo 2
ATTTCCCCTAGGTAGA
ATTTCCCCTAGGTAGA
Aplotipo 1
ATTTGCACTAGGCAGA
gli alleli della regione non ricombinante del cromosoma Y (NRY)
sono sempre associati a formare aplotipi, così come gli alleli del
genoma mitocondriale (mtDNA). Infatti queste due porzioni del
genoma non ricombinano, essendo ereditate con modalità
uniparentali, paterna la prima, materna la seconda.
Aplogruppo:
gruppo di aplotipi di cui si ipotizza un’origine comune,
grazie alla condivisione di mutazioni caratteristiche
(generalmente ad evoluzione lenta)
Aplogruppo mtDNA
Si definisce sulla base della condivisione di mutazioni
specifiche in posizioni con un basso tasso di mutazione
(stabili)
1
Aplotipi non identici possono appartenere allo stesso
aplogruppo
2
3
4
Aplogruppo Y
Si definisce sulla base della condivisione di mutazioni specifiche per marcatori biallelici (SNPs), e
non per i microsatelliti (tasso di mutazione troppo alto)
Filogenia:
Struttura ad albero che rappresenta le relazioni evolutive tra un
insieme di taxa (dove per taxon si intende un’unità evolutiva,
quindi dall’aplotipo… alla specie!)
La filogenia fornisce una dimensione temporale ed evolutiva
ANCESTORE COMUNE
AAAGGTACC
G T mutation
AAATGTACC
A G mutation
AAATGTACC
AAATGTGCC
A T mutation
AAATGTGCC
TAATGTGCC
Filogenia MtDNA
SNPs selezionati
nella regione codificante meno
variabile e più stabile
Sequenze complete dell’intera
molecola di mtDNA
(circa 16000 seq. complete
pubblicate)
Filogenia cromosoma Y
Filogenia a più alta risoluzione rispetto ad
altri loci genetici.
Filogeografia
Analisi della distribuzione geografica di diversi rami all’interno di
una filogenia.
La filogenia fornisce una dimensione temporale ed evolutiva che è
combinata con la dimensione spaziale della geografia
Cromosoma Y
MtDNA
60 SNP’s e 17 STR
HVRI e II è17 SNPs della
regione codificante
Approccio gerarchico
13 SNPs che definiscono i
principali aplogruppi
Ulteriori SNPs per
identificare le sottolineee
di specifici aplogruppi
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Analisi di
laboratorio
Strategia di
campionamento
Elaborazione ed
interpretazione
dei dati
•ORIGINARI DELLA ZONA DA
ALMENO DA 3 GENERAZIONI
(regola dei nonni)
•Cognomi fondatori, linee
mtDNA fondatrici
•Previa firma del consenso
informato
SCOPO DEL PROGETTO
SPIEGAZIONE DETTAGLIATA DELLA PROCEDURA DI CAMPIONAMENTO E UTILIZZO DEI
DATI
SPIEGAZIONE DETTAGLIATA DELLA PROCEDURA DI CAMPIONAMENTO E UTILIZZO DEI
DATI
DIRITTI DEI PARTECIPANTI
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Analisi di
laboratorio
Strategia di
campionamento
Elaborazione ed
interpretazione
dei dati
•Popolazioni “aperte” Italiane ed Europee
•Popolazioni soggette a fattori di isolamento geografico e/o
linguistico Italiane ed Europee
57 popolazioni
10 popolazioni soggette a fattori di isolamento
geolinguistico
16 popolazioni soggette a fattori di isolamento
geografico
3 popolazioni soggette a fattori di isolamento
linguistico
28 popolazioni “aperte”
Sequenze mtDNA HVRs
>Ind1
TATTGTACGGTACCATAAATACTTGACCACCTGTAGTACATAAAAACCCAATCCACATCAAA
ACCCCCTCCCCATGCTTACAAGCAAGTACAGCAATCAACCCTCAACTATCACACATCAACTG
C
SNPs cromosoma Y e mtDNA (metodo snapshot)
STR cromosoma Y
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Analisi di
laboratorio
Strategia di
campionamento
Elaborazione ed
interpretazione
dei dati
Caso studio: The Arbereshe: between Italy and the Balkans
Una delle minoranze linguistiche più numerose in Italia
50 comunità con circa 100000 individui
Arbereshe Calabresi
30 comunità circa 60000 individui
Arbereshe Siciliani
3 comunità circa 15000 individui
Lingua: Arbereshe (albanese)
Religione: Cristiana ortodossa
Origine: Albania meridionale (Toskeria)
Età insediamento in Italia: 1400-1500 DC
Caso studio: The Arbereshe: between Italy and the Balkans
Dati biodemografici evidenziano una differenziazione rispetto al contesto Italiano indicando un certo grado
si isolamento culturale e geografico
Analisi di mescolamento
Cromosoma Y
mtDNA
Caso studio: An analysis of isolated populations in Sardinia
Popolazioni:
Carloforte (isola di San Pietro) isolamento geo/lingustico
Origine: Fondazione 1738 AC da parte di migranti Liguri
provenienti dall’isola di Tabarka(Tunisia).
Lingua: dialetto ligure antico di Pegli (Tabarkino)
Popolazione: 6420
Benetutti isolamento geografico
Popolazione: 2010
Caso studio: An analysis of isolated populations in Sardinia
Entrambe le popolazioni mostrano segnali di riduzione della variabilità interna sia per mtDNA che per il
cromosoma Y
Cromosoma Y
mtDNA
Probabile mescolamento differenziale, maschi di Carloforte e femmine Tunisine
Linguistic, geographic and genetic
isolation in Italian populations
mtDNA
57 popolazioni
Cromosoma Y
46 popolazioni
Valori più bassi di HD per le popolazioni isolate
In particolare significativi per Sappada, Ogliastra
Valori più bassi di HD per le popolazioni isolate
In particolare significativi per Luserna, Timau e
Sappada
Valori più alti di Fst medio per le popolazioni isolate
In particolare significativi per Sappada, Vallepietra e Sauris
Valori più alti di Fst medio per le popolazioni isolate
In particolare significativi per Luserna, Timau e Sappada
Multidimensional scaling (MDS)
basato sulla variabilità del mtDNA
Multidimensional scaling (MDS) basato
sulla variabilità del cromosoma Y
Queste analisi non ci permettono di valutare quanto pesano i fattori linguistici
e geografici sul pattern osservato
Analisi degli outliers tramite il metodo dell’interquartile
Quanto i valori delle popolazioni soggette a fattori di isolamento si discostano dalla distribuzione osservata per le
popolazioni aperte
MAT
COM
LEC
COS
AQU
AVI
BNV
PIC
TVA
ORI
PIL
VIC
CAS
TER
CMB
BOZ
CAD
NSA
BRE
TRE
CUN
SPZ
SGL
UDI
0.995
0.995
0.995
0.994
0.993
0.990
0.989
0.988
0.988
0.986
0.983
0.981
0.979
0.978
0.977
0.976
0.976
0.976
0.973
0.969
0.963
0.962
0.926
0.903
i quartili sono quei valori che ripartiscono la popolazione in quattro parti
di uguale numerosità
Interquartile=Q3*1.5(Q3-Q1)
=Q3*3(Q3-Q1)
Media Q3=0.989
Media Q2=0.980
Media Q1=0.975
6 su 10 (60%)
5 su 16 (31%)
0 su 3 (0%)
2 su 9 (22%)
5 su 10 (50%)
1 su 3 (33%)
Linguistic, geographic and genetic isolation in Italian populations
•Analizzare la struttura genetica di numerose popolazioni Italiane allo scopo di
valutare possibili associazioni tra fattori geografici e linguistici e isolamento genetico
•In Italia sono presenti numerose popolazioni che mostrano forti segnali di isolamento
genetico
•Questi segnali sono più frequenti in popolazioni soggette contemporaneamente sia a
fattori di isolamento geografici che linguistici
Re-assessing genetic diversity of Italian populations
AMOVA (within group)
Italiani senza isolati
0.38%
Europei senza isolati
0.33%
Italiani con isolati
1.89%
Europei con isolati
1.52%
AMOVA (within group)
Italiani senza isolati
3.19%
Europei senza isolati 6.89%
Italiani con isolati
8.95%
Europei con isolati
10.60%
Re-assessing genetic diversity of Italian populations
AMOVA (within group)
Italiani senza isolati
0.38%
Europei senza isolati
0.33%
Italiani con isolati
1.89%
Europei con isolati
1.52%
AMOVA (within group)
Italiani senza isolati
3.19%
Europei senza isolati 6.89%
Italiani con isolati
8.95%
Europei con isolati
10.60%
Re-assessing genetic diversity of Italian populations
Alti valori di Fst in Italia anche per distanze
brevi (0-200 Km)
Effetto dovuto probabilmente alle isole
germanofone delle Alpi orientali
Alti valori di Fst in Italia anche per distanze
700-800 Km)
Da attribuirsi alle distanze tra le
popolazioni Sarde e quelle del continente
Re-assessing genetic diversity of Italian populations
Alti valori di Fst in Italia anche per distanze
brevi (0-200 Km)
Effetto dovuto probabilmente alle isole
germanofone delle Alpi orientali
Alti valori di Fst in Italia anche per distanze
700-800 Km)
Da attribuirsi alle distanze tra le
popolazioni Sarde e quelle del continente
Re-assessing genetic diversity of Italian populations
•Comparare il grado di variabilità genetica delle popolazioni Italiane con quelle Europee,
prendendo in considerazione sia quelle “aperte” che quelle isolate
•Una estrema variabilità genetica in Italia, talvolta anche maggiore rispetto a quella
osservata nel resto del continente Europeo, in particolare per mtDNA
•La variabilità genetica nelle popolazioni Italiane mostra un pattern comparabile con
quanto osservato, a livello di specie, per piante ed altri animali
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
Scrittura dell’articolo
SCELTA DELLA RIVISTA
Le riviste hanno scopi ed interessi diversi (linee guida)
Specifiche (di settore):es. America Journal of Physical Anthropology, American Journal of
Human genetics etc...
generaliste : es. Science, Nature, Plos One etc...
AMERICAN JOURNAL OF PHYSICAL ANTHROPOLOGY
The Editor-in-Chief welcomes for consideration manuscripts that contribute to an
understanding of the evolution of members of the order Primates, with particular
emphasis on human biological evolution and variation.
PLoS ONE is designed to communicate primary scientific research. We welcome
submissions in any discipline that will significantly contribute to the base of knowledge in
the sciences.
Scrittura dell’articolo
• Titolo, autori, affiliazioni
•Abstract
• Introduzione
• Materiali e metodi
• Risultati
• Discussioni
• Conclusioni
abstract should
not exceed 300 words. It should:
TheThe
introduction
should:
•Describe
main objective(s)
of the
study into
•Provide
somethe
background
to put the
manuscript
•Explain
study outside
was done,
any
context
andhow
allowthe
readers
theincluding
field to understand
organisms
used, without
detail
themodel
purpose
and significance
of themethodological
study
•Summarize
the provide
most
important
and
their
This
section
should
enough
detail
allow
•Define
the problem
addressed
and results
why itto
is
important
significance
suitably
skilled
fully
replicate your
•Include
a briefinvestigators
review of thetokey
literature
These
may
all
be separate,
ordisagreements
may befor
combined
study.
Specific
information
and/ororprotocols
newin theto
•Notesections
any relevant
controversies
create
Results/Discussion
section
methods
should
be included in detail.
field a mixed
Together,
these
should
describe
the
results
Methods
sections
of papers
with
data
should
•Conclude
withsections
a brief
statement
of thethat
overall
aim of
ofthe
the
experiments,
the
interpretation
of
these
results,
and
the
be
deposited
in a publicly
available
work
and a comment
about
whetherdatabase
that aim should
was achieved
conclusions
that
can
be
drawn.
Authors
should
explain
specify where the data have been deposited and
how
the results
relateaccession
to the hypothesis
as the
provide
the relevant
numberspresented
and version
basis
of theifstudy
and provide a succinct explanation of the
numbers,
appropriate.
implications of the findings, particularly in relation to
previous related studies and potential future directions for
research.
Scrittura dell’articolo
BIBLIOGRAFIA
Ogni rivista ha le proprie regole per la bibliografia
The American Journal of Human Genetics
Nelson, M.R., Bryc, K., King, K.S., Indap, A., Boyko, A.R., Novembre, J., Briley, L.P.,
Maruyama, Y., Waterworth, D.M., Waeber, G., et al. (2008). The Population Reference
Sample,
POPRES: A resource for population, disease, and pharmacological genetics research.
Am. J. Hum. Genet. 83, 347–358.
Genome Biology
Price AL, Patterson NJ, Plenge RM, Weinblatt ME, Shadick NA, Reich D: Principal
components analysis corrects for stratification in genome-wide association studies. Nat
Genet 2006, 38:904-909.
Molecular Ecology
Pavoine S, Ollier S, Pontier D (2005) Measuring diversity from dissimilarities with Rao’s
quadratic entropy: are any dissimilarities unsuitable? Theoretical Population Biology, 67,
231–239.
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
Sottomissione alla rivista
Cover letter
You should supply an approximately one page cover letter that:
Concisely summarizes why your paper is a valuable addition to the scientific literature
Briefly relates your study to previously published work
Specifies the type of article you are submitting (for example, research article, systematic
review, meta-analysis, clinical trial)
TABELLE E FIGURE
Figure legends
Figures should not be included in the manuscript file, but figure legends should be.
Figure legends should describe the key messages of a figure. Legends should have a short
title of 15 words or less. The full legend should have a description of the figure and allow
readers to understand the figure without referring to the text. The legend itself should be
succinct, avoid lengthy descriptions of methods, and define all non-standard symbols and
abbreviations.
Tables
Tables should be included at the end of the manuscript. All tables should have a concise
title. Footnotes can be used to explain abbreviations. Citations should be indicated using
the same style as outlined above. Tables occupying more than one printed page should be
avoided, if possible. Larger tables can be published as Supporting Information.
Sottomissione
referaggio
Identificazione
della
problematica
Referaggio
(peer review)
Stato dell’arte
(ricerca
bibliografica)
Dati di
confronto
Sottomissione
a rivista
Scrittura
articolo
Messa a punto
della ricerca
Ricerca
finanziamenti
Analisi
statistiche
Scelta
marcatori
Strategia di
campionament
o
Analisi di
laboratorio
Elaborazione ed
interpretazione
dei dati
Peer Review
La procedura di selezione degli articoli o dei progetti di ricerca proposti da membri
della comunità scientifica, effettuata attraverso una valutazione esperta eseguita da
specialisti del settore per verificarne l'idoneità alla pubblicazione scientifica su riviste
specializzate o, nel caso di progetti, al finanziamento degli stessi.
MOTIVAZIONI:
La ragione principale della revisione dei pari sta nel fatto che è spesso molto difficile per
un singolo autore, o per un gruppo di ricerca, riuscire a individuare tutti gli errori o i difetti
di un proprio studio, che sia questo più o meno complesso.
La PEER REVIEWsottopone il lavoro o le idee di un autore allo scrutinio di uno o più esperti
del medesimo settore. Ognuno di questi esperti fornisce una propria valutazione includendo
anche suggerimenti per l'eventuale miglioramento. Le valutazioni solitamente includono
raccomandazioni esplicite su cosa fare del manoscritto, spesso scelte tra opzioni proposte
dal giornale o dall'editore.
• il lavoro è accettato senza riserve
• il lavoro è accettato, a patto che l'autore lo migliori sotto determinati aspetti (Major or
Minor revision)
• il lavoro è respinto, ma se ne incoraggia una revisione e una riproposta
The ms by Coia et al "Evidence of high genetic variation among linguistically diverse populations on a
microgeographic scale:the cae study of Italian Alps" presents mtDNA data from nine population from
the Eastern Italian Alpine region. The authors claim three main results
1) Different demographic histories for the west-central and eastern areas;
2) German speaking Cimbri as outliers, with signatures of founder effect and low growth rate
3) Different ladin groups being different from each other but similar to neighbours
These results are indeed of interest and worth being presented but I think that the results presented so
far do not fully support some of their conclusions.
In details:
1-the indication of different demographic histories seem to rely on the results presented in Table 1 and
2. However, it is worth noticing that there seems to be a correlation between sample size and H, HD
(table 1) as well as with the Fu'sFs values and HRI (table 2). The claimed differences are related to the
fact that they "failed to detect any signatures of past demographic expansion on the Luserna plateau
and Fersina valley, whiche were, by contrst, statistically robust for the Adige, Giudicarie, NOn and Sole
valleys and to a lesser extent in the Fiemme, Fassa and Primiero valleys". It should be noted that
Fersina and Luserna samples have the lowest sample sizes (25 and 21), followed by Fiemme, Fassa and
Primiero (41, 47 and 40) and then Adige, Giudicarie, NOn and Sole (56, 52, 48 and 63), raising the issue
of how sample size is affecting their results.
2-The Cimbri uniqueness might be again an artefact of the small (smallest) population size. How would
a resampling of the larger samples available to authors behave when small subset of 20 samples are
analysed? Authors are aware of this issue and address it on page 13 with a list of explanation of why
the small sample size is representative of a small population. This is well discussed but it is still
necessary to prove that 20 random samples from the populations with the strongest signals of
expansion do not lose such signal.
Also, they should also show how the bio-demography of the other groups is truly different from that of
the Cimbri (page13) so that it does not appear as an ad-hoc explanation
Page 8:authors underline the high frequencies of hg H6 and K in the Fersina sample and in
the Cimbri: are these frequencies significantly different from the other samples?
Page 8: Can authors explain what the ages of the lineages are meant to indicate? Age of
expansion or the coalescent time?
Page 8: Authors propose a cultural model for the Neolithic colonisation of the Alps. Does
this find a correspondence in other part of the continent? How does it correlate with the
recently proposed Neolithic origin of the most common European Y chromosome type R1b
(Balaresque et al, 2010), whose frequency is more than 50% in the Alpine region (hg1 in
Pichler et al, 2006; M173xM17 in Capelli et al, 2007)?
Page 10, First paragraph: Are authors referring to genetic differentiation (instead of genetic
diversity as mentioned in the text)
Page 10:please mention in their completeness the three other populations showing the
highest genetic diversity (but see above)
On page 11 authors indicate that "the observed pattern of genetic variation cannot be
simply explained by geography . The Authors should explain more clearly the contribution
of geography that they implicitly recognise
Please include references on ancient DNA works to back up the end of the "conclusions"
section
RFLP (Restriction Fragment Length Polymorphism)
G
C
T A A
C
G
A T T
Enzima di restrizione
120 bp
300 BP
180 BP
120 BP
300 bp
180 bp