Il cancro è una malattia
genetica
•
•
•
•
Deriva da alterazioni della sequenza del DNA
Mutazioni somatiche
Mutazioni che non vengono trasmesse alla
generazione successiva e che non possono
essere perciò ereditate
Tali mutazioni vengono trasmesse alle
cellule figlie dopo la divisione cellulare
Il cancro è una patologia
monoclonale
Tutte le cellule neoplastiche di un individuo
originano da una singola cellula progenitrice
La cellula clonale tumorale esposta ad un
agente mutageno subisce un danno
irreversibile al DNA
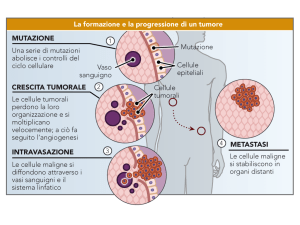
Lo sviluppo del tumore è un processo che
avviene in diverse fasi
Nuovi sub-cloni originano dai discendenti della cellula
tumorale originale (espansione monoclonale).
Anche se in origine i tumori sono monoclonali, nel
momento in cui il tumore diventa evidente le sue
cellule presentano un estrema eterogeneità.
Con la progressione la massa tumorale si arricchisce di varianti cellulari tumorali più
adatte a eludere le difese dell’ospite e tendenti ad una maggiore aggressività
La genotipizzazione del tumore ha il potenziale di classificare i
tumori permettendo la pianificazione di strategie cliniche
mirate ed identificare i pazienti che rispondono ai diversi
farmaci. Questa genotipizzazione è permessa dall’analisi di
specifici biomarcatori.
•Durante la progressione del tumore però i biomarcatori possono
variare (eterogeneità) .
•Pertanto l’analisi del genoma tumorale su campioni in serie
dovrebbe rappresentare un prerequisito fondamentale per la
terapia personalizzata al fine di monitorare la risposta alla
terapia.
COME SI SVILUPPA IL CANCRO
Lo sviluppo dei tumori nell’uomo è il risultato di una complessa
interazione tra:
Fattori genetici
Sono necessarie diverse
mutazioni in geni diversi
per la trasformazione
neoplastica della cellula
Fattori ambientali
Vie cellulari alterate dal
cancro
•
Tre sono i processi importanti che regolano
il numero globale delle cellule di un
individuo:
•
La divisione cellulare
La morte cellulare
Il differenziamento
•
•
Tre tipi di geni
•
Esistono tre tipi principali di geni mutati che
contribuiscono allo sviluppo di un tumore
•
Oncogeni
Geni oncosoppressori
Geni coinvolti nel riparo del DNA
•
•
Oncogeni
Oncogeni: geni che controllano positivamente la
proliferazione cellulare
•
Scoperti nei virus trasformanti ad RNA
•
Il nome deriva dal virus in cui sono stati identificati:
e.g. ras da Rous Sarcoma Virus
•
Controparti cellulari normali: proto-oncogeni che
stimolano crescita e divisioni cellulari
Proto-oncogeni
Oncogeni
Mutazione per acquisizione di
funzione
Ruolo degli oncogeni
•
•
Ruolo normale: stimolazione crescita e
proliferazione cellulare
Mutazione > Aumento della funzione >
trasformazione, invasività
Funzioni dei protoncogeni:
stimolazione della crescita
•
•
•
•
Fattori di crescita secreti
Recettori di superficie cellulare
Fattori intracellulari di trasduzione del
segnale
Proteine nucleari che legano il DNA
Mutazioni
•
Mutazioni puntiformi
•
Traslocazioni cromosomiche
Mutazione puntiforme
•
•
•
Oncogene ras (carcinoma della
vescica) sul cromosoma 12
Mutazione puntiforme: Gly12Val
Blocco della conversione di ras dalla
forma attiva, legata al GTP, alla forma
inattiva, legata al GDP, che deregola la
crescita cellulare
Ras signaling pathway
Traslocazione cromosomica: CML
t(9;22)(q34;q11)
t(9;22)(q34;q11)
•
•
•
9q34: ABL (abelson virus) protooncogene
22q11: BCR (breakpoint cluster region)
Fusione BCR-ABL sul cromosoma 22
(cromosoma Philadelphia)
Oncogeni
•
•
Mutazioni negli oncogeni sono di solito
acquisite
Mutazioni dominanti: una sola copia
genica mutata è sufficiente a produrre il
cancro per acquisizione di funzione “gain of function”
Tumor-suppressor genes
Oncosoppressori
•
Controllo negativo della proliferazione
cellulare
Prevenzione di crescita cellulare abnorme
– Normalmente inibiscono la crescita e la
divisione cellulare
– PRb: protoncogene presente su 13q14
–
Tumour suppressor genes
•
Retinoblastoma (Rb): bilaterale nel 30%
dei casi
•
Questo tumore può esistere sia in
forma ereditaria che in forma sporadica
•
•
•
Nella forma familiare della malattia, una mutazione
germinale del gene Rb è trasmessa al bambino ed è
presente in tutte le cellule.
Una seconda mutazione viene acquisita in un
particolare retinoblasto che dà origine ad un tumore
della retina.
La trasmissione ereditaria di un gene mutato
aumenta di molto la prpbabilità che possa avvenire
una seconda mutazione
•
•
Nella forma sporadica entrambe le mutazioni
avvengono a livello somatico nello stesso
retinoblasto.
Questo è un evento abbastanza raro ecco
perchè i casi sporadici di solito colpiscono
soltanto un occhio rispetto ai casi familiari
Ipotesi dei due colpi
•
•
•
1971: Knudson’s two-hit hypothesis (ipotesi del
doppio colpo)
Casi familiari di Rb: sono necessarie due mutazioni
separate, una in ciascuno dei due alleli del
retinoblastoma per inattivare le due copie dell’allele
di Rb ed impedire l’espressione della proteina rb
– 1° mutazione germinale (inattivazione di un allele)
– 2° mutazione somatica (perdita del secondo allele)
Casi sporadici: 2 mutazioni somatiche nella stessa
cellula
Ereditarietà degli oncosoppressori
•
•
La maggior parte dei tumori ereditari sono dovuti
all’ereditarietà di un gene oncosoppressore mutato
(first hit)
Ma è necessario un secondo colpo (second hit) per
lo sviluppo del tumore, pertanto il gene agisce in
modo recessivo a livello cellulare
•L’ereditarietà degli oncogeni è di tipo dominante in
quanto basta che sia mutata una copia del gene
affinchè compaia il tumore.
•Nel caso degli oncosoppressori è necessario che
entrambe le copie siano mutate.
Geni coinvolti nel DNA repair
• Riparano i danni al DNA
• sono considerati un sottogruppo di Geni
Oncosoppressori
• La perdita della loro funzione determina
l’accumulo di mutazioni in altri geni
cruciali
Il gene p53
•
•
•
•
•
La più comune modificazione genetica
associata a cancro
oncosoppressore, “guardiano del genoma”
Espresso in grande quantità in cellule con
danni al DNA
Coinvolto nella fase G1/S del ciclo cellulare
(blocca la replicazione di cellule
danneggiate)
Coinvolto nell’apoptosi
BRCA1 e BRCA2
•
Proteine la cui espressione si modifica in
caso di “risposta al danno del DNA”
•
hanno attività di oncosoppressori
•
Responsabili dell’insorgenza del cancro
della mammella
PROTEINA APC (Adenomatous Polyposis Coli)
•E’ un gene oncosoppressore coinvolto nel controllo
del processo di divisione cellulare
•Mutazioni della proteina APC determinano
aumento della divisione cellulare
•Tumor suppressor gene nel cancro del colon retto
LEUCEMIE
•
La leucemia è una malattia che deriva dalla
proliferazione neoplastica di cellule emopoietiche
•
Le leucemie sono suddivise in due classi principali
in base alla morfologia della linea cellulare da cui
originano:
–
–
•
Leucemia mieloide
Leucemia linfoide
Inoltre la leucemia mieloide e linfoide vengono
distinte in:
–
–
croniche caratterizzate da cellule neoplastiche relativamente
mature e più simili alle corrispondenti cellule normali.
acute caratterizzate da cellule neoplastiche più immature
Cellula emopoietica primitiva in grado di
sostenere la produzione di tutte le linee cellulari
ematopietiche
EMOPOIESI
Erythrocyte
BFU-E
LEUCEMIE MIELOIDI
Erythroblast
Granulocyte
CFU-G
Myelocyte
Monocyte
CFU-M
LTC-IC:
Monoblast
Multipotent
Stem cells
Platelets
CFU-MK
LEUCEMIE LINFOIDI
Megakaryocyte
Lymphocyte
CFU-L
Proliferation
Apoptosis
Differentiation
MECCANISMI DI LESIONE GENICA NEI TUMORI EMOPOIETICI
Traslocazioni
t(9;22) in LMC
t(15;17) in LMA
t(9;22) in LLA
t(4;11) in LLA
11q23 in LLA LMA
Inv(16) in LMA
X
Delezioni cromosomiali parziali
5q- in LMA
6, 13, 11q- in LLC
Duplicazioni cromosomiali
Trisomia 12 in LLC
Mutazioni puntiformi
..AGCTCGG..
...AGTTCGG..
Attivazione RAS in LMA, LLA
Internal tandem duplication
Duplicazioni in tandem
nel gene FLT3
MECCANISMI MOLECOLARI DI
TRASFORMAZIONE NEOPLASTICA
1
3
2
1: fattori di crescita
2: recettori dei fattori di crescita
3: trasduttori del segnale
4: fattori di trascrizione
5: proteine regolatorie del ciclo
cellulare
4
5
Apoptosi
Differenziamento
Proliferazione
Alterazione di geni che
codificano:
Leucemia Mieloide Cronica (LMC)
Incidenza : 1–2 per 100.000
Mediana dell’età di insorgenza: 53 anni
Aumento di incidenza con l’età (30% dei
pazienti ha più di 60 anni
Unica lesione genetica: Cromosoma
philadelphia
Alla presentazione
50% diagnosticati attraverso analisi di
laboratorio
LMC: le 3 fasi del decorso clinico
Fase cronica
Durata media
4-6 anni
Fase avanzata
Accelerata
Fase blastica
Durata media
sino a 1 anno
Sopravvivenza media
3-6 mesi
Cromosoma Philadelphia:Traslocazione
t(9;22)
BCR
•
•
•
Breakpoint cluster region gene
Locus at 22q11
Codifica per la proteina bcr, di 160-kDa
proteina attivatore di GTPase (GAP)
ABL
•
•
Abelson’s murine leukemia viral oncogene homolog
Locus at 9q34
è una proteina con attività chinasica strettamente
regolata dall’interazione sterica tra domini di SH1 con
SH2 e SH3 e l’ATP
BCR-ABL
•
In BCR sono state identificate 3 differenti breakpoint cluster
regions
1. M-bcr
2. m-bcr
3. μ –bcr
m-bcr
M-bcr
μ-bcr
BCR-ABL
Punti di rottura
In ABL, i breakpoints di solito cadono in un’ampia regione che parte a
monte dell’esone 1b e finisce a valle dell’esone 1a.
•
a2
•
Il contributo di ABL all’mRNA ibrido è invariabile a
causa dello splicing dell’esone 1
La proteina di fusione Bcr-Abl
m-BCR
BCR
►e1a2
►b2a2
►b3a2
►e19a2
B
2
B
3
MBCR
m-BCR
A2
ABL
Tyrosine kinase
BCR-ABL
Tre diversi trascritti di fusione
e1a2
b2a2
b3a2
e19a2
BCR
►p190
kD
►p210
kD
►p230
kD
ABL
Contributo costante di ABL → Potere trasformante
Contributo variabile di BCR → Fenotipo della patologia
BCR-ABL
•
BCR incrementa l’attività kinasica di
ABL
Incremento della proliferazione
– Riduzione dell’apoptosi
– Riduzione della adesione delle cellule allo
stroma midollare
–
Bcr-Abl tirosina-chinasi: la proteina ‘chimerica’
che causa la trasformazione neoplastica
Proteina
Bcr-Abl
Substrato
PP
P
Y
ATP
P
Substrato
fosforilato
Y
Effettore
Y = Tirosina
P = Fosfato
Proteine tirosin-chinasi
Le tirosin-chinasi appartengono a diverse classi familiari,
soprattutto quelle di classe tre, sono costituite da 3
domini:
Dominio di attivazione
Dominio di legame all’ATP
Dominio catalitico
Sono proteine
finemente regolate,
quindi, quando vengono
fosforilate, inviano dei
segnali che raggiungono
il nucleo. Una volta che
la cellula ha ricevuto il
messaggio, la proteina
viene defosforilata ed il
segnale si spegne.
ATTIVAZIONE COSTITUTIVA DELLE VIE DI
TRASDUZIONE DEL SEGNALE
L’attività tirosin-chinasica
della proteina BCR-ABL
attiva numerose vie di
traduzione del segnale
che inducono:
Incremento della
migrazione dal
midollo osseo
Inibizione
morte
cellulare
Proliferazione
incontrollata
• Un incremento della
frazione proliferante;
• Una riduzione
dell’apoptosi;
• Riduzione dell’adesione
delle cellule allo
stroma midollare.
APPROCCIO DIAGNOSTICO ALLA LEUCEMIA
MIELOIDE CRONICA
ESEMPIO: PAZIENTE CON SOSPETTA
LEUCEMIA MIELOIDE CRONICA
INDAGINI DI I°LIVELLO
LMC in fase cronica: presentazione clinica
Asintomatica nel 50% dei casi
Sintomatologia clinica
• Astenia
• Febbricola
• Perdita di peso/anoressia
Obiettività clinica
Splenomegalia (50% dei casi)
DIAGNOSI DI LMC:
ESAMI DI LABORATORIO
Sangue periferico:Emocromo
FASI
CRONICA
LEUCOCITI
(granulociti)
BLASTI
PIASTRINE
Valori di riferimento
4.500
a 11.000 per
ACCELERATA
µL.
BLASTICA
20000/µL
0
>10%
30%
O NORMALI
Sangue periferico normale Sangue periferico LMC
Puntato midollare
Richiesta esami
Tipizzazione genetica leucemia mieloide cronica traslocazione t(9;22)
INDAGINI DI II LIVELLO
• Citogenetica Convenzionale
• Citogenetica Molecolare
– Fluorescence in-situ hybridization (FISH)
• Biologia Molecolare
– Diagnostica basata su PCR Qualitativa
– Monitoraggio basato su PCR Quantitativa
CITOGENETICA CONVENZIONALE
La citogenetica convenzionale è una tecnica
che permette lo studio del numero e della
struttura dei cromosomi (studio del cariotipo)
I cromosomi vengono esaminati “bloccati” in
metafase.
Step:
•prelievo del campione (sangue midollare)
•allestimento delle colture cellulari
•bandeggio dei cromosomi
CITOGENETICA
Citogenetica convenzionale
Limiti
•
•
•
•
Riarrangiamenti cromosomici
complessi
Cattiva morfologia cromosomica
Basso indice mitotico delle cellule
neoplastiche
Bassa crescita o fallimento della
coltura
INDAGINI DI II LIVELLO
• Citogenetica Convenzionale
• Citogenetica Molecolare
– Fluorescence in-situ hybridization (FISH)
• Genetica Molecolare
– Diagnostica basata su PCR Qualitativa
– Monitoraggio basato su PCR Quantitativa
CITOGENETICA MOLECOLARE (FISH)
La Fluorescent In Situ Hybridization
(FISH) è una tecnologia che utilizza
sonde nucleotidiche marcate (DNA
probes) per identificare specifiche
regioni di un cromosoma ovvero
determinate sequenze del DNA.
Step:
•denaturazione del DNA
•incubazione sonda + DNA denaturato
(“annealing”)
•rilevazione del segnale della sonda
con microscopio a fluorescenza
FISH: tappe della metodica
Sonda BCR-ABL utilizzata per la
Fish
FISH (fluorescence in situ hybridization)
INDAGINI DI II LIVELLO
• Citogenetica Convenzionale
• Citogenetica Molecolare
– Fluorescence in-situ hybridization (FISH)
• Biologia Molecolare
– Diagnostica basata su PCR Qualitativa
– Monitoraggio basato su PCR Quantitativa
La proteina di fusione Bcr-Abl
m-BCR
BCR
B
2
B
3
MBCR
m-BCR
A2
ABL
Tyrosine kinase
►e1a2
►b2a2
►b3a2
►e19a2
p210
METODOLOGIE D’INDAGINE: LA PCR
BCR
ABL
PCR qualitativa (t 9;22)
PCR qualitativa (t 9;22)
1
2
3
4
5
6
7
8
9 CN
CP
b2a2
b3a2
proteina p210
Controlli di qualità interni
Controllo di integrità dell’RNA: amplificazione di un gene
sempre espresso per validare la buona qualità dell’RNA
del campione da analizzare.
1 2 3 4 5 6 7 8 9 CN CP
Gene di controllo PBG (porfobilinogeno
deaminasi)
1
2
3
4
5
6
7
Controlli di qualità interni: controlli
positivi
Controllo positivo: amplificazione dell’mRNA della linea cellulare
caratterizzata geneticamente dal gene di fusione d’interesse.
K562
1
2
3
4
5
6
7
CP
Controlli di qualità interni:controli negativi
Controlli negativi procedura: amplificazione dell’mRNA derivante da cellule
mononucleate di soggetti non affetti (NAC:no amplification control).
Controlli negativi PCR: amplificazione di un’aliquota di acqua distillata. NTC: (no
template control).
NAC:no amplification control
Contaminazione dei campioni o dei reagenti
NAC NTC
NAC NTC
NTC:no template control
CP
VARIABILI PRE-ANALITICHE
VOLUME SANGUE:
DX: 10 ML in EDTA SANGUE PERIFERICO
F.UP: 20 mL in EDTA SANGUE PERIFERICO
TEMPI DI CONSEGNA: in giornata/max entro le 24 ore
TERAPIA
LMC è una malattia devastante con un prognosi terribile per molti
pazienti
Fino a pochi anni fa le opzioni terapeutiche erano poche e il goal
terapeutico rimaneva solo il ritardo della progressione di malattia
Anni 20
Anni 50
1920 viene introdotto
il primo trattamento
per la LMC con scarsi
risultati: Radiation
Anni 70
Anni 80
Anni 90
1970:approccio al trapianto:
cure in termini di remissione a
lungo termine delle cellule
cancerose
1950 e 1960 è il periodo
degli agenti
chemioterapici: aumento
survival rate fino a 5 anni
È mirato e blocca la
principale causa della
leucemia
11980 debutta l’IFNalfa con poco
successo
TKIs Have Changed the Way CML Is Monitored
The Treatment Milestones in Ph+ CML Continue to Evolve: Earlier and Deeper
1960
1970
1980
BUSULFAN
Goal of
Therapy
CHR1
1990
IFN
CCyR2
2000
Today
IMATINIB
NILOTINIB
MMR3
Deeper
molecular
responses4
Leukemic
Reduction5,a
≤ 10% ≈ 1-log
≤ 1% ≈ 2-log
≤ 0.1%IS ≈ 3-log
≤ 0.0032%IS ≈ 4.5-log
Leukemic Burden
1. Cortes JE, et al. Am J Med. 1996;100(5):555-570.
2. Rosti G, et al. Semin Hematol. 2003;40(2 suppl 2):56-61.
3. Deininger M, et al. Blood. 2005;105(7):2640-2653.
4. Saglio G, et al. N Engl J Med. 2010;362(24):2251-2259.
5. Kantarjian HM, et al. Blood. 2011;117(4):1141-1145.
6. Radich JP. Clin Lymphoma Myeloma. 2009;9(suppl 4):S391-S394.
a
7. Hughes TP, et al. Blood. 2010;116(19):3758-3765.
8. Press RD, et al. Clin Cancer Res. 2007;13(20):6136-6143.
MR4.5 and beyond
Compared with baseline levels.
IS, international scale; MCyR, major cytogenetic response; MR4,
molecular response ≥ 4-log reduction; MR4.5, molecular response
≥ 4.5-log reduction; TKI, tyrosine kinase inhibitor.