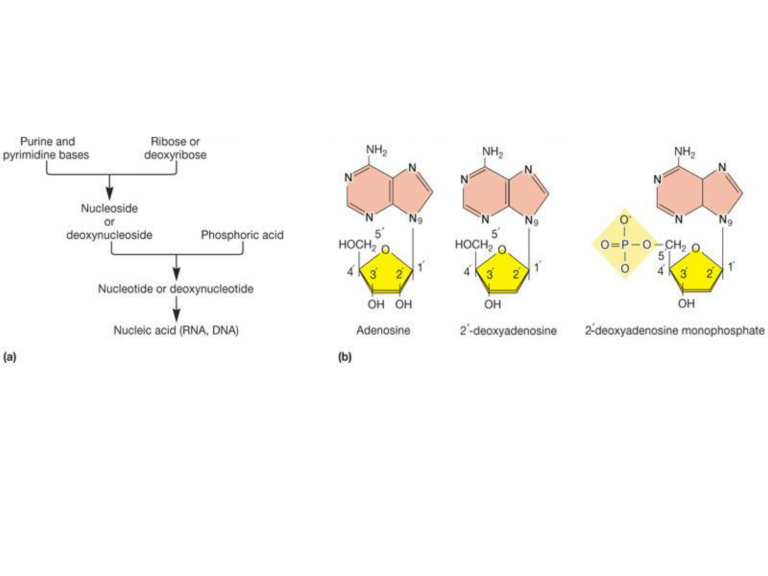
Caratteristiche del DNA
• Macromolecola, polimero di deossiribonucleotidi
• 4 nucleotidi: adenosina, citosina,
guanosina, timidina
• Appaiamento A-T e G-C (Regola della
complementarietà o di Chargaff)
• Struttura a doppia elica
Appaiamento G-C ed A-T
il “CROMOSOMA BATTERICO” (nucleoide)
• Il DNA di un microrganismo è concentrato in
un’unica molecola, il cromosoma batterico
(alcuni batteri possono avere 2 cromosomi)
- dimensioni tipiche: da 3 a 5.000.000 bp, (1.7 mm)
• il cromosoma è circolare (ma alcuni importanti
gruppi hanno un cromosoma lineare)
• Il DNA è sempre associato a proteine, ma non è
altamente organizzato come negli eucarioti
DNA batterico – domini superavvolti
La replicazione del DNA è detta
semiconservativa
La duplicazione del DNA da parte del suo
specifico macchinario enzimatico è detta
replicazione
Nel microrganismo, la replicazione del
DNA è associata alla sua riproduzione,
che avviene per scissione binaria.
La replicazione del DNA è
semiconservativa: cioè ognuna delle due
eliche complementari di una molecola di
DNA servirà da “stampo” per una nuova
elica.
La replicazione del DNA dipende
dalle DNA polimerasi
La sintesi della nuova elica di DNA procede in
direzione 5‘-3‘
Viene catalizzata dalle DNA polimerasi (almeno 3
enzimi differenti con questa funzione in E. coli)
Le DNA polimerasi sono solo parte di un
macchinario replicativo molto complesso che
coinvolge numerosi altri enzimi e proteine (DNA
girasi, elicasi, SSB protein, etc.)
Richiedono dNTP‘s come precursori della sintesi
della macromolecola, un‘elica di DNA „stampo“ e
un „primer“ (RNA) da cui può partire la sintesi
della nuova elica
Problemi legati alla replicazione in
direzione 5‘-3‘
„Frammenti di Okazaki“
I PLASMIDI
• Molti batteri oltre al cromosoma contengono molecole di
DNA più piccole (da 1-2,000 a 100-500,000 pdb), dette
plasmidi.
• Queste molecole non sono indispensabili per le funzioni
fondamentali del batterio (ceppi della stessa specie possono
esserne privi).
• I plasmidi sono generalmente molecole di DNA circolare e
superavvolto.
• I plasmidi replicano indipendentemente dal cromosoma del
batterio, anche se necessitano del medesimo “macchinario
enzimatico” del cromosoma perché avvenga la loro
replicazione.
Funzioni codificate dai plasmidi
• I plasmidi possono codificare per diverse funzioni che
conferiscono al batterio nuove proprietà:
• RESISTENZA AD ANTIBIOTICI E, SOSTANZE TOSSICHE,
RADIAZIONI…
• CAPACITA’ DI PRODURRE ANTIBIOTICI, TOSSINE,
FATTORI DI VIRULENZA…
• NUOVE CAPACITA’ METABOLICHE
•…
Ma tornando al cromosoma
batterico……..
Perché è così importante il DNA?
Perché contiene l’informazione genetica…..
Un gene da un punto di vista “genetico”:
GENE= unità fondamentale dell’informazione
genetica
Tipicamente un gene corrisponde ad una
proteina
(o ad un RNA con funzione specifica)
Un gene da un punto di vista “biochimico”:
Sequenze di DNA che, una volta trascritte e
tradotte contengono l’informazione minima
necessaria per la produzione di una proteina
specifica
Il genoma batterico medio
Genoma=insieme dei geni di un
organismo
Un batterio come Escherichia coli
possiede un genoma di 4.300.000 bp
circa che codifica per circa 4300 geni
1 gene medio=1000 nucleotidi
(regioni non-codificanti escluse)
Sequenziamento genomico
• Il DNA contiene l’informazione genica di
un organismo, contenuta nelle sue
specifiche sequenze nucleotidiche.
• Conoscere la sequenza di un genoma
(l’insieme dei geni di un organismo) ci
permette di avere importanti informazioni
sulla sua biologia.
• A tutt’oggi sono state rese disponibili le
sequenze di circa 500 microrganismi.
Interpretazione delle sequenze
di DNA
5’
3’
3’
3’
5’
3’
5’
AATAAAAATTTAACTCAATTTGTATCAAAAAATAACAGAAATCTAGCAGTTTTTGTAT
TTTATTTTTAAATTGAGTTAAACATAGTTTTTTATTGTCTTTAGATCGTCAAAAACATA
3’
5’
5’
3’
TTGCTGCTGGTGCTGCAATGGCTGATGAAGCTGTTGTTCATGACAGTTATGCATTCG
AACGACGACCACGACGTTACCGACTACTTCGACAACAAGTACTGTCAATACGTAAGC
3’
Sequenze codificanti e noncodificanti nel genoma batterico
= 1000 bp
Geni:
CDS= Coding Sequences
ORF= Open Reading Frames
N.B.: nei microrganismi le sequenze geniche non presentano interruzioni
(es. da sequenze introniche)
Che cosa ci permette di identificare le sequenze
codificanti (cioè i geni) da una sequenza di DNA?
Risposta:
Dalla nostra conoscenza
del codice genetico!
AUG o GUG (ATG e GTG nel
DNA) sono il codone di inizio
per la sintesi proteica
UAA (TAA), UAG (TAG) e
UGA (TGA) sono i codoni di
stop
Una “Open Reading Frame”, cioè una “lunga” sequenza di
codoni in frame rappresenta un potenziale gene
921 paia di basi
GTG CGA ATA AAT TTC GCA CAA……………………........GGC TAC TAA
Met Arg
Ile Asn Phe Ala Gln……………………………Gly
307 amino acidi
Tyr STOP
Operoni: gruppi di geni parte di una
unica unità trascrizionale
L’organizzazione di geni in operoni è tipica dei Bacteria.
Il numero di geni presente in un operone è variabile (2-15 geni)
Generalmente i geni di un operone codificano per proteine con
funzioni correlate tra loro (es. enzimi di una stessa via
metabolica)
Il flusso dell’informazione:
l’espressione genica
La trascrizione
L’espressione genica: la “messa in atto”
dell’informazione contenuta nel DNA
Quali sequenze di DNA vengono
trascritte?
• A) tutto il DNA
• B) le sequenze codificanti geni
• C) le sequenze codificanti ed una parte delle
sequenze non codificanti
(quali? Dove comincia e dove finisce la
trascrizione?)
I „confini“ di un gene (coding
sequence) sono molto chiari…
• Un gene è identificabile dalla presenza di un
codone di inizio (ATG, più raramente GTG) che
dà inizio ad una sequenza codificante più o
meno lunga, che termina con un segnale di stop
(TAA, TAG o TGA).
• Il gene fa parte di un „trascritto“ (una molecola di
RNA) che parte da un promotore e finisce ad
un terminatore (il trascritto può comprendere
più geni che appartengono allo stesso operone)
• L‘enzima responsabile del processo di
trascrizione prende il nome di RNA polimerasi
L‘RNA polimerasi è un enzima complesso e
composto da diverse subunità
a
b
w
b‘
s
L‘RNA polimerasi è un enzima complesso e
composto da diverse subunità
b
w
b‘
a
s
Il complesso a2bb‘(w) possiede l‘attività
catalitica per la sintesi dell‘RNA da una
molecola stampo di DNA e viene definito
„core enzyme“
Non è però in grado di legare i promotori
(siti di inizio della trascrizione) in
assenza della subunità (o fattore) s.
L‘assemblaggio di questa subunità porta
alla formazione del cosiddetto
oloenzima
I tre momenti base del processo di
trascrizione
1: Denaturazione
locale del DNA
2: Inizio della
trascrizione (pol.
di 20 nt di RNA
circa)
3: Allungamento
della molecola di
RNA nascente
La trascrizione è un processo
„asimmetrico“
Template strand/elica stampo
b
w
b‘
a
Coding strand
Il core enzyme è in grado di legare il
DNA ma non di iniziare la trascrizione
b
w
b‘
Complesso chiuso
a
b
w
s
b‘
a
Complesso aperto
A sua volta, l‘inizio della trascrizione si
compone di diverse reazioni
Fattore s
Core Enzyme
Alcuni antibiotici inibiscono l‘RNA
polimerasi (es. Rifampicina)
Antibiotico scoperto in Italia negli anni
60
Fondamentale per capire meccanismo di
trascrizione (ne inibisce l‘inizio)
b
w
s
b‘
a
Utilizzato per gran parte delle infezioni
batteriche („Rifocin“) fino a metà anni
70
Abbandonato per alta frequenza di
batteri resistenti
A tutt‘oggi principale antibiotico nella
cura della tubercolosi
Il sito di inizio della trascrizione:
il promotore
Il „mappaggio“ dei promotori
Il “Promotore perfetto” (?)
UP element
-35
Ext.
D +1
-10
AAATAAAATTTTTAAn..nTTGACAnnn…nnnTGnTATAATnnattAn
4-6nt
14nt
4-6nt
17nt
Riconosciuto dalla subunità a
Riconosciuti dalla subunità s
Come si ferma una macchina in
corsa? Il terminatore
Terminazione “intrinseca”
(rho-indipendente)
Terminazione dipendente dal
fattore Rho (r)
Operoni: gruppi di geni parte di una
unica unità trascrizionale
L’organizzazione di geni in operoni è tipica dei Bacteria.
Il numero di geni presente in un operone è variabile (2-15 geni)
Generalmente i geni di un operone codificano per proteine con
funzioni correlate tra loro (es. enzimi di una stessa via
metabolica)
Operoni: gruppi di geni parte di una unica
unità trascrizionale e controllati da un
unico promotore
= presenza di un promotore
La trascrizione negli Archaea
• Somiglianze con eucarioti
• Maggiore complessità dell’RNA polimerasi
(circa 12 subunità assemblate in modi
alternativi)
– introni presenti in alcuni geni
– Insensibilità a rifampicina e ad altri antibiotici che
inibiscono la trascrizione dei batteri
• Ma anche…..somiglianze con i procarioti
– mRNA policistronico










![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

