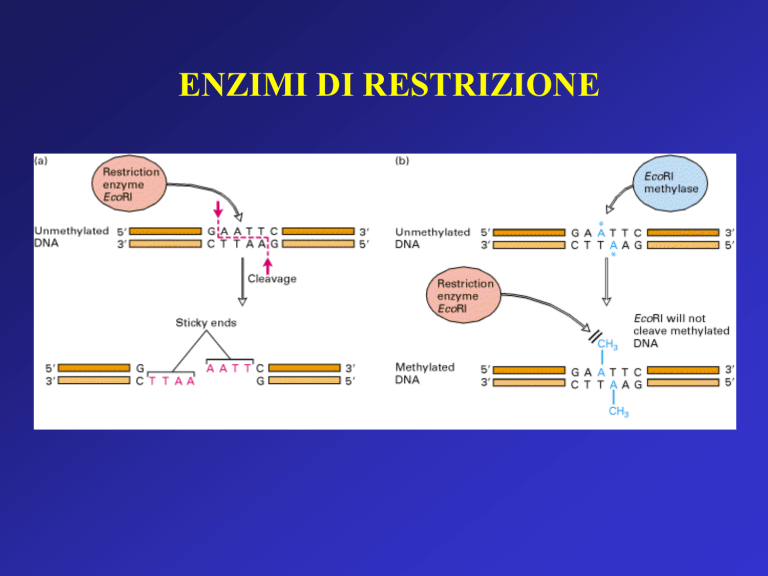
ENZIMI DI RESTRIZIONE
Reazione
ligasica di
frammenti di
restrizione
DNA RICOMBINANTE: DUE MOLECOLE DI DNA VENGONO
UNITE IN PROVETTA SFRUTTANDO LE PROPRIETA’ DI
ENDONUCLEASI DI RESTRIZIONE DI TIPO II E DELLA DNA
LIGASI
CLONAGGIO
-IN BIOLOGIA CLONARE UN ORGANISMO SIGNIFICA OTTENERE UN
INDIVIDUO UNA POPOLAZIONE DI ORGANISMI CHE SONO
GENETICAMENTE IDENTICI
-IN BIOLOGIA MOLECOLARE CLONARE UN TRATTO DI DNA SIGNIFICA
OTTENERE UNA POPOLAZIONE DI DNA IDENTICHE ALLA MOLECOLA
DI PARTENZA
Fig 4.20 separation of poly-A
mRNA
mRNA Purification
1. Total RNA Purification
2. PolyA+ RNA purification using
oligo(dT)
Gubler Hoffman cDNA Synthesis - 1
mRNA
primed mRNA
AAAAAn
Anneal oligo dT primer
AAAAAn
TTTTT
Reverse Transcriptase
and dNTPs
mRNA/cDNA
hybrid
AAAAAn
TTTTT
Gubler Hoffman cDNA Synthesis - 2
mRNA/cDNA
hybrid
AAAAAn
TTTTT
RNase H
nicked RNA
AAAAAn
TTTTT
DNA Pol I
nicked RNA used
as primers by Pol
AAAAA
TTTTT
Gubler Hoffman cDNA Synthesis - 3
2nd strand cDNA
in pieces
AAAAA
TTTTT
E. coli DNA
Ligase
AAAAA
TTTTT
ds cDNA
Clone into vector
cDNA Library
cDNA synthesis
TTTTTTTTT
First strand
Nick translation
cDNA library
Vettori e clonaggio
Vettori
Plasmidi
Fagi
Cosmidi
YAC
Vettori e clonaggio
Plasmide
Molecola circolare di DNA a doppio
filamento, normalmente presente
nella cellula batterica, in grado
di replicarsi autonomamente
dal cromosoma.
VETTORI DI CLONAGGIO
Vettori e clonaggio
Replicazione di un plasmide
INSERIMENTO DI UN TRATTO DI DNA ESOGENO
IN UN VETTORE DI CLONAGGIO
CLONAGGIO:
-TRASFORMAZIONE
-SELEZIONE
-CRESCITA COLONIE
IDENTIFICAZIONE
DELLA COLONIA
BATTERICA DI INTERESSE
Bromo-chloro-indoyl--galactopyranosidase or X-Gal (Clear)
-galactosidase
Bromo-chloro-indoyl (Deep blue insoluble)
+
galactose
Inserting chromosomal DNA into a vector
Chromosome
GAATTC
CTTAAG
Vector
GAATTC
CTTAAG
GAATTC
CTTAAG
Cut with EcoRI and add DNA ligase
Recombinant vector
GAATTC
CTTAAG
GAATTC
CTTAAG
Ampicillin resistant; -galactosidase negative (White on X-Gal)
LacZ gene codes for -galactosidase
Ampicillin resistance gene
Bacteria from ligation plated
on ampicillin and X-Gal
Contains
wild type
plasmd
Contains
recombinant
plasmd
Vettori e clonaggio
Vettori e clonaggio
Lunghezza massima del
DNA clonabile in un plasmide:
circa 20 Kb
Amino acid sequence
Met-Asn-Lys-Trp-Glu-Met
Met = ATG; Asn = AAT or AAC; Lys = AAA or AAG;
Trp = TGG; Glu = GAA or GAG
How many probes must we make?
ATG AAT AAA TGG GAA ATG
ATG AAT AAA TGG GAG ATG
ATG AAT AAG TGG GAA ATG
ATG AAT AAG TGG GAG ATG
ATG AAC AAA TGG GAA ATG
ATG AAC AAA TGG GAG ATG
ATG AAC AAG TGG GAA ATG
ATG AAC AAG TGG GAG ATG
Screening the Library
ATTAGCGCCTTTACGCA
……………………TAATCGCGGAAATGCGT…………
Il metodo Sanger
(o metodo a terminazione di catena)
1) Il sequenziamento a terminazione di catena coinvolge la sintesi di
nuovi filamenti di DNA, complementari ad uno stampo a singolo
filamento
Per la sintesi del DNA si utilizzano DNA polimerasi caratterizzate da
•Elevata processività
•Bassa attività esonucleasica 5’-3’
•Bassa attività esonucleasica 3’-5’
(es. Klenow, Sequenasi)
(1) Produzione dello stampo a filamento singolo
Utilizzo
della PCR
Utilizzo di un fago helper
Per M13 e produzione della
Forma a singolo filamento
Utilizzo di
fagemidi
Denaturazione del vettore
Il metodo Sanger
(o metodo a terminazione di catena)
Nel sequenziamento a terminazione di catena la reazione oltre ad
1) Uno stampo a singola elica
ha bisogno di:
2) un innesco specifico (primer)
3) La marcatura con un dNTP* marcato, di solito con
35S
(2,3) Sintesi del filamento marcato
PRIMER
DNA Polimerasi
5’-A T C T T T T A G A GT A C C
3’-T A G A A A A T C T C A T G G A C T C T C T A C T A T C T A C A T G T A -5’
PRIMER
5’-A T C T T T T A G A GT A C C T G AG*AGAT GA T AG*A DNA Polimerasi
3’-T A G A A A A T C T C A T G G A C T C T C T A C T A T C T A C A T G T A -5’
Il metodo Sanger
(o metodo a terminazione di catena)
Nel sequenziamento a terminazione di catena la reazione oltre ad
1) Uno stampo a singola elica
2) un innesco specifico (primer)
3) La marcatura con un dNTP* marcato, di solito con
35S
ha bisogno di:
4) una miscela di ddNTP, uno per ogni reazione di sequenza
Terminazione della catena
La sintesi del filamento compementare, tuttavia non prosegue
indefinitivamente, perché la miscela di reazione contiene, in quattro
distinte reazioni piccole quantità di specifici dideossinucleotidi
trifosfati, ddATP, ddTTP, ddCTP e ddGTP, che bloccano l’allungamento perché
posseggono un solo atomo di idrogeno al posto del gruppo -OH in 3’
PRIMER
5’-A T C T T T T A G A GT A C C T G AG*AGAT GA T AG*A DNA Polimerasi
3’-T A G A A A A T C T C A T G G A C T C T C T A C T A T C T A C A T G T A -5’
+ ddNTP ( per es. ddCTP)
STOP
5’-A T C T T T T A G A GT A C C T G AG*AGAT GA T AG*AT G T AddC
3’-T A G A A A A T C T C A T G G A C T C T C T A C T A T C T A C A T G T A -5’
Il risultato è una serie di frammenti interrotti ciascuno
in corrispondenza di ogni dCTP
ddCTP
ddCTP
ddCTP
ddCTP
ddCTP
ddCTP
Schema di sequenziamento a terminazione di catena
DNA stampo a
singola elica
3’-GGCTAAC
5’
Ibridazione con
Il primer
3’
3’-GGCTAAC
+
[35S]dATP+dCTP,dGTP,dTT (dNTP) +Sequenasi
ddATP, dNTP
ddCTP, dNTP
-CCG ddA
-ddC
-C ddC
A C
ddGTP, dNTP
ddTTP, dNTP
-CC ddG
-CCGATT ddG
G
-CCGA ddT
-CCGAT ddT
T
-CCGATT ddG
-CCGAT ddT
-CCGA ddT
-CCG ddA
-CC ddG
-C ddC
-ddC
G
T
T
A
G
C
C
Sequenza: 5’-CCGATTG
Direzione di
lettura
Nuovi metodi di sequenziamento
Il sequenziamento a ciclo termico ( PCR asimmetrica)
Il sequenziamento automatizzato con marcatori fluorescenti
Il sequenziamento a ciclo termico ( PCR asimettrica)
DNA stampo
PCR con un solo primer
ddATP
ddA
ddA
ddA
ddA
ddA
Vengono effettuate 4 rezioni di PCR, ognuna contiene un solo primer e uno dei 4
dideossi nucleotidi trifosfati. Si generano le consuete famiglie di filamenti a catena
terminata che vengono risolti per elettroforesi su gel di poliacrilammide
Il sequenziamento automatizzato con marcatori fluorescenti
Coniugando a ciascun ddNTP
un diverso marcatore fluorescente, è
possibile effettuare le quattro reazioni di
sequenziamento in un unico tubo da saggio
e caricare il tutto in solo pozzetto di gel
ddA
ddT
ddC
ddG
Le emissioni fluorescenti vengono captate da un rilevatore e le
informazioni vengono integrate e trasformate in picchi di colore
diverso, con aree proporzionali all’intensità di emissione.
Fluorescent DNA Sequencing
AGTAC T G G GAT C
Gel
Electrophoresis
Detection of Fluorescently Tagged
DNA
DNA Fragments
Separated by
Electrophoresis
Optical
Detection System
Scanning Laser
Excites
Fluorescent Dyes
Output to Computer
Fluorescent DNA Sequencing
Data
Fluorescent DNA Sequence Trace


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)






![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)

