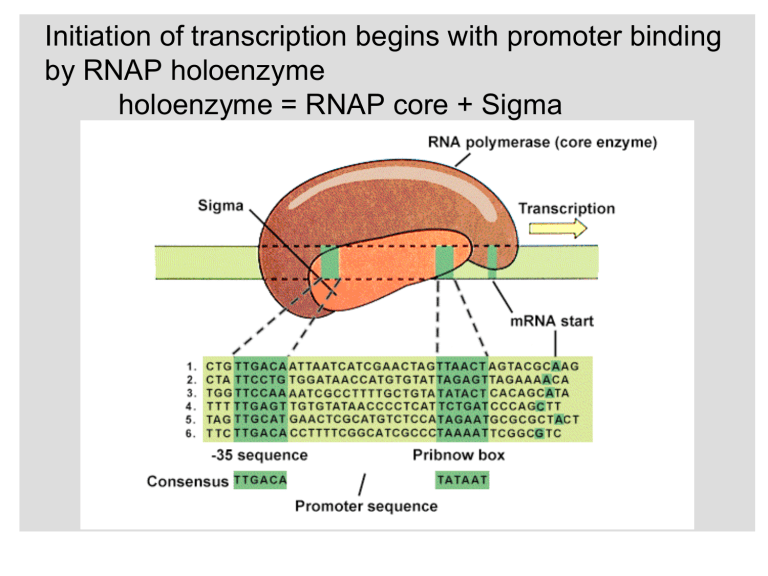
Initiation of transcription begins with promoter binding
by RNAP holoenzyme
holoenzyme = RNAP core + Sigma
Brock Biology of Microorganisms, vol. 9, Chapter 6
Controllo della Trascrizione
NTPs
k1
k2
RPC
R+P
k-1
k3
RPO
k-2
k4
RPI
RPE
k-3
Trascritti abortiti
Costante di
equilibrio: KI
KI = RPC/(R + P)
La velocità di
formazione del
complesso aperto è
k2
Questa
transizione è
la “promoter
clearance”con velocità
k4
Il problema del riconoscimento del DNA
• Il genoma umano contiene 3 · 109 coppie di
basi (bp)
• Le proteine che legano il DNA devono
individuare siti di azioni UNICI nel genoma
–
–
–
–
Promotori
Origine della Replicazione
Posizioni dove il DNA è danneggiato
…
Composizione del DNA
•
•
•
•
•
Basi Nucleotidiche
Deossiribosio
Fosfati
Acqua (36-46 molecole per bp)
Ioni / Leganti
– 0.75 ioni monovalenti/PO4
Nei genomi più sempici
• Procarioti (p.es. E. coli) 5 · 106 bp
– 3-4 ordini di grandezza più semplice del genoma
umano o delle piante
• L’evoluzione ha prodotto proteine che
riconoscono siti di 8-10 basi
– La frequenza casuale è 2·10-5 - 1·10-6
– Alta selettività
Cosa è riconosciuto?
• La struttura MEDIA del DNA
• Due catene polianioniche con solchi
di geometria diversa
– Major groove: ampio, piatto
– Minor: stretto, profondo
• Contraioni e idratazione
• Flessibilità (dinamica)
Gruppi funzionali delle basi
• Accettori/donatori di legami a idrogeno
• Superfici di interazione di van der Waals
Gruppi funzionali del backbone
• Ripetuti – ma non equivalenti
• Gruppi fosfato – controioni
– Il DNA ha alta densità di carica
• Contraioni: K+, Na+, Mg++, poliammidi (p.es.
spermidina)
– L’identità dei controioni dipende dalla
sequenza e della cellula
• Deossiribosio
– Diversità conformazionale
– Distingue ssRNA dal ssDNA
Le proteine riconoscono il DNA generalmente nel
major groove
Esempio di riconoscimento nel major groove
Proteine zinc finger, tipo CCHH.
•Alcuni monomeri, altri dimeri.
•Spesso sono presenti più di un
finger e quindi la superficie di
riconoscimento può essere ampia.
•Il riconoscimento delle basi è dato
dalle catene laterali degli a.a.
Homeotic selector gene
Regolatori batterici
Helix turn helix.
•Molti regolatori procariotici sono del tipo helix turn helix.
•Legano come dimeri.
•Il riconoscimento delle basi è dato dalle catene laterali degli a.a.
che fuoriescono dalle eliche
Leucine zipper protein.
•Dimero coiled-coil contenente
leucine ogni 7 amino acidi e un
residuo idrofobico ogni 3-4 a.a.
Helix-loop-helix protein.
•Diverso da helix-turn-helix!
Vantaggi della dimerizzazione:
• La formazione di diversi eterodimeri consente di riconoscere
un numero maggiore di sequenze di basi a parità di proteine
• Il riconoscimento può essere represso tramite la formazione di
eterodimeri in cui una subunità è priva del dominio di legame
del DNA
• I leganti possono causare cambiamenti allosterici che alterano
la posizione reciproca dei domini che riconoscono il DNA
• Modificazioni post-traduzionali come la fosforilazione e
l’acetilazione possono promuovere o inibire la dimerizzazione e
quindi determinare il legame
Il legame come omo o eterodimeri
aumenta il reportorio di siti
riconosciuti
Il legame di piccole molecole
determina il riposizionamento dei
domini che legano il DNA, in modo da
allinearli con il solco maggiore
Partner di eterodimerizzazione privi del
dominio di legame del DNA fungono da
repressori
promotori procariotici
La trascrizione nei batteri è generalmente sottoposta ad un controllo di tipo negativo in
cui la RNA polimerasi oloenzima (associata al fattore sigma) si lega direttamente al
promotore, dirigendo la trascrizione del gene immediatamente a valle a meno che venga
repressa dalla presenza di un repressore.
Il promotore è la regione di DNA al 5’ di un gene, dove si lega la RNA polimerasi.
L’analisi della sequenza dei primi promotori non rivelò, come atteso, la stessa sequenza
ma regioni molto eterogenee all’interno delle quali si riconoscevano due sequenze
conservate una a -10, con consensus TAATAT e un altra a -35 con consensus TTCAGA.
Studi più recenti hanno caratterizzato un’altra regione regolativa, detta UP
Situata ancora più a monte.
Promotore procariotico
+1
ACCATG TTCAGA AGCATTCTCAACTGGTATAGTTAACTATAAT
-35
CTGGATTC
-10
Studi di footprinting e analisi di mutazioni hanno confermato l’importanza funzionale di
queste regioni regolative.
Meccanismo di repressione o co-repressione
P.es.: arginina, uno dei 20 amino acidi essenziali
I batteri possono produrre arginina, ma se ne trovano nell’ambiente non
esprimono i corrispondenti geni per la biosintesi
Niente arginina
La cellula deve produrne
Il repressore non lega
Geni x biosintesi espressi
Alta [] di arginina
Non c’è bisogno di produrne
Il complesso repressore + Arg lega
I geni x biosintesi non sono espressi
Arginine
Meccanismo di repressione inducibile
P.es.: lattosio
I batteri usano il lattosio come fonte di carbonio se presente, ma altrimenti
non c’è bisogno di esprimere i geni per l’uso
Niente lattosio
Il repressore lega all’operatore
dell’operatore lac
Nessun enzima è espresso
In presenza di lattosio
La cellula lo usa come nutriente
Il complesso repressore+lattosio si
stacca dal DNA
Gli enzimi lac sono espressi
Lactose
Promoters regulated by alternate sigma factors
I promotori regolati da fattori sigma alternativi
may have completely different consensus
possono avere sequenze consenso diverse
sequences
70
TTGACA – 17 bp – TATAATN3-6-A
-35
-10
32
CTTGAAA – 16 bp – CCCCATNTN3-10-T/A
-35
-10
54 GG – N12 – GC/T – 12bp – A
-24
-12
Most factors are in the 70 family and hence
function like 70
54 is different – must be activated by activator proteins
after binding – kII is affected – RPO is formed
Gralla, 2000, J. Bacteriol. 182:4129-4136
Fig. 1. EM analysis of the PspF(1-275)-ADP.AlFx-{sigma}54 complex
54
M. Rappas et al., Science 307, 1972 -1975 (2005)
Published by AAAS
Meccanismo di attivazione della trascrizione
P.es.: maltosio
I batteri usano maltosio se presenti, ma non esprimono i corrispondenti
geni se il maltosio non è presente.
(Cfr. lattosio)
Niente maltosio
L’attivatore non lega il DNA
Nessun enzima è espresso
In presenza di maltosio
La cellula lo usa come nutriente
Il complesso attivatore+maltosio lega il DNA
Gli enzimi mal sono espressi
Promotore debole
Maltosio
ATTIVATORI TRASCRIZIONALI
Class I – upstream of –35
Class II – overlaps with –35
αNTD: amino-terminal domain of the RNAP α subunit
αCTD: carboxy-terminal domain of the RNAP α subunit
Barnard A. et al., Current Opinion in Microbiology Volume 7, 2004, pp.102-108
La stessa proteina può fare da attivatore e repressore
Multipartite
repression loop
Class I transcription
activator
Schleif, 1996, Chapter 83, Escherichia coli and Salmonella, ASM Press
1. Forte repressione in assenza di arabinosio e presenza di
glucosio
2. Forte attivazione in presenza di arabinosio
1 operone
2 operoni
Regolazione (negativa) della degradazione del bifenile in
Pseudomonas KKS102
HOPDA
Gli enzimi sono espressi debolmente in assenza di bifenile
Gene, Volume 256, 2000, Pages 223-228 Y. Ohtsubo et al.
Regolazione della degradazione del bifenile in P. pseudoalcaligenes KF707:
due regolatori
1
2
La regolazione di BphR2 non è nota
Saggi per la caratterizzazione delle proteine che
legano sequenze promotrici
?
TATA
5’
-35
trascrizione
A
AA
TA T G
TATA box
1. DNA mobility shift assays
2. DNase I footprinting assays
3’
Saggio di ritardo elettroforetico
(EMSA)
Se si sospetta e si vuole confermare la presenza di una regione di legame al DNA uno
dei metodi più immediati è quello noto come gel shift o EMSA (electrophoretic
mobility shift assay) o bandshift o gel di ritardo elettroforetico.
E’ una tecnica relativamente semplice ma con un potere
di risoluzione modesto. Consiste essenzialmente nella
preparazione di un estratto nucleare che viene mescolato
in vitro, in appropriate condizioni sperimentali, con un
frammento marcato contenente la regione di legame
putativo.
Se una o più proteine dell’estratto si legano al frammento
di DNA marcato, la sua mobilità elettroforetica in un gel
di poliacrilammide o agarosio diminuirà, come se il
frammento fosse rallentato. Il ritardo elettroforetico può
essere facilmente evidenziato comparando la mobilità
elettroforetica di un frammento di controllo senza estratto
proteico.
1. DNA mobility shift assays (gel-shift assays)
Successive column fractions
Protein fractions separated by column
chromatography assayed for ability to
bind to radiolabeled DNA fragment containing
a known regulatory element
Electrophoretic Mobility Shift Assay
- “EMSA”, “gel-shift”.
TFII E
-
-
-
-
-
-
+
Pol II
-
-
-
-
-
+
+
TFII F
-
-
-
-
+
+
+
TFII B
-
-
-
+
+
+
+
TFII A
-
-
+
+
+
+
+
TFII D
-
+
+
+
+
+
+
A
TFIID
A
6
A
5
Complessi
proteine-DNA
TFIID
TFIID
4
3
promotore
libero
TFIIB
F
Pol II
TFIIB
F
TFIIB
A
TFIID
2
1
TFIIB
F
Pol II
A
TFIID
TFIID
E
DNA footprinting
Il DNA footprinting è una tecnica che riesce a determinare l’esatta posizione della
proteina legata. Un frammento di DNA, supposto contenere una regione di legame,
viene marcata terminalmente, fatta interagire con la proteina in esame, e digerita
parzialmente con la DNasi I. L’eventuale legame alla proteina proteggerà la regione di
legame dalla degradazione endonucleasica.
Marcatura
terminale
5’*
5’*
5’*
5’*
Aggiungere il
fattore di trascrizione
5’*
5’*
5’*
Digestione parziale
con la DNAsi I
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
5’*
nessun taglio in
questa regione
DNase I Footprinting
Pancreatic DNase I is a relatively nonspecific endonuclease.
Regione - 10
-10
-20
-30
Regione - 35
-40
T
A
T
A
C
G
C
T
G
A
T
T
G
A
T
T
C
G
G
A
C
C
C
C
A
C
G
G
A
T
T
Footprinting del
promotore UV5:
La RNA polimerasi
protegge una regione
di circa 50-60 bp
contattando le regioni
-10 e -35
Figure 34-53a Identification of the Sp1-binding sites on the SV40
early promoter (Fig. 34-52, top).
two-component regulatory system:
signal transduction:
• sensor kinase autophosphorylates in
response to signal
• response regulator
phosphorylated in turn
• phosphorylated
regulator acts (e.g.,
DNA-binding repressor
or activator)
• phosphatase activity
removes PO4
numerous two-component regulatory systems are known:
Quorum sensing:
• Un tipo di sistema di controllo che consente la regolazione
dell’espressione genica in risposta alla densità della
popolazione
Il “segnale” = presenza di altri microorganismi
• Molecole segnale diffusibili (e.g., acylated homoserine
lactone, AHL) possono essere prodotte ed escrete da tutte
le cellule in una popolazione
• Una maggior densità della popolazione risulta in una
maggiore concentrazione di AHL
• Le molecole di AHL attivano specifici attivatori trascrizionali
→ risulta nell’induzione di trascrizione specifica
bioluminescent Vibrio fischeri
• photo taken in dark room; light generated solely by
luciferase-expressing bacteria growing on the streak plate
Vibrio bioluminescence:
• Vibrio fischeri inhabits
the open sea (in low
densities), as well as
the light organs of
squid (in high
densities)
• luminescence occurs
only at high bacteria
cell concentrations
quorum-sensing model
Bioresource technology, 2013


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)






![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)
