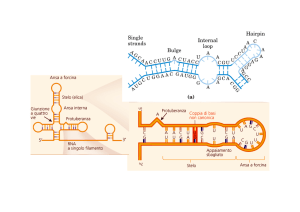
Organi, tessuti ecc.
STRUTTURA
FUNZIONE
EVOLUZIONE
Geni o segmenti genomici
STRUTTURA
(FUNZIONE)
EVOLUZIONE
Tempo
Gene ancestrale
Duplicazione
Diversificazione
Paraloghi
Linea di discendenza 1 (specie 1)
Linea di discendenza 2 (specie 2)
Ortologhi
Paraloghi
Ortologhi
Paraloghi
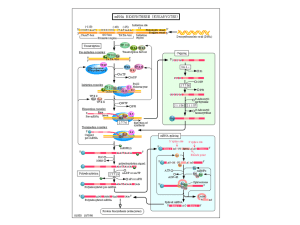
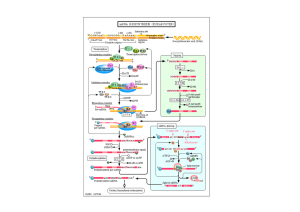
rDNA
I PRECURSORI DEGLI rRNA SONO PIU' LUNGHI
DELLA SOMMA DEI DUE rRNA MATURI
Fig. 1. Relationships between the nucleolus, secondary constriction, NOR and ribosomal RNA genes. Typically, only a subset of the rRNA genes within a nucleolus organizer
region (NOR) is active; subsequent transcription by RNA polymerase I and pre-rRNA processing causes the nucleolus to form. At metaphase, the rRNA genes that had been
active during the preceding interphase give rise to a secondary constriction due to the persistent binding of transcription factors. The secondary constriction is
decondensed to the point that it is generally not visible using standard light microscopy techniques. Within an NOR the tandemly arrayed rRNA genes are separated from
one another by an intergenic spacer that typically contains repeated elements as well as the rRNA gene promoter, at which the transcription start site is denoted as + 1.
The transcribed portion of the gene includes an external transcribed sequence (ETS) and two internal transcribed sequences, ITS1 and ITS2 that separate the 18S, 5.8S and
25S structural rRNA sequences. Multiple RNA processing events are needed to generate the structural RNAs from the primary transcript.
Maturazione dell’RNA ribosomale negli eucarioti
Tutti i genomi contengono copie multiple
dei geni per l’rRNA e per i tRNA
Specie
18S/28S rRNA
genes
E.coli
S.cerevisiae
D.discoideum
D.melanogaster
X
Y
X.laevis
H.sapiens
7
140
180
250
150
450
280
5S RNA
genes
tRNA
genes
7
140
180
165
60
250
?
850
24,000
2,000
1,150
1,300
tRNA
miRNA
GENI SOVRAPPOSTI (es. MHC)
GENI SOVRAPPOSTI
CLUSTER GENICI
PSEUDOGENI PROCESSATI
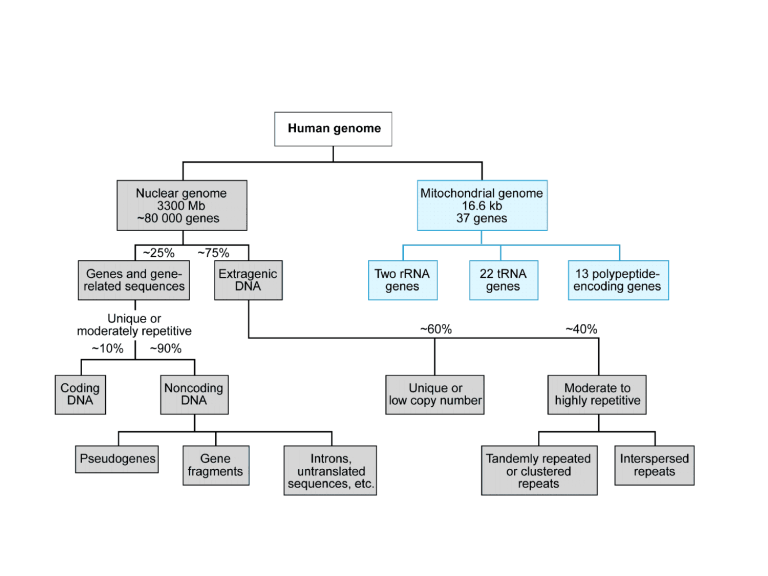
ARCHITETTURA DEL GENOMA
Protein coding: ca. 21,000 di cui >90% con splicing alter. De novo
origin = very rare
Conserved elements: Transcription?? Conserved, Highly C., Ultra
C. Enriched in regulatory elements
3.6 million constrained elements can be detected
encompassing 4.2% of the genome, including a
substantial fraction of newly detected bases (blue)
Lindblad-Toh K, et al. (2011) Nature 478:476-482
Small non-coding RNA: 100 families of miRNA ca. 200 target
mRNAs per ogni miRNA
Large non-coding RNA: 10% del genoma si trova sotto forma di
trascritti maturi non derivati da esoni. Molti con bassi livelli di
espressione.
lincRNA coinvolti in cell-cycle regulation, immune response, brain
processes, gametogenesis.
Mappe epigenomiche: mappatura delle porzioni funzionalmente
attive.
Promotori attivi: ipersensitivita’ alla DNAsi, acetilazione degli
istoni, istone 3 lys-4 trimetilata.
Mappe epigenomiche: mappatura delle porzioni funzionalmente
attive.
Promotori attivi: ipersensitivita’ alla DNAsi, acetilazione degli
istoni, istone 3 lys-4 trimetilata.
Large non-coding RNA: 10% del genoma si trova sotto forma di
trascritti maturi non derivati da esoni. Molti con bassi livelli di
espressione.
lincRNA coinvolti in cell-cycle regulation, immune response, brain
processes, gametogenesis.