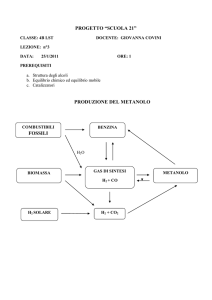
Metanogenesi
E’ la reazione di formazione del metano ad opera di microorganismi
(phylum Euryarchaeota – per lo più mesofili).
E’ una forma di respirazione anaerobica in cui accettore di
elettroni è il carbonio di composti a basso peso molecolare, quali CO2,
HCOOH, CH3OH. Quasi esclusivamente C1 o cmq acidi carbossilici con
gruppo metile (CH3COOH, CH3COCOOH)
La reazione avviene solo in assenza di O2 (è tossico per i
metanogeni), in ambienti dove si ha decadimento della materia
organica:
• sedimenti anossici: paludi, acquitrini, sedimenti lacustri, risaie, …
• zone umide di discarica
• tratto digestivo di animali: rumine, intestino cieco (cavalli, conigli, …) o
crasso (uomini, cani, …), intestino superiore di insetti cellulolitici (p.es.
termiti)
• sorgenti geotermali di H2 e CO2.
• impianti di biodegradazione: digestori a fanghi attivi
Produzione di metano nel rumine
Gli Archaea e i metanogeni
Ci sono 3 tipi di Archaea
Crenarchaeota (Estremofili (termofili e psicrofili), acidofili)
Euryarchaeota (metanogeni e alofili)
Korarchaeota (nessun organismo isolato)
Metanogeni: sono anaerobiotici obbligati
Methanococcus jannischii was originally isolated from a sample taken from an oceanic depth of 2,600
meters on the East Pacific Rise. It can be grown in a mineral medium containing only H2 and CO2 as
sources of energy and carbon for growth within a temperature range of 50 to 86 degrees.
Cells are irregular cocci that are motile due to two bundles of polar flagella inserted near the same
cellular pole. Its genome has been sequenced completely.
GLI ARCHAEA METANOGENI
ORDINE: METHANOBACTERIALES
Famiglia: Methanobacteriaceae
Genere:Methanobacterium
Genere: Methanobrevibacter
H2/CO2
Genere: Methanosphaera
formiato
Famiglia: Methanothermaceae
Genere: Methanothermus
ORDINE: METHANOCOCCALES
Famiglia: Methanococcaceae
Genere: Methanococcus
LE REAZIONI
4H2 + HCO3- + H+
CH4 + 3H2O
CH3OH + H2
CH4 + H2O
CH3COO- + H2O
CH4 + HCO3-
H2/CO2
ORDINE: METHANOMICROBIALES
Famiglia: Methanomicrobiaceae
Genere: Methanomicrobium
Genere: Methanogenium
H2/CO2
Genere: Methanoculleus
Genere: Methanospirillum
Famiglia: Methanocorpusculaceae
Genere: Methanocorpusculum H2/CO2
Famiglia: Methanoplanaceae
Genere: Methanoplanus
H2/CO2
Famiglia: Methanosarcinaceae
Genere: Methanosarcina
acetato H2/CO2
Genere: Methanococcoides
Genere: Methanolobus
methanolo
Genere: Methanohalophilus
Genere: Methanosaeta/Methanotrix acetato
Acetotrofia
CO2 reduction
Acetate fermentation
Metanogenesi
Rappresenta il passaggio finale della decomposizione della
materia organica.
Durante tale decadimento accettori di elettroni come
come O2, SO4 2-, NO3-, Mn(VI), Fe(III) vengono eliminati mentre si
accumulano H2 e CO2.
In queste condizioni avvengono
Fermentazione
Metanogenesi
Fermentazione = è la degradazione anaerobica di
composti organici come nutrienti (es glucosio) per ottenere
composti organici più piccoli (es. lattato, acido acetico, etanolo)
senza ossidazione.
Metanogenesi = rimuove i prodotti finali di questo
decadimento e permette di convertire rifiuti organici in “biogas”
Acetate fermentation
CO2 reduction
CH4 prodotto/anno da batteri metanogenici = 109 tonnellate.
Metà è utilizzato dai batteri metanotrofici, metà va nell’atmosfera (effetto serra).
La biotrasformazione impiega cofattori che trasportano un atomo di C
Metanofurano
Tetraidro-metanopterina
Coenzima M
CoM-SH è unico per i metanogeni, gli altri sono presenti anche in altri Archaea
formil-MFR
formil-H4MPT
metenil-H4MPT
metilene-H4MPT
metil-H4MPT
metil-S-CoM
Ruolo fisiologico delle idrogenasi
L’attività dei batteri metanogenici è collegata al metabolismo di H2.
La reazione di ossidazione di H2 produce e- e H+ sul lato esterno delle membrane.
Gli elettroni attraversano la membrana e vengono utilizzati per la riduzione di HCO3a CH4.
A questa reazione è associata la sintesi di ATP.
E’ presente una ADP/ATP translocasi che trasferisce ADP all’interno della membrana
esterno
membrana
interno
HCO3- + 9 H+
4 H2
8 e8 H+
CH4 + 3 H2O
ATPasi
ADP + Pi
2
H+
ATP
Formil-metanofurano deidrogenasi
L’enzima (FDH) catalizza la riduzione di CO2 +
MFR a N-formilmetano furano.
Contiene Mo, un molibdopterina dinucleotide e
cluster Fe-S
Il coinvolgimento nella reazione del MFR rende lo step unico, tuttavia il meccanismo
non lo è: sia Mo che Mo-pterina sono presenti anche in altri enzimi simili
(xantina ossidasi che catalizza ox. di xantina ad acido urico).
Methanobacterium wolfei contiene 2 FDH (I e II). FDHI predomina in cellule
che crescono in presenza di Mo, FDH II in cellule che crescono in presenza
di W. W-enzimi sono presenti anche in altri batteri anaerobici.
Donatori di elettroni nella metanogenesi
F420H2 assomiglia a FMN e a FAD.
Trasferisce 2 e- come H- (come NADH)
Metil-H4MPT:CoM metiltransferasi
E’ un’enzima di membrana (Mtr) costituito
da 8 subunità. Ognuna porta come gruppo
prostetico la 5’-idrossibenzimidazolil-cobamide
Quando il substrato e’
metanolo/metilammina
/metilsolfuro si parte
direttamente da qui,
ossia dalla formazione
di metil-CoM
Metil-H4MPT:CoM metiltransferasi
Nella forma super-ridotta come cob(I)amide il cofattore accetta il gruppo metilico
dal CH3-H4MPT producendo metil-cob(III)amide.
CH3-H4MPT + E:CoB(I)
H4MPT + E:CH3-CoB(III)
In una seconda reazione il gruppo metilico viene trasferito a HS-CoM rigenerando
la forma ridotta (Co(I)) dell’enzima e metil-CoM
E:CH3-CoB(III) + HS-CoM
CH3-S-CoM + E:CoB(I)
Simili traferimenti di metili (ad es. formazione di metionina ad opera di
metionina sintasi) avvengono in batteri ed eucarioti, ma utilizzano proteine solubili,
con un sola subunità.
Zn(II)
H
Zn(II)
H
O
CH3
Zn(II)
O
CH3
+HS-CoM
-CH3S-CoM -H2O
Co(I)
Co(III)
Co(I)
His136
His136
His136
Metil-CoM riduttasi
Catalizza la formazione di CH4 da metil-CoM.
Contiene come gruppo prostetico F430, un
nichel porfinoide con i 4 N pirrolici che
coordinano il Ni.
La riduzione dell’etero
disolfuro e’ esoenergetica e
associata alla sintesi di ATP
Il Nickel (I) lega il CH3, che viene
ridotto e rilasciato come CH4. A
differenza di prima, quando col Co si
aveva trasferimento e non riduzione.
CoB-SH, metil –S-CoM e F430 sono tipici di
archaeobatteri metanogenici.
Quando non si parte da CO2 …
METANOGENESI: RESE ENERGETICHE E SINTROFIA
E’ un processo metabolico molto meno esoergonico della respirazione
aerobica o delle respirazioni anaerobiche
La conversione di un esoso a CH4 rilascia solo il 15% dell’energia ottenibile
dalla degradazione aerobica
C6H12O6 + 6O2
C6H12O6
6CO2 + 6H2O (DG°’ = -2870 kJ/mole)
3CO2 + 3CH4 (DG°’ = -390 kJ/mole)
La bassa resa energetica della metanogenesi costringe i microrganismi
coinvolti a cooperare molto efficientemente ed a stabilire relazione di
“SINTROFIA”
la cooperazione tra 2 organismi che dipendono l’uno dall’altro. La mutua
dipendenza non può essere sostituita dall’aggiunta di nutrienti. Ad es. la cocoltura di Methanobacillus omelianskii degrada l’etanolo a:
Strain S
2CH3CH2OH + 2H2O
2CH3COO- + 2H+ + 4H2 (DG°’ = +19 kJ per 2 mol of ethanol)
Strain M.o.H.
4H2 + CO2
CH4 + 2H2O (DG°’ = -131 kJ per mol of methane)
Co-Culture
2CH3CH2OH + CO2
2CH3COO- + 2H+ + CH4 (DG°’ = -112 kJ per mol of methane)
Obligate syntrophy between an acetogen and a methanogen is common
Each species (e.g., a methanogen and an acetogen) requires the other:
the acetogen provides the hydrogen; the methanogen prevents a buildup of hydrogen (which inhibits the acetogens)
METANOGENESI: RESE ENERGETICHE E SINTROFIA
Un processo che favorisce la sintrofia è il
TRASFERIMENTO INTERSPECIE DI H2, HCOO- E CH3COOin cui un efficiente “scavenger” per questi substrati (es. metanogeni o
solfatoriduttori) ne mantiene una bassa concentrazione, favorendone la
produzione da parte degli acetogeni sintrofici
Fermentazione
Glucosio + 4 H2O -> 2 acetato + 2 HCO3- + 4 H+ + 4 H2
DG=-207 kJ mol-1 // -319 kJ mol-1
2 Etanolo + 2 H2O -> 2 acetato + 2 HCO3- + 2 H+ + 4 H2
DG=+19,4 kJ mol-1 // -37 kJ mol-1
Metanogenesi
4 H2 + HCO3- + H+ -> CH4 + 3 H2O
DG=-136 kJ mol-1 // -3,2 kJ mol-1
Acetogenesi
4 H2 + 2 HCO3- + H+ -> acetato + 4 H2O
DG=-105 kJ mol-1 // -7,1 kJ mol-1
Sintrofia
Condizioni standard // In acqua dolce
CH4, 0.6 atm; H2, 10-4 atm
What happens to all this methane?
Diffusive transport up into oxic zone – aerobic methane
oxidation
Bubble ebullition (in shallow seds, with strong temperature
or pressure cycles) followed by oxidation in atmosphere
Anaerobic methane oxidation coupled to sulfate reduction
Gas hydrate formation
Degradazione anaerobica del metano in sedimenti marini
SO42- + 4 H2 + H+ -> HS- + 4 H2O DG=-156 kJ mol-1
Inverso della
metanogenesi
CH4 + 2 H2O -> CO2 + 4 H2
DG=+131 kJ mol-1
Totale: SO42- + CH4 -> HCO3- +HS- + H2O
Used fluorescent
probes to label, image
aggregates of archaea
(methanogens, red) and
sulfate reducers (green)
in sediments from
Hydrate Ridge (OR) –
observed very tight
spatial coupling.
Boetius et al., 2000
(CH2 O)n
Fotosintesi
Batteri
chemolitotrofi
Respirazione
Metanotrofi
+O2
CH4
CO2
-O2
Metanogeni
Batteri
fototrofi
Respirazione
anaerobica
Fermentazione
(CH2 O)n
Low T + high P + adequate gas (methane, trace other HC,
CO2) => gas hydrate
Why do we care about methane hydrates?
Resource potential
Fluid flow on margins
Slope destabilization / slope failure
Chemosynthetic biological communities
Climate impact potential
1 m3 hydrate => 184 m3 gas + 0.8 m3 water
DIC = 980
Terr bio = 830
Peat = 500
Atm = 3.5
Mar bio = 3
Total fossil fuel =
5000 x 1015 gC
total hydrate =
10,000 x 1015
gC (a guess!)
METANOGENESI: I REATTORI PER REFLUI
•Reattori a biomassa libera
•Reattori a biomassa adesa
Migliore contatto tra le cellule e migliore sintrofia
(es. la rimozione dell’H2 prodotto dagli acetogeni sintrofici, Sintrophomonas e Sintrophobacter,
da parte dei metanogeni idrogenotrofi che agiscono da “scavenger” dell’H 2 tossico per gli
acetogeni)
Stabilità della biomassa nel reattore
(basso “washout” cellulare)
Reattori UASB
(upflow anaerobic sludge bed)
Reattori a letto fisso
espanso fluidizzato
Strati di microrganismi
La biomassa è organizzata in
biofilm autoimmobilizzato in
granuli compatti (diam. 0.1-1
cm) ad alta sedimentabilità
Materiale di supporto
Le cellule sono
immobilizzate in un biofilm
adeso ad un supporto inerte
(plastica, legno,ecc.)
METANOGENESI: I REATTORI UASB
Biogas
Ricircolo
Effluente
Influente
MESOFILI
TERMOFILI
The ribosome is the site of
translation of messenger RNA
into protein. It is composed of
two subunits. In prokaryotes, the
large subunit is called 50S and
the small subunit is called 30S.
The 30S subunit consists of a
single strand of RNA (the 16S
rRNA, 1542 bases), and 21
proteins ranging in molecular
weight from 9 kD to 61 kD. The
30S subunit can exist
independently from the 50S
subunit and be reconstituted
from its components in vitro.
The 30S subunit is the site of
translation initiation. An
understanding of its structure is
useful for understanding the
mechanisms of protein
translation, and perhaps for
designing drugs to modify these
mechanisms.
DGGE:
elettoforesi su gel
in gradiente
denaturante
rid.
idrogenasi
2 eformil-MFR
formil-MFR deidrogenasi
Fe-S, MPTdinucleotide, Mo
enzima di membrana – diverse subunità
formil-H4MPT
metenil-H4MPT
idrogenasi
rid.
(b-e) enzimi solubili,
1 subunità, senza gruppi
prostetici
2 emetilene-H4MPT
rid.
idrogenasi
2 emetil-H4MPT
Metil-H4MPT:CoMtransferasi
Fe-S, corrinoide, di membrana
metil-S-CoM
Eterodisolfuro rid.
riduttasi2 e-
metil-S-CoM reduttasi
3 subunità, F430=nichel tetrapirrolo